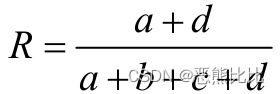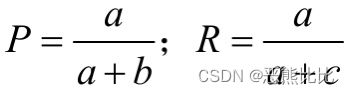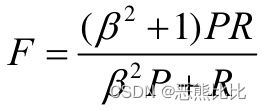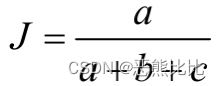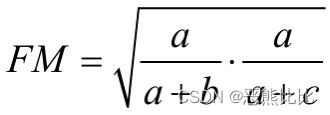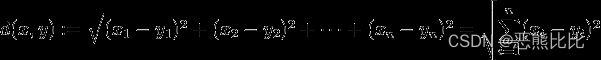K-means聚类算法原理及实现
文章目录
-
- 1.聚类分析
-
- 1.1概念
- 1.2 K 均值和层次聚类
- 2. 聚类分析的度量
-
-
- 2.1 外部指标
- 2.2 内部度量
-
- 3.K-means概念
- 4.K-means算法步骤
- 5.K-means案例1(python代码)
1.聚类分析
1.1概念
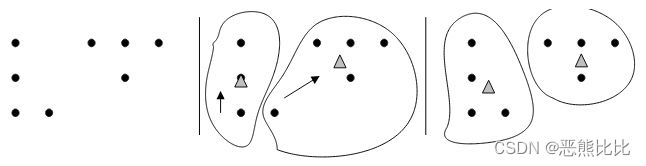
聚类分析,也称为分割分析或分类分析,可将样本数据分成一个个组(即簇)。同一簇中的对象是相似的,不同簇中的对象则明显不同。
Statistics and Machine Learning Toolbox™ 提供了几种聚类方法和相似性度量(也称为距离度量)来创建簇。此外,簇计算可以按照不同的计算标准确定数据的最佳簇数。
1.2 K 均值和层次聚类
K 均值聚类是一种分区方法,它将数据中的观测值视为具有位置和相互间距离的对象。它将对象划分为 K 个互斥簇,使每个簇中的对象尽可能彼此靠近,并尽可能远离其他簇中的对象。每个簇的特性由其质心或中心点决定。当然,聚类中使用的距离通常不代表空间距离。
层次聚类是通过创建聚类树,同时在多个距离尺度内调查数据分组的一种方法。与 K-均值法不同,树并不是一组簇的简单组合,而是一个多级层次结构,较低级别的簇在相邻的更高级别合并成新的簇。使用这种方法,可以选择最适合您的应用场景的聚类尺度或级别。
Fisher 鸢尾花数据的聚类分析:
https://ww2.mathworks.cn/help/stats/cluster-analysis-example.html
2. 聚类分析的度量
度量指标对聚类结果进行评判;
- 外部指标:以事先指定的聚类模型为参考;
- 内部指标:参与聚类的样本;
2.1 外部指标
对含有n个样本的数据集S,其中两个样本点x1,x2,假定通过聚类给出的簇划分结果为C,外部参考模型给出的结果为P;对于x1,x2存在四种关系:
- SS:x1、x2在C和P中属于相同的簇;
- SD:在C中相同,P中不同;
- DS:C中不同,P中相同;
- DD:x1、x2在C和P中属于不同的簇;
a,b,c,d分别表示上述关系对应的关系数目,x1和x2存在且唯一存在上四种关系之一;
a+b+c+d = n(n-1)/2;
外部度量指标:
- Rand统计量
- 以上四个度量结果越大,表明聚类结果和参考模型的划分结果越吻合;
2.2 内部度量
2.2.1 样本点和聚类中心的距离度量
计算欧氏空间两个点之间的距离
也称城市街区距离度量,相当于沿着两个街道行驶的距离(实际行驶距离)
- 切比雪夫距离
向量空间距离的度量,相当于象棋盘移动格子之间的距离
- 可明夫斯基距离
欧氏和曼哈顿的推广
聚类性能度量
- 紧密性
指每个样本到聚类中心的平均距离
- 分隔度
是个簇的簇心之间的平均距离。分割度值越大说明簇间间隔越远,分类效果越好,即簇间相似度越低。
- 戴维森保丁指数
衡量任意两个簇的簇内距离之后与簇间距离之比。该指标越小表示簇内距离越小,簇内相似度越高,簇间距离越大,簇间相似度低。
- 邓恩指数
任意两个簇的样本点的最短距离与任意簇中样本点的最大距离之商。该值越大,聚类效果越好。
- 轮廓系数
对于一个样本集合,它的轮廓系数是所有样本轮廓系数的平均值。轮廓系数的取值范围是[-1,1],同类别样本距离越相近不同类别样本距离越远,分数越高。
3.K-means概念
k-means算法 :又名k均值算法,是基于划分的聚类,K-means算法中的k表示的是聚类为k个簇,means代表取每一个聚类中数据值的均值作为该簇的中心,或者称为质心,即用每一个的类的质心对该簇进行描述。
算法思想:先从样本集中随机选取 k个样本作为簇中心,并计算所有样本与这 k个“簇中心”的距离,对于每一个样本,将其划分到与其距离最近的“簇中心”所在的簇中,对于新的簇计算各个簇的新的“簇中心”。
- 四个要点:
(1)簇个数 k 的选择
(2)各个样本点到“簇中心”的距离
(3)根据新划分的簇,更新“簇中心”
(4)重复上述2、3过程,直至"簇中心"没有移动
优点:
计算速度快、易于理解;
4.K-means算法步骤
- K值的选择: 选取K个簇类的质心(通常为随机);
- 距离度量: 计算剩余样本到各质心的距离(一般为欧氏距离),归类到相互距离最小的质心所在的簇;
- 新质心计算: 剩余点归类完毕后计算新质心,然后再重新计算各样本到质心的距离;
- 停止条件: 迭代计算完毕所有的样本点的距离,当样本的距离划分情况基本不变时,说明已经到最优解,返回结果;
5.K-means案例1(python代码)
参考链接: https://blog.csdn.net/qq_43741312/article/details/97128745
代码:
import random
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
# 计算欧拉距离
def calcDis(dataSet, centroids, k):
clalist = []
for data in dataSet:
diff = np.tile(data, (k,
1)) - centroids # 相减 (np.tile(a,(2,1))就是把a先沿x轴复制1倍,即没有复制,仍然是 [0,1,2]。 再把结果沿y方向复制2倍得到array([[0,1,2],[0,1,2]]))
squaredDiff = diff ** 2 # 平方
squaredDist = np.sum(squaredDiff, axis=1) # 和 (axis=1表示行)
distance = squaredDist ** 0.5 # 开根号
clalist.append(distance)
clalist = np.array(clalist) # 返回一个每个点到质点的距离len(dateSet)*k的数组
return clalist
# 计算质心
def classify(dataSet, centroids, k):
# 计算样本到质心的距离
clalist = calcDis(dataSet, centroids, k)
# 分组并计算新的质心
minDistIndices = np.argmin(clalist, axis=1) # axis=1 表示求出每行的最小值的下标
newCentroids = pd.DataFrame(dataSet).groupby(
minDistIndices).mean() # DataFramte(dataSet)对DataSet分组,groupby(min)按照min进行统计分类,mean()对分类结果求均值
newCentroids = newCentroids.values
# 计算变化量
changed = newCentroids - centroids
return changed, newCentroids
# 使用k-means分类
def kmeans(dataSet, k):
# 随机取质心
centroids = random.sample(dataSet, k)
# 更新质心 直到变化量全为0
changed, newCentroids = classify(dataSet, centroids, k)
while np.any(changed != 0):
changed, newCentroids = classify(dataSet, newCentroids, k)
centroids = sorted(newCentroids.tolist()) # tolist()将矩阵转换成列表 sorted()排序
# 根据质心计算每个集群
cluster = []
clalist = calcDis(dataSet, centroids, k) # 调用欧拉距离
minDistIndices = np.argmin(clalist, axis=1)
for i in range(k):
cluster.append([])
for i, j in enumerate(minDistIndices): # enymerate()可同时遍历索引和遍历元素
cluster[j].append(dataSet[i])
return centroids, cluster
# 创建数据集
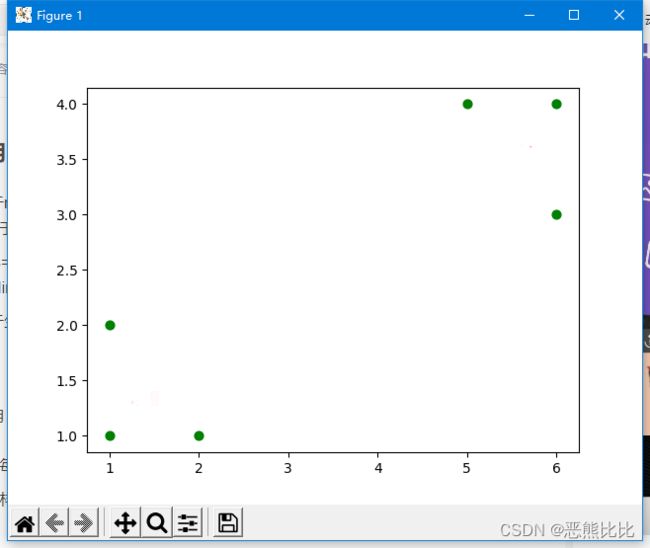
def createDataSet():
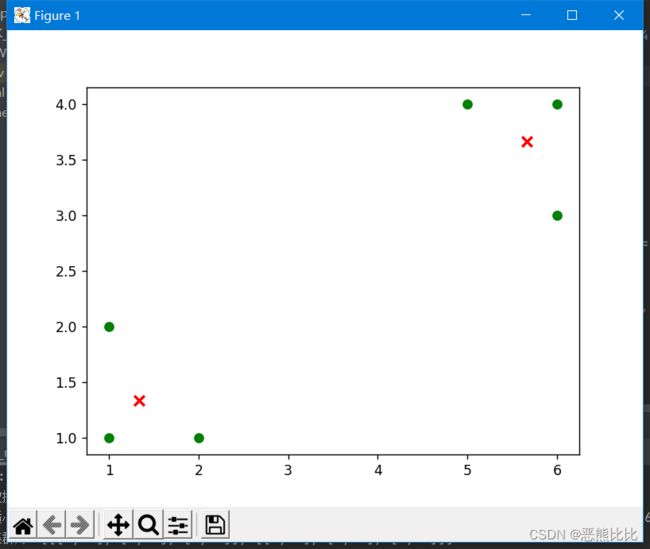
return [[1, 1], [1, 2], [2, 1], [6, 4], [6, 3], [5, 4]]
if __name__ == '__main__':
dataset = createDataSet()
centroids, cluster = kmeans(dataset, 2)
#print("数据集为:%s"%dataset)
print('质心为:%s' % centroids)
print('集群为:%s' % cluster)
for i in range(len(dataset)):
plt.scatter(dataset[i][0], dataset[i][1], marker='o', color='green', s=40, label='原始点')
# 记号形状 颜色 点的大小 设置标签
for j in range(len(centroids)):
plt.scatter(centroids[j][0], centroids[j][1], marker='x', color='red', s=50, label='质心')
#plt.show()
plt.show()
E:\PythonStudy\venv\Scripts\python.exe E:/PythonStudy/DeepLeraring/K_means_demo.py
质心为:[[1.3333333333333333, 1.3333333333333333], [5.666666666666667, 3.6666666666666665]]
集群为:[[[1, 1], [1, 2], [2, 1]], [[6, 4], [6, 3], [5, 4]]]
Process finished with exit code 0