R实战 | 置换多元方差分析(以PCoA的PERMANOVA分析为例)
置换多元方差分析(Permutational multivariate analysis of variance,PERMANOVA),又称非参数多因素方差分析(nonparametric multivariate analysis of variance)、或者ADONIS分析。它利用距离矩阵(如欧式距离、Bray-Curtis距离)对总方差进行分解,分析不同分组因素或不同环境因子对样品差异的解释度,并使用「置换检验」对各个变量解释的统计学意义进行显著性分析。
一个例子
比如,对宏基因组检测的物种丰度数据进行PCA/NMDS/PCoA降维可视化后,不同组的样品之间存在一些重叠,那怎么判断这些组之间的样品构成是否存在显著差别呢?这就需要用到PERMANOVA检验了,检验不同组的样品中心点是否重叠。
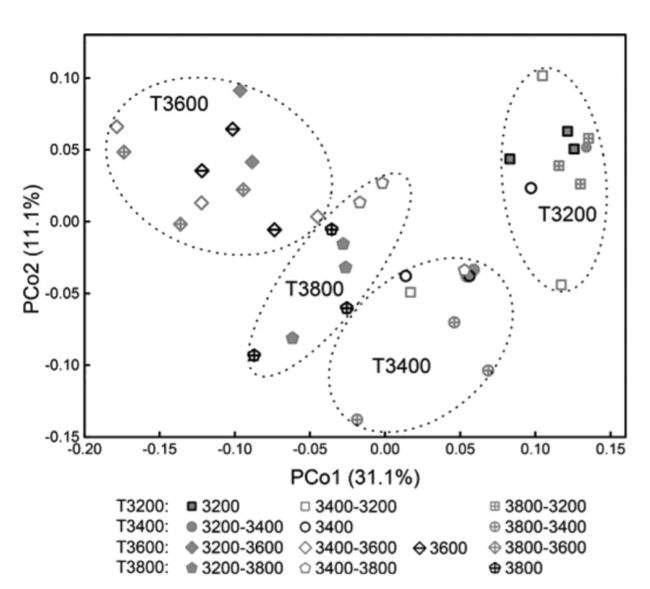 example
example 

以上面的PCoA图为例,椭圆圈出的四组样品点正好对应四个海拔分组,这四组样品之间的群落差异是否显著呢?检验组间群落差异本质上是检验距离矩阵之间的差异,普通的ANOVA分析无能为力。而基于距离矩阵的PerMANOVA分析则表明,这四个分组两两之间差异是显著的(p<0.05)。
❝Rui J, Li J, Wang S, et al. Responses of Bacterial Communities to Simulated Climate Changes in Alpine Meadow Soil of the Qinghai-Tibet Plateau. Appl Environ Microbiol. 2015;81(17):6070-6077. doi:10.1128/AEM.00557-15
❞
示例数据和代码领取
点赞、在看 本文,分享至朋友圈集赞20个并保留30分钟,截图发至微信mzbj0002领取。
「木舟笔记2022年度VIP可免费领取」。
❝「注:」2022马上过去了,为了方便各位读者朋友,现推出
❞木舟笔记永久VIP,售价169¥。2022VIP仅需支付差价进行升级。木舟笔记永久VIP享本号所有资源(限定课程除外),后续不再推出VIP企划。
木舟笔记2022年度VIP企划
「权益:」
「2022」年度木舟笔记所有推文示例数据及代码(「在VIP群里实时更新」)。
 data+code
data+code 木舟笔记「科研交流群」。
「半价」购买
跟着Cell学作图系列合集(免费教程+代码领取)|跟着Cell学作图系列合集。
「收费:」
「99¥/人」。可添加微信:mzbj0002 转账,或直接在文末打赏。

实战
PCoA
# Load package
library(vegan)
library(ggplot2)
library(ggthemes)
# Load data
otu <- read.table('otu.txt',row.names = 1,header = T)
group <- read.table('group.txt',header = T)
#pcoa
# vegdist函数,计算距离;method参数,选择距离类型
distance <- vegdist(otu, method = 'bray')
# 对加权距离进行PCoA分析
pcoa <- cmdscale(distance, k = (nrow(otu) - 1), eig = TRUE)
## plot data
# 提取样本点坐标
plot_data <- data.frame({pcoa$point})[1:2]
# 提取列名,便于后面操作。
plot_data$ID <- rownames(plot_data)
names(plot_data)[1:2] <- c('PCoA1', 'PCoA2')
# eig记录了PCoA排序结果中,主要排序轴的特征值(再除以特征值总和就是各轴的解释量)
eig = pcoa$eig
#为样本点坐标添加分组信息
plot_data <- merge(plot_data, group, by = 'ID', all.x = TRUE)
head(plot_data)
# 计算加权bray-curtis距离
dune_dist <- vegdist(otu, method="bray", binary=F)
dune_pcoa <- cmdscale(dune_dist, k=(nrow(otu) - 1), eig=T)
dune_pcoa_points <- as.data.frame(dune_pcoa$points)
sum_eig <- sum(dune_pcoa$eig)
eig_percent <- round(dune_pcoa$eig/sum_eig*100,1)
colnames(dune_pcoa_points) <- paste0("PCoA", 1:3)
dune_pcoa_result <- cbind(dune_pcoa_points, group)
head(dune_pcoa_result)
library(ggplot2)
ggplot(dune_pcoa_result, aes(x=PCoA1, y=PCoA2, fill=group)) +
geom_point(shape = 21,color = 'black',size=4) +
stat_ellipse(level=0.95)+
scale_fill_manual(values = c('#73bbaf','#d15b64','#592c93'))+
labs(x=paste("PCoA 1 (", eig_percent[1], "%)", sep=""),
y=paste("PCoA 2 (", eig_percent[2], "%)", sep="")) +
theme_classic()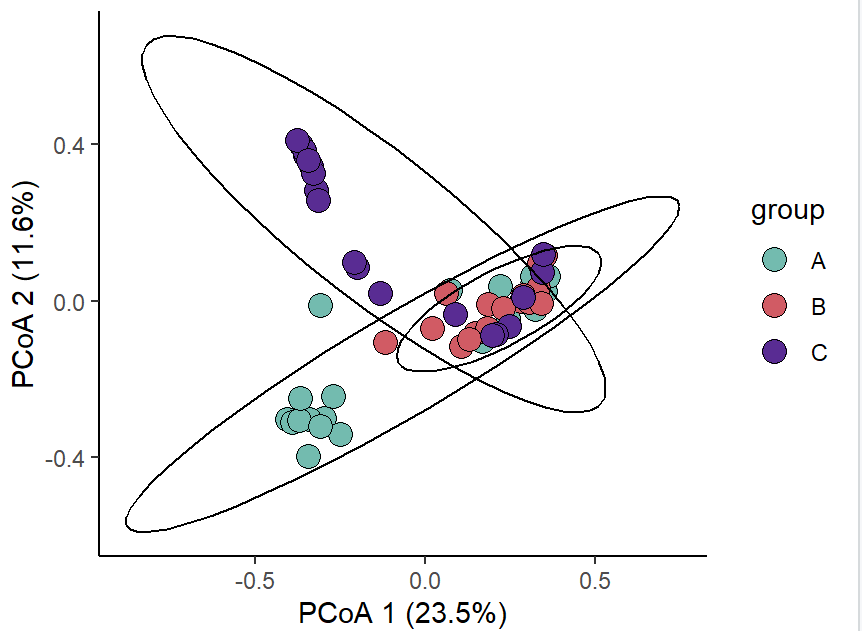
PERMANOVA
# 基于bray-curtis距离进行计算
dune.div <- adonis2(otu ~ group, data = group, permutations = 999, method="bray")
dune.div
library(ggalt)
dune_adonis <- paste0("adonis R2: ",round(dune.div$R2,2), "; P-value: ", dune.div$`Pr(>F)`)
p <- ggplot(dune_pcoa_result, aes(x=PCoA1, y=PCoA2, fill=group)) +
geom_point(shape = 21,color = 'black',size=4) +
stat_ellipse(level=0.95)+
scale_fill_manual(values = c('#73bbaf','#d15b64','#592c93'))+
labs(x=paste("PCoA 1 (", eig_percent[1], "%)", sep=""),
y=paste("PCoA 2 (", eig_percent[2], "%)", sep=""),
title=dune_adonis) +
theme_classic()
p image-20221228004115608
image-20221228004115608
配对Adonis
# 配对Adonis确定两两分组之间对物种组成差异的影响
#devtools::install_github("pmartinezarbizu/pairwiseAdonis/pairwiseAdonis")
library(pairwiseAdonis)
dune.pairwise.adonis <- pairwise.adonis(x=otu, factors=group$group,
sim.function = "vegdist",
sim.method = "bray",
p.adjust.m = "BH",
reduce = NULL,
perm = 999)
library(ggpubr)
library(patchwork)
tab2 <- ggtexttable(dune.pairwise.adonis[,c("pairs","R2","p.value","p.adjusted")], rows = NULL,
theme = ttheme("blank")) %>%
tab_add_hline(at.row = 1:2, row.side = "top", linewidth = 1) %>%
tab_add_hline(at.row = nrow(dune.pairwise.adonis)+1, row.side = "bottom", linewidth = 1)
p + tab2 + plot_layout(design=c(area(1,1), area(2,1)))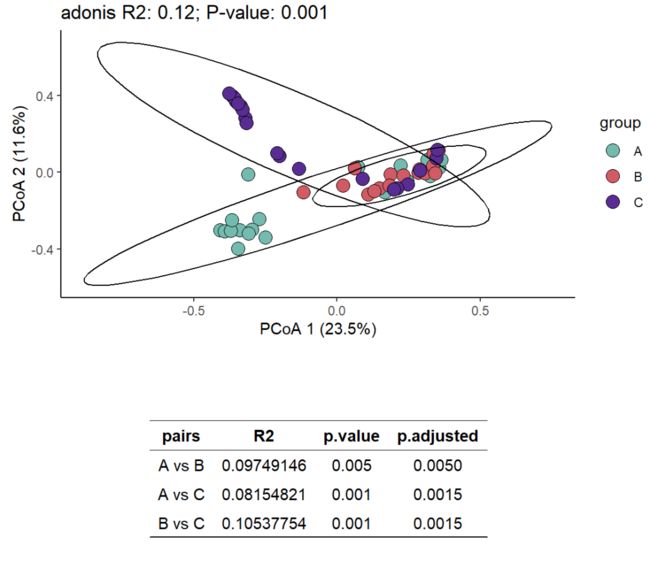
往期内容
CNS图表复现|生信分析|R绘图 资源分享&讨论群!
这图怎么画| 有点复杂的散点图
这图怎么画 | 相关分析棒棒糖图
组学生信| Front Immunol |基于血清蛋白质组早期诊断标志筛选的简单套路
(免费教程+代码领取)|跟着Cell学作图系列合集
Q&A | 如何在论文中画出漂亮的插图?
跟着 Cell 学作图 | 桑葚图(ggalluvial)
R实战 | Lasso回归模型建立及变量筛选
跟着 NC 学作图 | 互作网络图进阶(蛋白+富集通路)(Cytoscape)
R实战 | 给聚类加个圈圈(ggunchull)
R实战 | NGS数据时间序列分析(maSigPro)
跟着 Cell 学作图 | 韦恩图(ggVennDiagram)
 木舟笔记矩阵
木舟笔记矩阵
