【PaperReading】Linked open data-based framework for automatic biomedical ontology generation
《Linked open data-based framework for automatic biomedical ontology generation》
《链接开放的基于数据的框架,用于自动生成生物医学本体》
摘要
背景: 实现语义Web的愿景需要一个准确的数据模型来组织知识和分享对专业领域的通用理解。据此,本体是语义Web的基石,可以用于解决临床信息和生物医学工程的许多问题,例如词义消歧,,语义相似性,问答及本体对齐等等。本体的手工构建是劳动密集型的,并且需要领域专家以及本体工程师。为了缩小本体的劳动密集型性质,并且最大限度的减少对领域专家的需求,我们提出了一种新的自动本体生成框架,用于生物医学本体生成的链接开放数据方法(LOD-ABOG),其中由开放链接数据授权(LOD)。LOD-ABOG使用知识库(主要是UMLS和LOD)以及自然语言处理(NLP)操作来执行概念提取,并且使用LOD,广度优先搜索(BSF)图方法,和Freepal存储库模式应用于关系的提取。
结果: 我们的评估显示,与现有的框架相比,本体生成的大部分任务都有了改进的结果。我们使用CDR和SemMedDB数据集评估了所提出框架的各个任务(模块)的性能。对于概念提取,评估显示CDR语料库的平均F值为58.12%,SemMedDB的平均F值为81.68%;使用CDR和SemMed数据集分别额提取生物医学分类关系的F值为65.26%和77.44%;分别使用CDR语料库和SemMedDB进行生物医学非分类关系提取的F值为52.78%和58.12%。此外,与手动构建的极限阿尔兹海默症本体比较表明,在概念检测方面,F值为72.48%,关系提取方面,F值为83.28%。此外我们将我们提出的框架与成为“OntoGain”的本体学习框架进行了比较,该框架表明LOD-ABOG在关系提取方面的表现要好14.76%。
结论: 本文提出了LOD-ABOG框架,该框架表明当前的LOD资源和技术是一种很有前景的解决方案,可以自动生成生物医学本体,并且在更大程度上提取关系。此外,与在本体开发过程中需要领域专家的现有框架不同,我们提出的方法仅仅在本体生命周期的最后,即改进时需要领域专家的参与。
关键词: 语义网,本体生成,开放链接数据,语义丰度
背景
在大数据时代,以及当今网络充斥大量的信息和数据的时代,迫切需要我们建模、组织和改进数据的方式。对数据建模的一种方法是设计本体,并使用他们来最大化从结构化和非结构化数据访问,以及提取有价值的隐式和显式知识的益处。本体论是将文档Web转化为数据Web的重要部分。本体的基本原理是使用一种主要本体语言,以标准的格式来表示数据或者事实,即资源描述框架(RDF),资源描述框架模式(RDFs),Web本体语言(OWL),或简单的知识组织系统(SKOS)。
在过去的十年中,本体生成已经成为许多领域和生物信息学领域最具革命性的发展之一。有各种方法创建本体,这些方法包括:
基于规则的语法分析,基于句法模式的,基于字典的机器学习,以及基于知识的。 基于规则的方法涉及手动制定的一组规则,这些规则被形成以表示决定在各种场景中做什么或得出的结论知识。通常,它实现了非常高的精度,但召回率非常低,这种方法是劳动密集型的,适用于一个特定的领域,并且可扩展性非常低。另一方面,基于句法模式的方法在本体工程中得到了很好的研究,并且已经被证明在非结构化文本的本体生成中是有效的。与基于规则的方法不同,此方法包含大量精心设计的句法模式。因此它具有高召回率和低精度。这种设计好的模式很可能是可以广泛应用的,并且具有领域依赖。最著名的词汇-句法模式框架之一是Text2Onto。Text2Onto 是将机器学习方法与基本语言方法相结合,例如标记化和词性标记(POS)。这种方法存在不准确性和领域依赖性。Naresh等人提出了一个框架,用于从使用预定义字典的文本构建本体。他们的方法的缺点包括构建和维护综合字典的人工成本。最后,甚至手动创建了生成的本体。基于机器学习的方法使用各种监督和非监督方法来自动化本体生成任务。[18–22]中的研究提出了他们基于有监督学习方法的本体生成方法。Bundschus[18]等人的方法着眼于提取疾病、治疗方法以及基因之间的相互作用关系,而Fortuna [19]等人使用SVM主动监督学习方法来提取领域概念和实例。Cimiano [20]等人的研究基于形式概念分析方法结合自然语言处理的监督方法,从各种数据源中提取分类关系。Poesio[21]等人提出了一种基于核方法的监督学习方法,该方法仅利用了浅层的语言信息。Huang[22]等人提出了一种监督方法,它使用预定义的句法模式和机器学习来检测来自维基百科文本的两个实体之间的关系。这些有监督的机器学习方法的主要缺点是它们需要大量的训练数据和手动标记。这通常是耗时,昂贵且劳动密集的。因此,在[23, 24]中提出的无监督方法很少,[23]Legaz-García等人使用聚类构建概念的层次结构,并通过本体对齐来生成符合OWL格式的正式规范的输出。而Missikoff[24] 等人提出了一种无监督的方法,它结合了基于语言和统计的方法,从文本中执行自动本体生成的任务。
最近,已经提出了一些使用知识库来自动化本体构建的方法。例如Harris 等人利用自然语言处理和知识库,从原始文本构建本体知识结构。他们提出的方法使用预定义的概念词典来提取本体知识的“障碍类型概念”,例如可能在文本中出现的UMLS。另外,为了提取层次关系,他们使用句法模式来促进提取的过程。他们方法的缺点包括构建字典的人工成本,领域的特定性,以及模式的有限性。使用知识库方法的另一种尝试由Cahyani[25]等人提出,他们的方法使用受控词汇表来构建阿尔兹海默症的领域本体,并将阿尔兹海默症文本的语料作为输入。本研究使用Text2Onto工具识别概念和关系,并使用基于字典的方法过滤它们。此外,这项工作使用链接的数据模式映射来识别最终的概念和候选关系。这种方法产生了一些限制:疾病的特异性,需要与感兴趣的领域相关的预定义的字典,并且在概念和关系提取期间不考虑术语的语义。此外,Qawasmeh [27]提出的是一种半自动的引导方法,包括手动进行文本的预处理,概念提取以及LOD的使用,以抽取关系和类的实例。他们的方法的缺点在于都需要领域的专家以及在开发过程中涉及重要的手工劳动。表1显示了我们提出的方法和现有的基于知识的方法的比较。
表1

尽管在本体构建领域不断努力和进行了许多的研究,但本体生成和自动化的过程仍存在很多的挑战。这些挑战包括概念发现,分类关系的提取(定义一个概念层次)和非分类的关系。通常,本体是手动创建的,需要熟悉本体构建理论和实践的领域专家和本体工程师。一旦构建了本体,不断发展的知识和应用需求就需要不断的维护工作。此外,过去十年中,数据量的急剧增加使得几乎不可能在合理的时间限制下将所有现有数据手动转化为知识。在本文中,我们提出了一个名为“基于链接开放数据的自动生物医学本体生成框架”(LOD-ABOG),它可以立即解决上述的每个挑战,克服领域特定本体手动构建的高成本,转换大量数据,实现领域的独立性,并且实现高度的领域覆盖。
我们所提出的框架使用知识库(UMLS)和LOD(Linked life Data [34, 35] BioPortal [36]),使用了混合的方法以准确识别生物医学概念;利用LOD,将语义丰度用简单、简洁的方式提取更多的概念;使用广度优先搜索算法(BFS)来导航LOD存储库,并且创建高精度分类,并生成一个定义良好的本体,以满足W3C语义的Web标准。此外,我们提出的框架是专门为生物医学领域设计和实施的,因为它是围绕生物医学知识库(UMLS和LOD)建立的。并且,概念检测模块也将使用生物医学专用的知识库UMLS进行概念检测。当然,也可以将其扩展到非生物医学领域。因此,我们会在未来的工作中增加对非医疗领域的支持。
本文回答了一下的研究问题:LOD是否足以提取概念,以及生物医学文献中的概念之间的关系(例如Medline/PubMed)?使用LOD以及基于UMLS和Stanford API等传统技术进行概念提取会产生什么影响?虽然,LOD可以帮助提取层次关系,我们如何能够为结果本体有效地建立非层次关系?与自动化OntoGain框架或者手动构建本体相比,我们提出的方法在精度,召回率以及F值等方面的表现是什么?
与现有的基于知识的方法相比,我们的主要贡献如下:
- 为了克服弱点,并且提高当前自动化和半自动化方法的质量,我们提出的框架整合了自然语言处理以及语义丰富技术来准确的检测概念;使用语义相关性进行概念消歧,将图搜索算法应用于三元组的挖掘,并且采用语义丰富技术来检测概念之间的应用。我们所提出的框架的另一个新颖的方面是Freepal的使用:用于关系提取的大量模式以及模式匹配算法,以提高非分类关系提取的准确性。此外,我们提出的框架通过使用提出的NLP方法以及基于知识的方法,具有从大量生物医学文献中进行大规模知识提取的能力。
- 与产生的概念、属性以及关系的集合的现有方法不同,我们提出的框架将生成定义明确的正式本体,其具有从现有的知识创建新知识的推理能力。
方法
我们从生物医学文献生成本体的方法如图1所示。表2给出了所有LOD-ABOG模块的简要描述。
图1
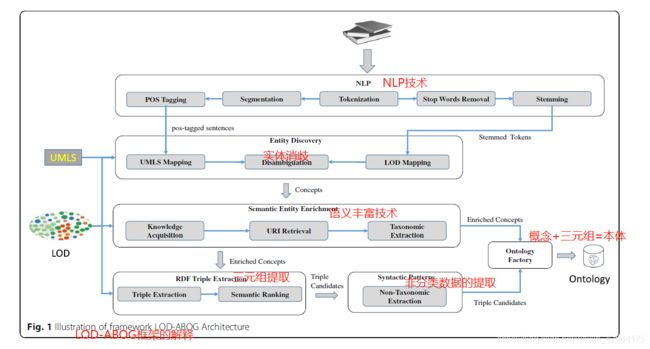
表2

NLP模块
NLP模块旨在分析,解释和操纵人类语言,已实现类似人类的语言处理。NLP模块的输入是来自MEDLINE/PubMed资源的非结构化生物医学文献。LOD-ABOG框架的NLP模块使用Stanford NLP API来计算句子的语法结构并执行标记化,分割,词干化,停用词删除和词性标注(POS)。算法1-文本处理显示了NLP模块的伪代码,分割是识别句子边界的任务(第3行),词性标注是为每个单词分配明确的词汇类别的过程(第4行)。标记化是将变形形式转换或删除为公共单词的形式(第6行)。例如,‘jumped’和‘jumps’被改为root术语‘jump’。停用词的移除是删除最常用单词的过程,例如‘a’和‘the’(第6行)。
算法1

实体发现模块
实体发现模块是我们提出的框架的主要构件模块之一。实体发现模块的主要任务是识别自由文本中的生物医学概念,应用n-garm算法,并且实现概念消歧。识别生物医学概念是一项具有挑战性的任务,我们通过将每个实体或者符合实体映射到UMLS概念和LOD类来攻克这些任务。算法2是实体检测和显示实体发现模块的伪代码。为了实现实体和UMLS概念ID之间的映射,我们使用了MetaMap API,它提出了一种基于计算语言技术的知识密集型方法(第3-5行)。为了执行实体和LOD类之间的映射,算法2执行了3个步骤:a)从句子中排除停用词和动词(第6行);b)它使用n-garm方法,识别了多词的实体(例如糖尿病,颅内动脉瘤),窗口大小在unigram和8-gram范围内(第7行);c)之后,它使用owl:类和skos:概念谓词(第9-13行)来查询LOD以识别概念。例如,算法2将Antiandrogenic 视为概念,如果LOD中有一个三元组,例如三元组“bio: Antiandrogenic rdf:type owl:Class”或者“bio: Antiandrogenic rdf:type skos:Concept”,其中bio是与本体相关的命名空间。我们的详细分析表明,使用UMLS和LOD(LLD或BioPortal)作为混合解决方案可以提高实体发现的准确性和召回率。但是,使用LOD发现概念有一个共同引用的问题,当单个URI标识多个资源时会发生该问题。例如,LOD中的许多URI用于标识单个坐着,实际上,有许多人具有相同的名称。在生物医学领域,“普通感冒”概念可能与天气或者疾病相关。因此,我们通过自适应Lesk算法来识别概念之间的语义相关性,将概念消歧应用于识别正确的资源(第15-17行)。基本上,我们使用概念的定义来测量与文本中其他发现的概念定义相重叠,然后我们将选择满足阈值并具有高重叠性的概念。
算法2

语义实体丰富技术模块
为了提高本体生成中的语义交互性,语义丰富模块旨在通过将概念与LOD中定义的相关概念相关联,能够自动地将标准的语义来丰富概念以及隐式的相关资源。语义实体丰富模块通过实体发现模块读取所有发现的概念,并且使用了可以被机器处理的其他明确定义的信息来丰富每个概念。图2中给出了语义实体丰富输出的示例,并且算法3给出了用于语义实体丰富模块的伪代码。
图2

算法3

我们提出的语义丰富过程总结如下:
- 算法3采用了由算法二提取出来的概念和λ (图中祖先的最大级别)作为输入(第1行)
- 对于LOD中具有谓词的三元组(label, altlabel, preflabel)(第6-19行)
2.1 应用精确匹配(输入概念,谓词的值)(第8-12行)
2.1.1将三元组提取为‘altlabel 或/和 preflabel’
2.2 通过查询skos从LOD中找出概念的定义:定义以及skos:关于优选资源的注释(第13-15行)
2.3 通过分析URIs来识别已定义概念的关系(第16行)
2.4 通过将概念映射到UMLS语义类型来获取概念的语义类型,由于一个概念可能映射到多个语义类型,因此我们会考虑所有的语义类型(第17行)
2.5 获取概念的层次结构,这是一项具有挑战性的任务。在我们提出的框架中,我们使用图算法,就因为我们将LOD视为大的有向图。广度优先搜索用于遍历具有skos:broader或owl:subclass或者skos:narrower edge的节点。它允许由输入λ 能够控制多级的层次结构(第18行)。
RDF三元组提取模块
RDF三元组提取模块的主要目标是识别LOD中明确定义了的三元组,它表示输入生物医学文本中两个概念之间的关系。我们提出的方法使用图表方法为三元组挖掘提供了一种独特的解决方案,测量LOD中现有的三元组相关性,以及生成了候选的三元组。
在我们提出的算法4三元组提取中,广度优先搜索的图形调用的深度是可以配置的,并且同时提供可伸缩性和效率。我们在第4行将深度设置为最佳值5,以获得最佳结果和性能。第5行使用BFS算法检索描述源输入概念的所有三元组。算法4仅考虑表示两个不同概念的三元组。第7-18行中的代码通过匹配标签、同义词、重叠定义和重叠层次结构来衡量相关性。为了尽可能的增强三元组的提取,我们将匹配阈值设置为70%(算法4第13,15,17行)以在我们的评估中消除三元组的噪声。有关深度和阈值的更多详细信息,请参阅后面的讨论部分。
此外,该模块还有一个子任务,通过使用我们的算法URI_Ranking对给定概念的URI进行语义排序。通过资源匹配的标签或altlabel从LOD检索URI。例如,资源http://linkedlifedata.com/resource/ diseaseontology/id/DOID:8440 中diseaseontology/id/DOID:8440 就是针对给定概念“ileus”的检索。检索URI的主要挑战之一是当一个概念可以由多个URIs表示时,例如,概念“ileus”可以由多个表示(表3)。
为了解决这个问题,我们提出了算法URI_Ranking,用于根据每个概念的相关性对URI进行排序。更确切的说,对于给定的概念,目标是生成URI的排名,因此会为每个URI分配一个正的实际值,如果需要,可以从中进行序数排序。在一个简单的形式中,我们的算法URI_Ranking首先为每个每个URI分配一个数字进行加权,一个包含UMLS语义类型和组类型的特征向量。然后它测量与算法5中写的相同概念相关的每两个URI的向量之间的平均余弦相关性。最后,它将根据它们的数字加权对它们进行排序。
表3

语法模式模块
在我们提出的方法中,语法模式模块执行模式识别找到自由文本中的两个概念之间的关系,如图3所示。模式库是通过使用Freepal的观察者关系提取所有生物医学的模式而构建的。之后,我们请专家将获得的模式与他们与健康生命自会的观察者关系进行了映射。在表4中,我们提供了模式样本及其相应的观察关系和映射谓词。在下一阶段,我们开发一种算法,该算法读取了句子,循环遍历所有的模式,应用解析,然后将匹配的模式转换为三元组。该算法利用语义丰富信息,例如如果模式和句子中任何发现的概念不匹配,则使用概念同义词,这将导致召回率的增加,重要的是该算法不区分大小写。
图3
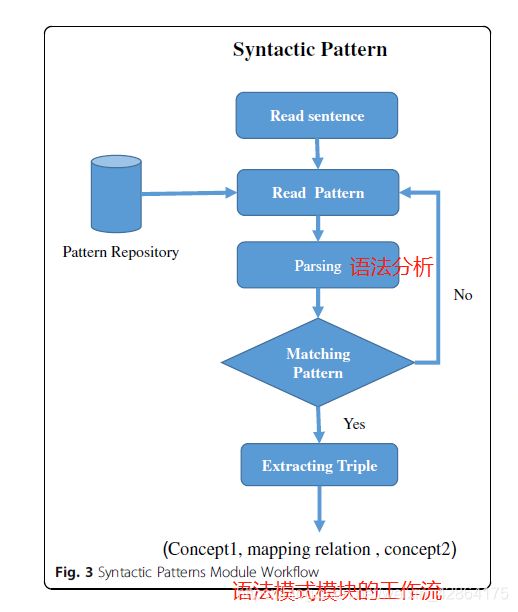
表4

本体库
该模块在我们提出的框架中起着核心的作用,在我们的框架中,它使用诸如RDF,RDFS,OWL和SKOS之类的本体语言自动化编码语义丰富信息的过程,并使候选三元组变成本体。我们在Open Biomedical Ontologies(OBO)格式中选择了W3C规范本体,因为它们为语义Web提供了明确定义的标准,从而加速了本体的开发和维护。此外,它们支持基于规则来推断复杂的属性。我们提出的框架生成本体的一个例子如图4所示。
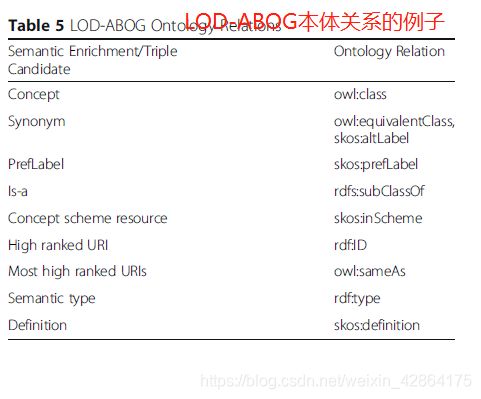
在本体库的背景下,有两个输入需要生成类,属性,is-a关系和关联关系。这两个输入是:1)来自语义丰富模块的概念语义丰富,2)来自RDF三元组提取以及句法模式模块的候选三元组。可以使用语义丰富技术来生成许多关系。最初,通过使用获得的概念简单地声明命名类来定义特定领域的根类。使用表示概念排名最高的URI为每个获得的类定义类标识符(URI引用)。在定义每个获得的概念的类之后,再定义其他的语义关系。例如,概念可以具有父概念和子概念,提供可以使用获得的层次关系定义的属性rdfs:subClassof。另外,如果概念具有同义词,则给出等价的定义,“preflabel”属性将用来获得优选概念, “inscheme”属性将用来获得关系。表5给出了LOD-ABOG生成关系的几个例子。
图4
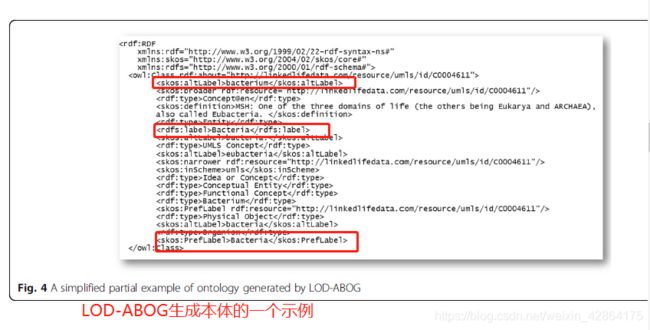
表5
评估
我们提出的方法提供了一个由LOD驱动的新颖、简介、简单的框架。我们使用了三种不同的本体演化方法来评估我们的自动本体生成框架。首先,我们开发并使用我们的自动化生成生物医学本体算法,使用CDR语料库和SemMedDB基于任务评估我们的框架。其次我们使用了阿尔兹海默症本体作为baseline进行了基于本体评估。第三,我们将我们提出的框架与成为“OntoGain”的最先进的本体学习框架进行了比较,我们使用了Apache Jena框架,这是一个提供丰富的交互式工具的开发环境,我们使用了4核的Intel® Core™ i7-4810MQ CPU @ 2.80 GHz和64位的Java JVM进行了实验。此外,在我们的评估过程中,我们发现尸体可以包含单个概念词或者多词的概念。因此,我们只考虑长概念匹配而忽略了短概念以提高精度。此外,我们还发现了一个限制,因为实体和缩写的数量很大,所有实体都无法映射到UMLS中的概念ID。例如,实体“Antiandrogenic”在UMLS中没有概念ID,为了解决这个问题,我们考虑了基于LOD的技术。此外,我们引用了不同的窗口大小,范围从1-8作为了N-gram方法的输入。然而,我们发现窗口大小等于4的时候是最佳的,因为其他值降低了实体检测模块的性能,召回率很低,并且当窗口大小小于4时的平均精度。另一方面,召回率增加时窗口大小大于4但精度非常低。
数据集
对于任务基础评估,首先我们使用CDR Corpus标题作为实体发现和评估的输入和标准:带注释的CDR语料库包含1500个PubMed标题的化学品,疾病和化学诱导的疾病关系,其中2017年医学主题标题(Mesh Synonym) 已被用作同义词提取和评估的标准。此外,我们还使用了疾病本体(DO)和生物利益化学实体(ChEBI)为CDR中所有发现的概念手动建立更广泛层次关系的黄金标准。
另一方面,我们使用DISEASE/TREATMENT实体数据集之间的关系作为非等级关系发现评估的标准。接下来,对于任务库评估,我们下载了语义MEDLINE数据库(SemMedDB)版本31,2017年12月发布,这是一个生物医学语义预测的存储库,由NLP程序SemRep从MEDLINE摘要中提取。我们从SemMedDB构建了基准数据集。数据集由50000个句子组成,代表SemMedDB中存在的所有关系类型。此外,我们为每个语义预测提取了所有语义预测和实体,并且分别将它们用作关系提取和概念提取评估的基准。
对于本体的基准评估,我们从2017年1月至2018年4月期间发布的MEDLINE引文中选择了40000个与“阿尔兹海默症”领域相关的标题。此外,我们提取了阿尔兹海默症本体的子图。从阿尔兹海默症本体中提取子图的过程是通过以下步骤完成的:a)我们从Bioportal下载了完整的阿尔兹海默症本体作为OWL文件,b)使用Jena API将OWL文件上传为模型图,c)检索到与实体“阿尔兹海默症”匹配的概念,d)在步骤c中检索的属性(同义词)和提取的概念的关系。这个结果子图包含500个概念,1420个关系和500个属性(同义词)。
结果
为了评估我们提出的实体发现能力来分类上下文中提到的概念,我们注释了化学品和疾病的CDR语料库标题。在此评估中,我们使用精度,召回率和F测量值作为评估参数。精度是注释的正确的概念与注释概念总数的比值,等式1.然而召回率是注释的正确的概念与和标准中的正确概念的总数的比值,等式2。F值是精度和召回率的调和平均值,如公式3所示。
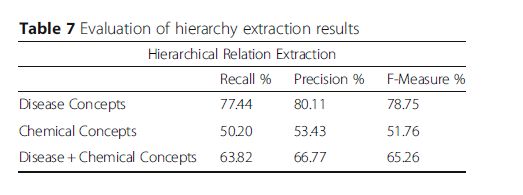
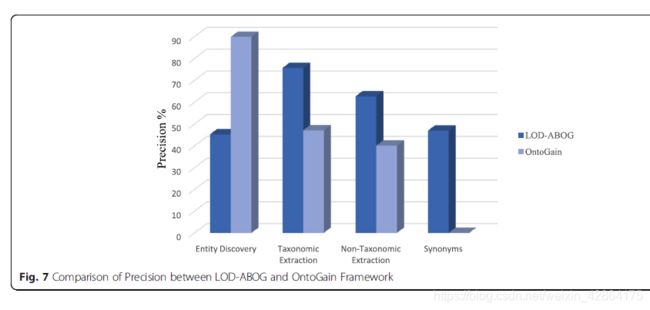
表6比较了MetaMap, LOD以及混合方法的精确度,召回率和F值。另外,非层次抽取的评估结果也将通过公式6的召回率来表示,精确度如公式7,F值如公式8表示。表8比较了非层次抽取的精度,召回率和F值。主要本体生成任务的结果在图5中以图形的形式进行了描述。然而,我们使用最先进的本体获取工具之一评估了我们提出的框架:即OntoGain。我们选择了OntoGain工具,因为它是最新的工具之一,已经使用医学领域进行了评估,输出的结果是OWL。图6和图7描述了我们提出的框架与使用召回率和准确率在OntoGain工具之间的比较,这些数字表明了LOD在本体生成中的有效性。
表6

表7
公式1-7

表8

图5

此外,我们将所提出的框架中生成的本体论与领域专家构建的阿尔兹海默症本体进行了比较。表9给出了比较的结果。结果表明,概念检测的F值为72.48%,关系抽取的F值为76.27%,属性抽取的F值为83.28%。这表明我们提出的框架的表现是让人满意的。但是,领域专家在验证阶段还可以进一步改进F值。表10比较了我们对SemMedDB的概念和关系的提取结果。
表9
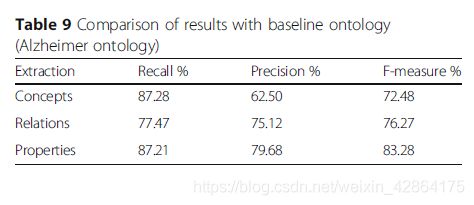
表10
讨论
我们的深度分析显示了LOD在自动本体生成中的有效性。此外,重新使用静心设计的本体将提高本体生成的准确性和质量。所有这些措施都解决了现有本体生成的一些缺点。此外,表6中的评价结果表明我们的概念发现方法非常好,并且与文献中报道的结果相匹配。但是,图6和图7显示OntoGain优于我们的概念发现方法。鉴于OntoGain仅考虑计算多词概念的精确度和召回率。在分层提取任务中,我们的层次结构提取比OntoGain具有显著的改进结果。同样,与OntoGain相比,我们对非分类学提取的句法模式方法提供了更好的结果。在算法4中,我们使用阈值参数δ来提高提取非层次关系的准确性。我们发现将δ值设置为低值时会产生很多噪声关系,而增加它会产生更好的准确度。但是,将δ设置为高于70%的值时,会产生较低的召回率。此外,我们使用深度参数γ来控制LOD中提取知识的深度。当γ在[1,2]范围内时,我们观察到较小程度的域覆盖,但当γ在范围内时,覆盖率逐渐提高[3,5]。然而,当γ> 5时,噪声数据增加得如此之快。
图6
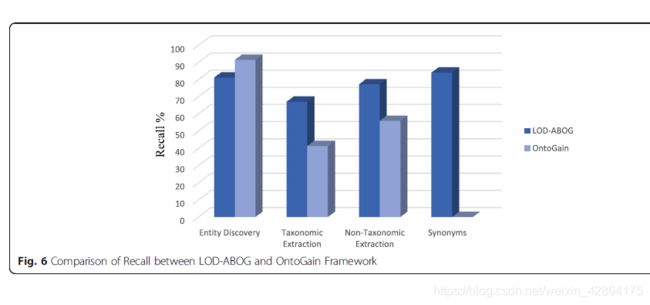
图7
虽然本体论中定义的关系是有限的,例如,疾病本体只定义了层次关系,很少有非层次关系被定义。这就像大多数现有的没有定义rdfs:domain等约束的本体一样,它有助于提高本体提取系统进行准确推理的能力。尽管Linked Open Data带来了好处,但由于其部分性能问题,它在工业互联网和医疗保健领域的应用未受到充分的欢迎。为了纠正它的缺陷,我们提出了一种使用广度优先搜索的图遍历方法,这种方法可以提高从一个节点移动到另一个节点的速度,而无需编写非常复杂的查询。如表10所示,与SemMedDB相比,概念提取和层次关系提取任务具有竞争力。然而,由于语法模式的限制,非层次结构提取显示了低的召回率,因此改进非层次结构的提取使我们未来工作的一部分。此外,领域专家在核查阶段可以进一步提高我们提出的框架的准确性和召回率。结果是令人兴奋的,表明我们可以缩小对劳动力的需求。此外,该框架将使专家能够以更有效的方式来执行本体工程。
结论
本体是语义网络愿景的基石。 此外,它提供了对特定域中的概念,重用域知识和数据互操作性的共同和共享理解。 然而,手动本体构建是一项复杂的任务,并且非常耗时。 因此,我们提出了一个完全自动化的本体生成框架,该框架由生物医学链接开放数据授权,集成了自然语言处理,语法模式,图算法,语义排序算法,语义丰富和RDF三元组挖掘,使自动大规模机器处理成为可能 ,最小化和缩小需求和复杂性,并提高本体生成的准确性。 本体不仅用于更好的搜索,数据互操作性和内容呈现,但更重要的是,它代表了管理休眠内容资产和将Web文档转换为Web of Data的未来创新方法的基础。
未来工作
我们未来的工作包括扩展框架以支持非生物医学领域本体生成。 此外,我们计划将机器学习和语义预测库(SemMedDB)集成到框架中,以进一步改进概念的F-度量和非层次关系提取。