R语言hdnom包进行高维惩罚 Cox 回归模型绘制列线图-校准曲线-时间依赖ROC-外部验证
Hdnom包可以用于用于给高维数据构建Cox 模型、绘制列线图-校准曲线-时间依赖ROC-外部验证,而且Hdnom包简化了建模过程,带有自动选择变量功能,将用户从繁琐且容易出错的调参过程中解放出来.
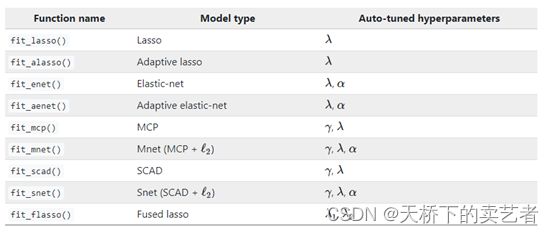
hdnom提供了多项自动调参和模型选择功能,包括以下模型类型:
下面我们通过数据来演示一下,我们先导入数据和R包,
library("hdnom")
smart<-read.csv("E:/r/test/tijian2.csv",sep=',',header=TRUE)
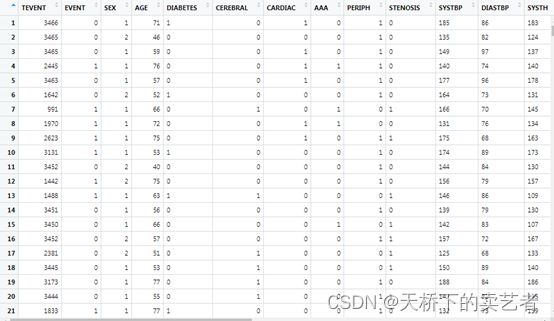
这是一个体检数据(公众号回复:体检数据2,可以获得数据),数据有29个变量,TEVENT:时间变量,EVENT:事件,这里是结局变量,SEX:性别,AGE年龄,DIABETES;糖尿病,CARDIAC:冠状动脉病史,AAA腹主动脉瘤。
和其他R包一样需把结局、时间、协变量分成不同的矩阵
x <- as.matrix(smart[, -c(1, 2)])
time <- smart$TEVENT
event <- smart$EVENT
y <- survival::Surv(time, event)
我们使用弹性网络需要加载doParallel包
suppressMessages(library("doParallel"))
registerDoParallel(detectCores())
建立模型,这里以弹性网络建立模型,nfolds交叉验证的折叠数,这里选10折,rule这里和cv.glmnet是一样的,这里选 lambda.1se,seed这里设置两个随机种子,parallel默认为F,选T的话要加载doParalle包
fit <- fit_aenet(x, y, nfolds = 10, rule = "lambda.1se", seed = c(5, 7), parallel = TRUE)
提取模型的一些数据,等下用到
model <- fit$model
alpha <- fit$alpha
lambda <- fit$lambda
adapen <- fit$pen_factor
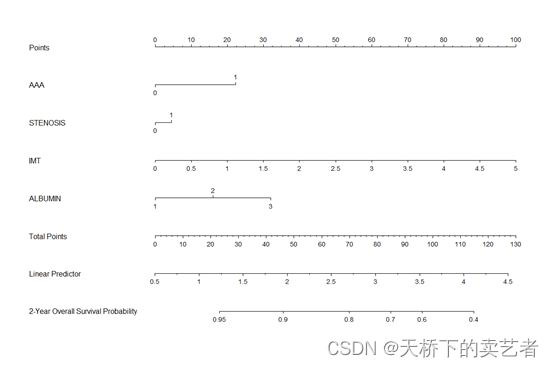
构建列线图
nom <- as_nomogram(
fit, x, time, event,
pred.at = 365 * 2,
funlabel = "2-Year Overall Survival Probability"
)
绘图
print(nom)
plot(nom)
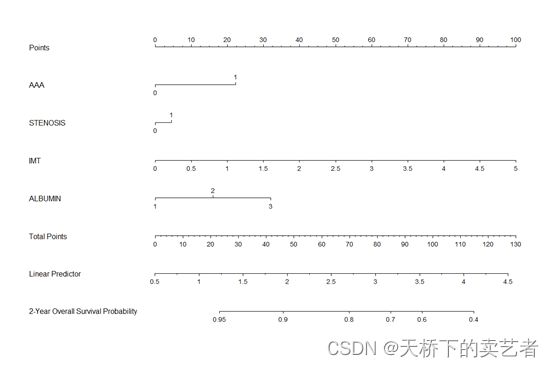
根据诺模图,自适应弹性网络模型从27个变量的原始集合中选择了5个变量,有效地降低了模型的复杂性。
接下来使用进行模型验证,先进行内部验证,使用validate函数进行
val_int <- validate(
x, time, event,
model.type = "aenet",
alpha = alpha, lambda = lambda, pen.factor = adapen,
method = "bootstrap", boot.times = 10,
tauc.type = "UNO", tauc.time = seq(1, 5, 0.5) * 365,
seed = 42, trace = FALSE
)
validate使用重采样的方法来验证惩罚Cox模型的预测性能,我们查看一下
print(val_int)

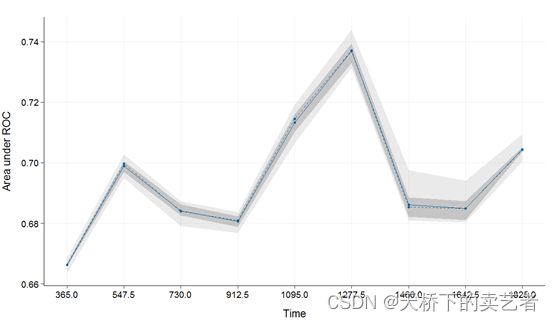
上图显示了重采样及模型的一些信息,以及tAUC的评估时间点,可以summary它显示具体数值
summary(val_int)

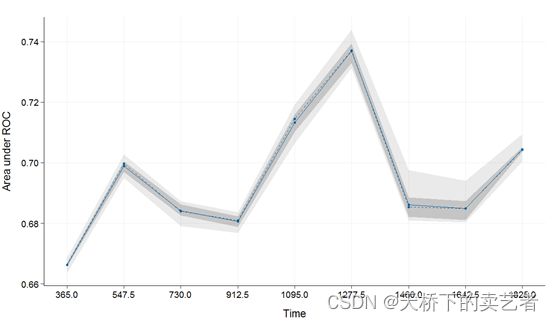
上图显示了每个时间点AUC的均值及分位数,我们还可以进一步绘图图示
plot(val_int)
下面进行外部验证,外部验证需要外部数据,我们从原数据抽一部分当外部数据
x_new <- as.matrix(smart[, -c(1, 2)])[1001:2000, ]
time_new <- smart$TEVENT[1001:2000]
event_new <- smart$EVENT[1001:2000]
,做好外部数据后,后面的步骤和之前一样
val_ext <- validate_external(
fit, x, time, event,
x_new, time_new, event_new,
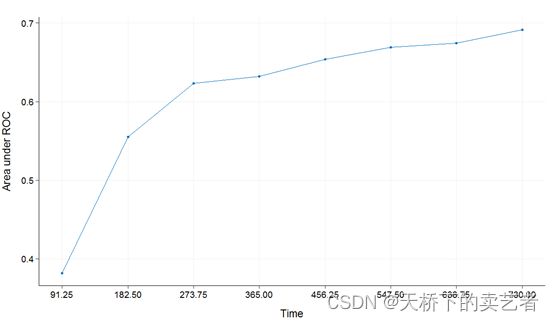
tauc.type = "UNO",
tauc.time = seq(0.25, 2, 0.25) * 365
)
summary(val_ext)
plot(val_ext)
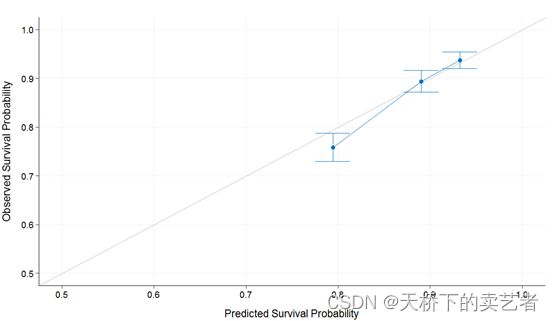
cal_int <- calibrate(
x, time, event,
model.type = "aenet",
alpha = alpha, lambda = lambda, pen.factor = adapen,
method = "bootstrap", boot.times = 10,
pred.at = 365 * 5, ngroup = 3,
seed = 42, trace = FALSE
)
plot(cal_int, xlim = c(0.5, 1), ylim = c(0.5, 1))
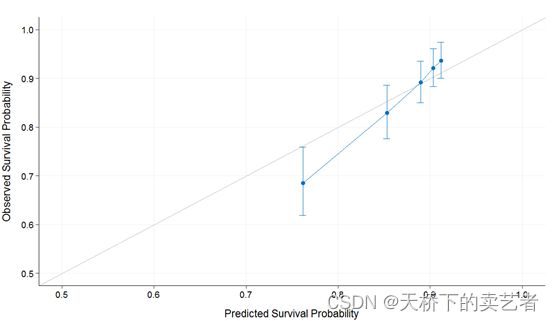
cal_int <- calibrate(
x, time, event,
model.type = "aenet",
alpha = alpha, lambda = lambda, pen.factor = adapen,
method = "bootstrap", boot.times = 10,
pred.at = 365 * 5, ngroup = 5,
seed = 42, trace = FALSE
)
plot(cal_int, xlim = c(0.5, 1), ylim = c(0.5, 1))
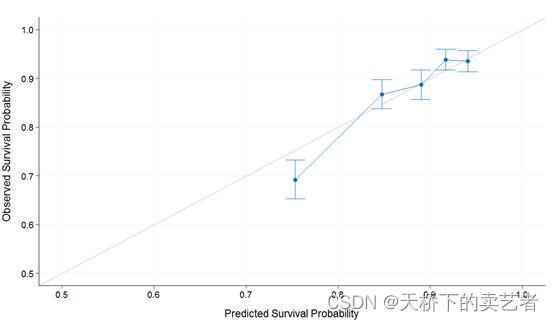
如果需要对外部数据进行校准,需要用到calibrate_external函数
cal_ext <- calibrate_external(
fit, x, time, event,
x_new, time_new, event_new,
pred.at = 365 * 5, ngroup = 5
)
plot(cal_ext, xlim = c(0.5, 1), ylim = c(0.5, 1))