细胞类型注释
细胞类型注释:
根据细胞的特征基因(marker基因)进行注释,一般情况是预先并不知道该特征基因对应的细胞类型,所以需要通过用细胞的特征基因对细胞类型进行注释,判断。
对于单细胞电脑分析可以进行数据过滤(quality control filtering), 基因比对结果定量(quantify the expression of each mapped gene),细胞聚类( identify groups of similar celld using clustering algorithm), 细胞聚类可视化( visulaize all cells in teo dimensions using techniques (t-distributed stochastic neighbor embedding, uniform manifold approximation and projection) )
细胞聚类后最关键的步骤就是细胞注释。在本指南中则对于细胞注释给出了guidline。
文中推荐的注释流程如下:自动注释,手动注释,证实。自动注释需要predefiend set of "marker genes"或者reference single-cell data (an exiting expertly annotated single-cell map)。手工注释则涉及了解基因以及基因针对于某一细胞的功能,进而辨析细胞簇和确定新的细胞类型。最后采用独立的方法去证实细胞的类型和功能。
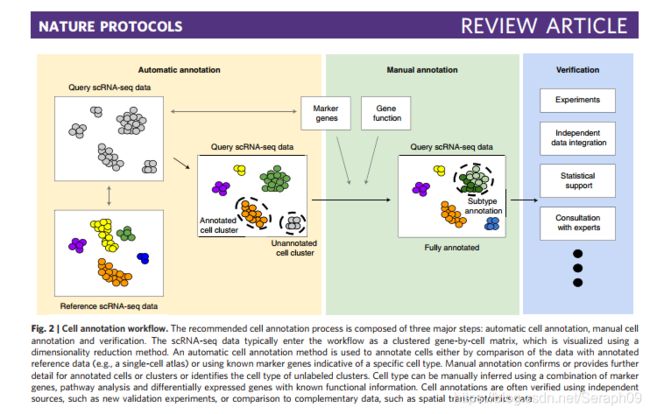
1. 自动注释:
自动注释就是利用算法和合适的先验数据集去标记细胞类型。通常的原则是确定单个细胞或者细胞簇中基因表达的特征(模式或者signature)
自动注释包括了基于marker(已知基因标记的细胞类型)的注释以及基于reference(参考基因集数据)的注释。每个标记也会给它一个置信度。
目前有两种主要的自动细胞注释方法,一种方法是利用已知的marker genes进行注释。在这种情况下,我们获得细胞类型和marker基因之间的关系,我们可以从数据库中获得这样的数据,例如:SCSig, PanglaoDB,以及CellMarker,或者从文献中获得。然后,细胞或者细胞簇就可以通过marker genes得到了区分。第二种方法是比较要进行注释的单细胞测序数据(query data set)到已知的,相似的,专家注释的单细胞测序数据(reference data set), 而参考基因数据来源于Gene Expresseion Omnibus(GEO), the Single Cell Expression Atlas或者cell atlas projects.
自动注释的方法可用于单个细胞(不管细胞是否聚类)和细胞簇。但是注释individual cell 是比较理想的状态,注释clusters更快,而且准确性更高。
但是对于自动注释而言,目前存在的问题是大多数细胞类型并没有well-charaterized gene expression signatures,进而导致注释并不准确。
自动注释对于主要的细胞类型比较适用,但是不能很好地区分细胞亚型。自动注释能够很好地确定已知的细胞类型,以及确定不能被注释的细胞雷影进行后续研究。
基于marker的注释
基于marker的注释对于well-characterized organisms and cells types(PBMc)比较好。基于marker的自动注释在具有大量相关的marker genes是比较使用的。
注释单个细胞,目前最为可信的工具是SCINA(semi–supervised category identificaiton and assignment)。SCINA假设每个marker gene都遵循bimodal gene expression distribution,也就是说该分布中的峰值对应细胞类型,而另一个峰值则是其它细胞。目前还有其它工具AUCell是另一个基于marker的注释工具,AUCell 将细胞表达的基因进行排序,然后细胞根据它们高表达的marker基因来判断细胞类型。AUCcell对于具有大量marker gene sets的细胞类型具有较好的注释结果。
为了给细胞簇进行注释,Gene Set Variation analysis(GSVA)很快。GSVA的作用方式和AUCell很相似,给marker gene sets数据库,根据基因表达富集的数据确定细胞类型。
基于marker的自动注释方法,通常在已知marker基因的细胞的注释效果更好。
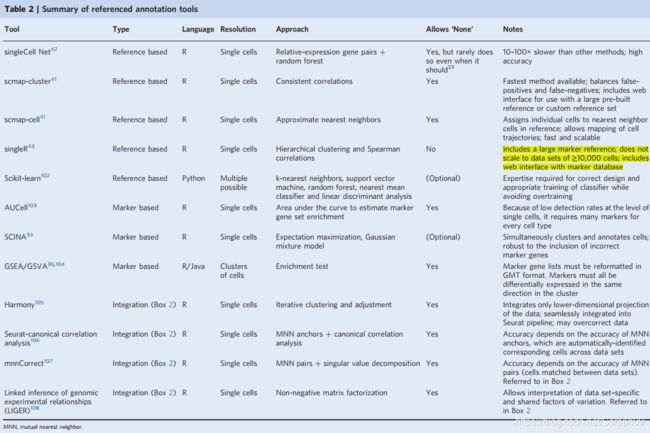
基于参考数据集的自动注释:
基于参考数据集的注释基于”guilt by association“这个理念,也就是说将参考数据集中的细胞或cluster标签转移到其它具有相似基因表达谱的未标记的细胞或cluster中。结果是,该方法适用于具有高质量和相关的注释参考单细胞基因数据集。( Tissue-specific
reference data can be obtained from public databases (e.g., the
GEO25 or the Expression Atlas26) or large cell atlas projects
(e.g., the Human Cell Atlas27, the Tabula Muris or Mouse Cell
Atlas5 or others4,28,38–40)。
scamp是一种基于reference-based的自动注释方法,其它的还有SingleCellNet42 and SingleR43. SingleCellNet has high accuracy when all cell types are well represented in the reference data but has low accuracy if the reference data are incomplete or represent a poor match
另外一种方法基于reference-based cell annotation 是将query data set 和参考数据集整合。
对于自动注释的数据,我们应该进行进一步的清理。
作者比较了marker的自动注释(marker来源不同,一个来源与scRNA-seq clustered derived, 一个来源于literature),结果发现基于marker的注释并不一致,见上图桑基图(Sankey图),发现marker注释出来的细胞类型并不相同。
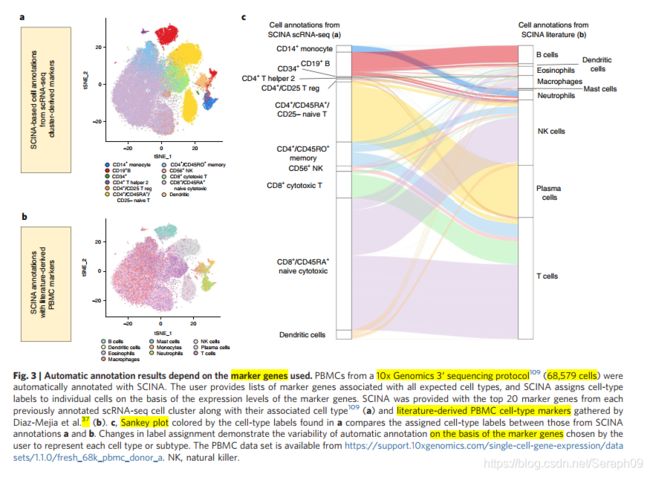
作者建议使用多个注释工具,但使用多个注释工具存在同一个细胞具有不同的cell type labels。解决这种标签冲突的方式有:
- confidence scores(打置信分数,但是每个置信分数并没有经过标准化,不太好评判)
- a majority-rule (标签大多数原则,出现最常见的标签为对的。)
- percent agreement between methods
2. 手动注释:经典的细胞marker, 来自于文献整理。
自动注释的置信度不高,细胞类型标签冲突,或者缺乏该细胞类型的注释的时候,需要采用专家手工注释。
手工注释主要是根据细胞功能注释的来源。手工注释主要在cluster层面进行注释。
通路富集分析(Pathway enrichment analysis)可以用于确定细胞簇的情况,利用GSVA或者(ssGSEA)进行分析。
手动注释有基于Marker的注释和基于基因数据集的注释
注释了细胞类型以后,要采用标准命名去注释细胞。(Cell Ontology)
3. 注释验证(annotation verification)
通过自动注释或手动注释,我们能够得到较为可信的cell-type labels,但是毕竟只是单纯的机器注释,我们要进一步确认注释结果的可靠性,可以通过统计学方法或者咨询专家获得更可靠的结果。
例如对于免疫细胞,我们可以使用TCR,BCR进行refined,对于肿瘤生物细胞,我们可以通过SNV(单核苷酸突变分析,复制子变异分析),可以使用工具HoneyBADGER,CaSpER,inferCNV。
scRNA-seq细胞注释中存在一些问题需要关注:
- 实验流程和分析参数的影响
实验流程:data filtering, data quality settings, select clustering resolution.
选择合适的clustering resolution对于准确的基因注释是非常关键的。
注释rare cell types可以采用Giniclust,但是这个方法可能会导致存在过度的overclustering。其它的方法例如scClustViz,Seurat和clustree可以帮助选择合适的clustering resolutions.
2.推荐的流程:
自动注释:需要编程技术,目前也有一些point-and-click的工具,例如:Loupe Browser, GSEA, Cerebro.
手动注释:建议在已知大类注释的前提下进行注释,目前细胞注释仍有很多挑战,例如:细胞亚型注释,gradients注释,高度同源的细胞注释,poorly defined cluster的注释。
基于marker基因的注释,如果基因没有被证实一致存在于这个细胞内,容易导致错误的结论