全文下载链接: http://tecdat.cn/?p=22482
在本文中,在R中拟合BRT(提升回归树)模型。我们的目标是使BRT(提升回归树)模型应用于生态学数据,并解释结果。
引言
本教程的目的是帮助你学习如何在R中开发一个BRT模型。
示例数据
有两套短鳍鳗的记录数据。一个用于模型训练(建立),一个用于模型测试(评估)。在下面的例子中,我们加载的是训练数据。存在(1)和不存在(0)被记录在第2列。环境变量在第3至14列。
> head(train)拟合模型
拟合gbm模型,你需要决定使用什么设置,本文为你提供经验法则使用的信息。这些数据有1000个地点,包括202条短鳍鳗的存在记录。你可以假设:1. 有足够的数据来建立具有合理复杂性的相互作用模型 2. 大约0.01的lr学习率可能是一个合理的初始点。下面的例子显示如何确定最佳树数(nt)。
step(data= train, x = 3:13,
family = "bernoulli", comp = 5,
lr = 0.01, bag.fr = 0.5)对提升回归树模型进行交叉验证优化。
使用1000个观测值和11个预测因子,创建10个50棵树的初始模型。
上面我们使用了交叉验证的。我们定义了:数据;预测变量;因变量--表示物种数据的列号;树的复杂度--我们首先尝试树的复杂度为5;学习率--我们尝试用0. 01。
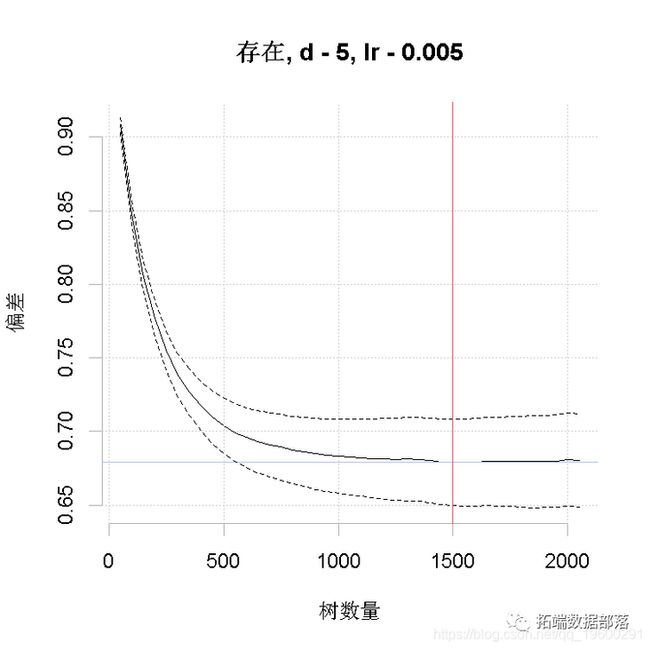
运行一个如上所述的模型,将输出进度报告,做出图形。首先,你能看到的东西。这个模型是用默认的10倍交叉验证法建立的。黑色实心曲线是预测偏差变化的平均值,点状曲线是1个标准误差(即在交叉验证上测量的结果)。红线表示平均值的最小值,绿线表示生成该值的树的数量。模型对象中返回的最终模型是在完整的数据集上建立的,使用的是最优的树数量。
length(fitted)返回的结果包含 fitted - 来自最终树的拟合值,fitted.vars - 拟合值的方差, residuals - 拟合值的残差,contribution - 变量的相对重要性。statistics - 相关的评估统计量。cv.statistics 这些是最合适的评估统计数据。
我们在每个交叉验证中计算每个统计量(在确定的最佳树数下,根据所有交叉验证中预测偏差的平均变化进行计算),然后在此呈现这些基于交叉验证的统计量的平均值和标准误差。weights - 拟合模型时使用的权重(默认情况下,每个观测值为 "1",即权重相等)。trees. fitted - 阶段性拟合过程中每一步所拟合的树的数量记录;training.loss.values - 训练数据上偏差的阶段性变化 ,cv.values - 阶段性过程中每一步所计算的预测偏差的CV估计值的平均值。
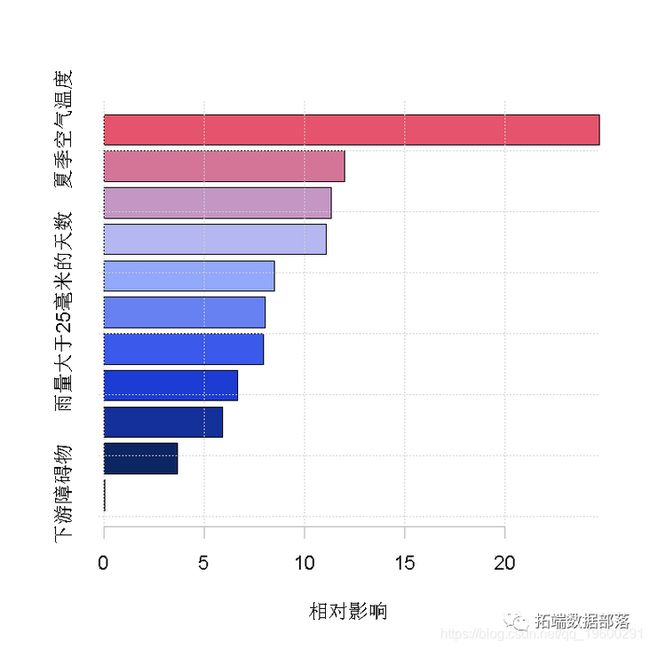
你可以用摘要函数查看变量的重要性
> summary(lr )选择设置
以上是对设置的初步猜测,使用了Elith等人(2008)中讨论的经验法则。它做出的模型只有650棵树,所以我们的下一步将是减少lr。例如,尝试lr = 0.005,争取超过1000棵树。
step(data=train, x = 3:13,
tree.co = 5,
+ lr = 0.005为了探索其他设置是否表现更好,你可以将数据分成训练集和测试集,或者使用交叉验证结果,改变tc、lr和bagging,然后比较结果。
简化模型
简化会建立了许多模型,所以它可能很慢。在其中,我们评估了简化lr为0.005的模型的价值,但只测试剔除最多5个变量("n.drop "参数;默认是自动规则一直持续到预测偏差的平均变化超过gbm.step中计算的原始标准误差)。
对于我们的运行,估计要剔除的最佳变量数是1;可以使用红色垂直线指示的数字。现在,建立一个剔除1个预测变量的模型,使用[[1]]表示我们要剔除一个变量。
点击标题查阅往期内容
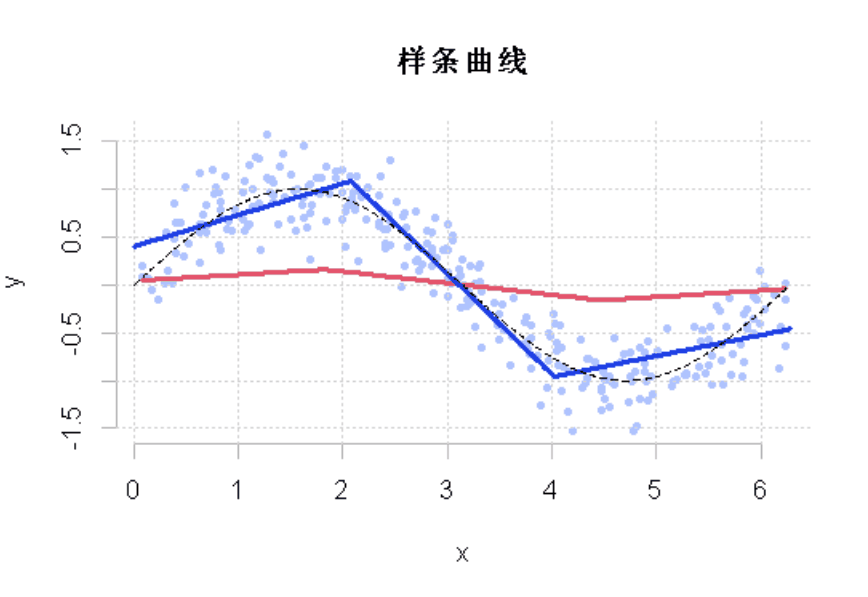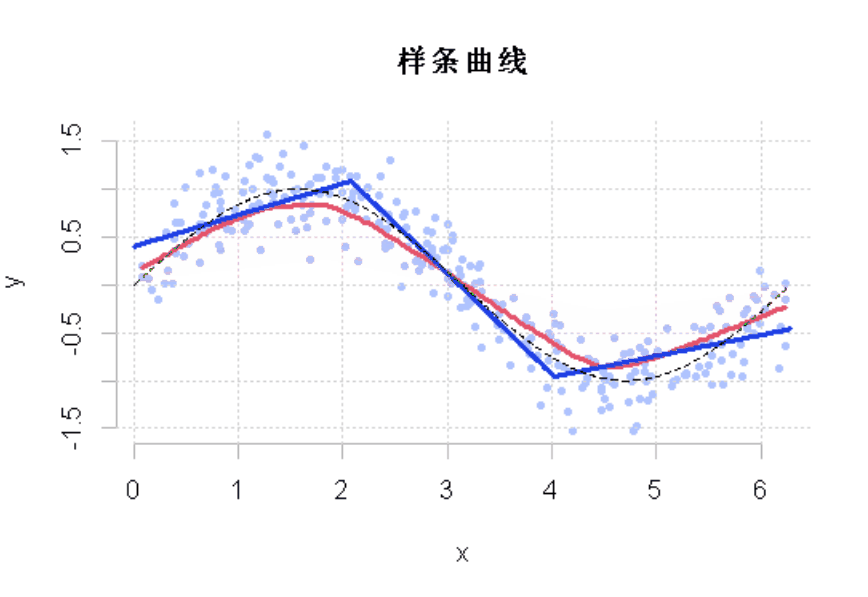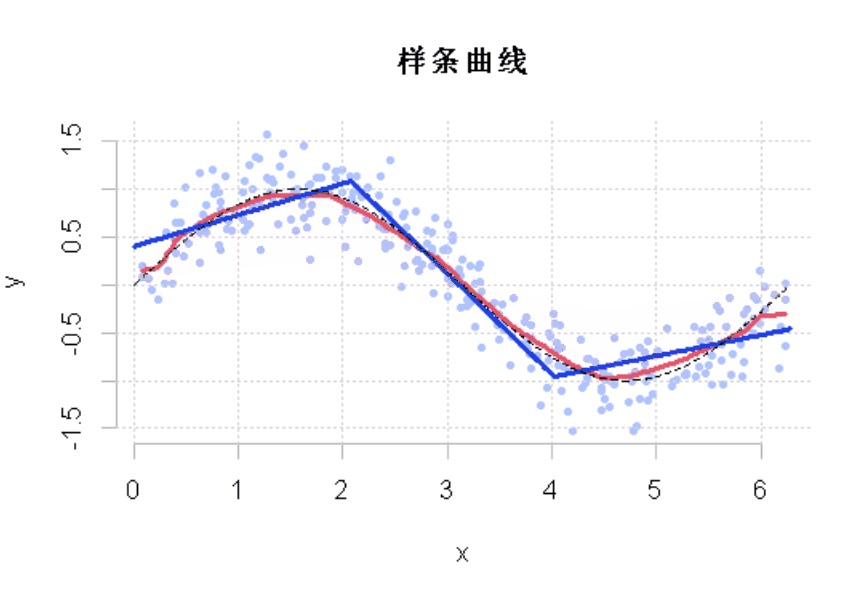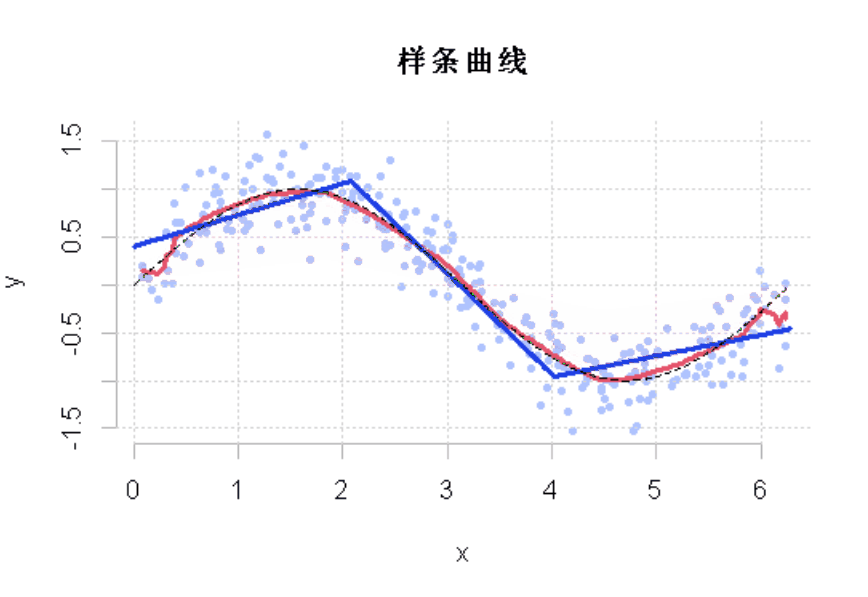
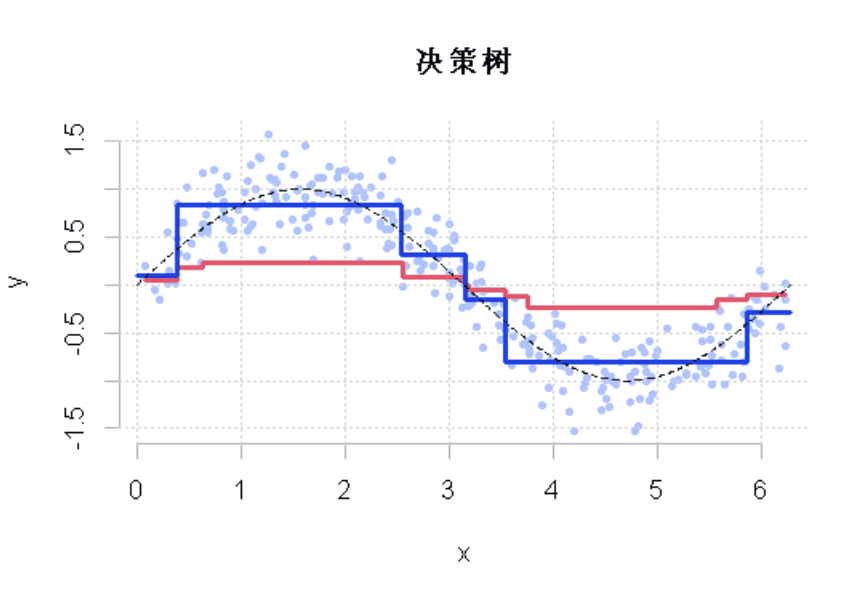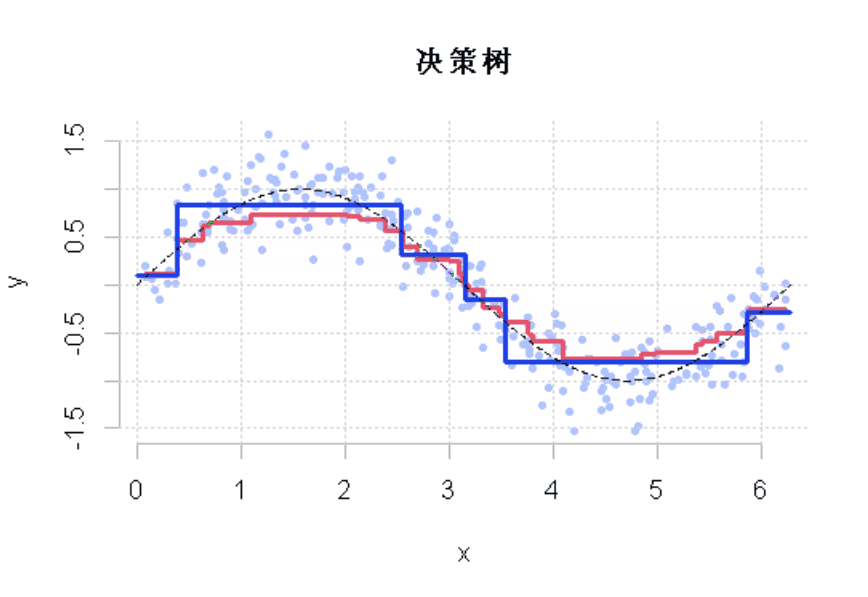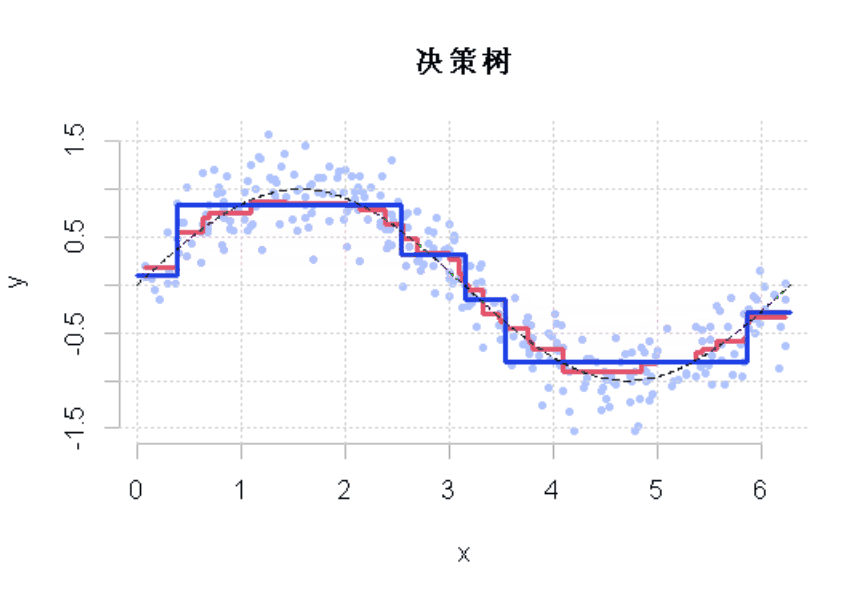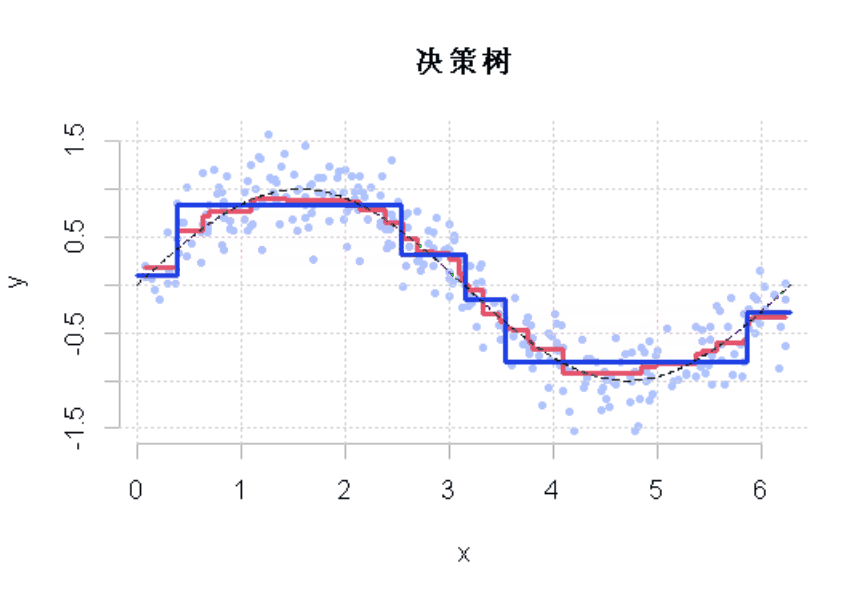
R语言样条曲线、决策树、Adaboost、梯度提升(GBM)算法进行回归、分类和动态可视化
左右滑动查看更多
01
02
03
04
step( x= pred.list[[1]], )现在这已经形成了一个新的模型,但是考虑到我们并不特别想要一个更简单的模型(因为在这种规模的数据集中,包含的变量贡献很小是可以接受的),我们不会继续使用它。
绘制模型的函数和拟合值
由我们的函数创建的BRT模型的拟合函数可以用plot来绘制。
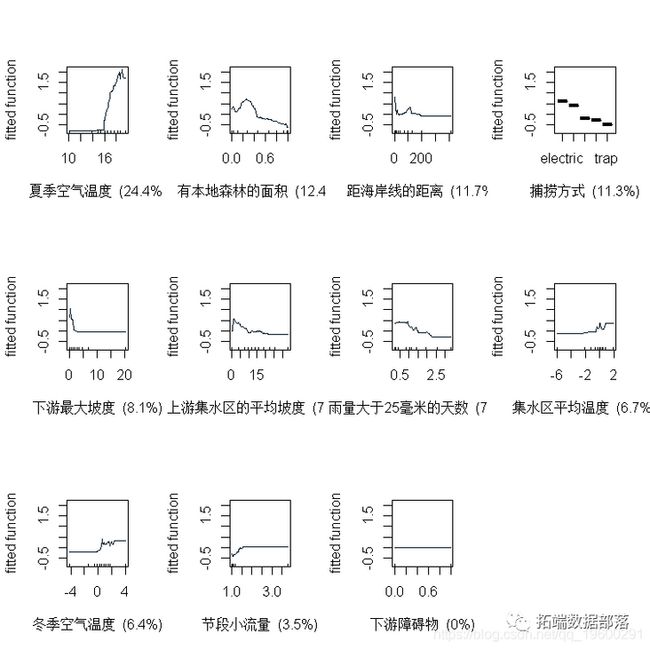
> plot( lr005 )这个函数的附加参数允许对图进行平滑表示。根据环境空间内观测值的分布,拟合函数可以给出与每个预测因子有关的拟合值分布。
fits( lr005)每张图上方的数值表示与每个非因素预测因子有关的拟合值的加权平均值。
绘制交互作用
该代码评估数据中成对的交互作用的程度。
inter( lr005)返回一个列表。前两个部分是对结果的总结,首先是5个最重要的交互作用的排名列表,其次是所有交互作用的表格。
f$intera你可以像这样绘制交互作用。
persp( lr005, z.range=c(0,0.6)对新数据进行预测
如果您想对一组地点进行预测(而不是对整个地图进行预测),一般的程序是建立一个数据框架,行代表地点,列代表您模型中的变量。我们用于预测站点的数据集在一个名为test的文件中。"列需要转换为一个因子变量,其水平与建模数据中的水平一致。使用predict对BRT模型中的站点进行预测,预测结果在一个名为preds的向量中。
preds <- predict(lr005,test,
deviance(obs=test, pred=preds)![]()
> d <- cbind(obs, preds)
> e <- evaluate(p=pres, a=abs)gbm中预测的一个有用的特点是可以预测不同数量的树。
tree<- seq(100, 5000, by=100)
predict( n.trees=tree, "response")上面的代码会形成一个矩阵,每一列都是模型对tree.list中该元素所指定的树数量的预测,例如,第5列的预测是针对tree.list[5]=500棵树。现在来计算所有这些结果的偏差,然后绘制。
> for (i in 1:50) {
calc.devi(obs,
+ pred[,i])
+ }
> plot(tree.list,deviance空间预测
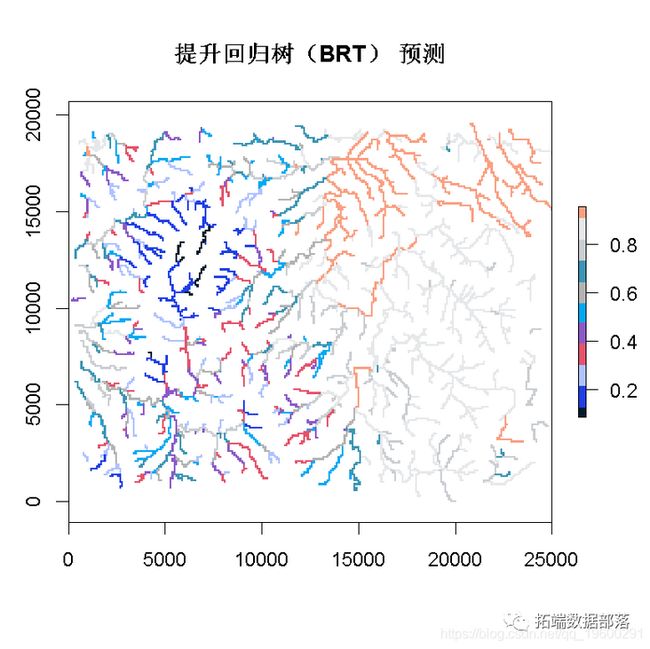
这里我们展示了如何对整张地图进行预测。
> plot(grids)我们用一个常量值("因子 "类)创建一个data.frame,并将其传递给预测函数。
> p <- predict(grids, lr005,
> plot(p)本文摘选 《 R语言生态学建模:增强回归树(BRT)预测短鳍鳗生存分布和影响因素 》 ,点击“阅读原文”获取全文完整资料。
点击标题查阅往期内容
Python决策树、随机森林、朴素贝叶斯、KNN(K-最近邻居)分类分析银行拉新活动挖掘潜在贷款客户
R语言逻辑回归(Logistic Regression)、回归决策树、随机森林信用卡违约分析信贷数据集R语言基于Bagging分类的逻辑回归(Logistic Regression)、决策树、森林分析心脏病患者
R语言样条曲线、决策树、Adaboost、梯度提升(GBM)算法进行回归、分类和动态可视化
R语言用主成分PCA、 逻辑回归、决策树、随机森林分析心脏病数据并高维可视化
matlab使用分位数随机森林(QRF)回归树检测异常值
R语言用逻辑回归、决策树和随机森林对信贷数据集进行分类预测
R语言中使用线性模型、回归决策树自动组合特征因子水平
R语言中自编基尼系数的CART回归决策树的实现
Python对商店数据进行lstm和xgboost销售量时间序列建模预测分析
R语言基于树的方法:决策树,随机森林,Bagging,增强树
R语言实现偏最小二乘回归法 partial least squares (PLS)回归
R语言多项式回归拟合非线性关系
R语言逻辑回归(Logistic回归)模型分类预测病人冠心病风险
R语言用局部加权回归(Lowess)对logistic逻辑回归诊断和残差分析
R语言混合效应逻辑回归(mixed effects logistic)模型分析肺癌数据