易基因:全基因组DNA甲基化测序(WGBS)揭示儿童哮喘增强子区域的整体低甲基化|科研速递
大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
哮喘(asthma)是儿童期最常见的慢性炎症性疾病,患病率2.6%~30.5%,儿童哮喘是世界范围内的一个主要健康问题。儿童哮喘是遗传和环境因素复杂互作结果,可导致表观遗传和免疫失调、气道炎症和肺功能受损。先前研究利用850K芯片对哮喘的DNA甲基化变化的靶点特异性分析或DNA甲基化微阵列进行了描述,覆盖人类基因组约28M CpG中的27K-850K个CpG位点。由于850K芯片对基因组覆盖范围有限,目前对儿童哮喘增强子区域(enhancer region)失调知之甚少,因此表观遗传学调控在哮喘发展中的影响仍然广泛未知。
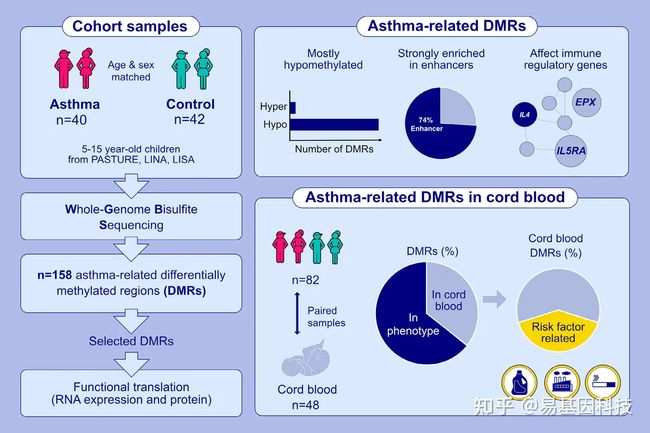
2023年1月27日,德国肺病研究中心(DZL)Irina Lehmann教授团队在《Allergy》杂志发表题为“Global hypomethylation in childhood asthma identified by genome-wide DNA-methylation sequencing preferentially affects enhancer regions”的研究论文,该研究利用全基因组重亚硫酸盐测序(WGBS)对三个独立前瞻性出生队列的40名哮喘儿童与42名性别和年龄匹配对照的的全血样本进行DNA甲基化比较分析,鉴定出增强子区域DNA甲基化显著失调是儿童哮喘的重要标记。
标题:Global hypomethylation in childhood asthma identified by genome-wide DNA-methylation sequencing preferentially affects enhancer regions(全基因组DNA甲基化测序鉴定儿童哮喘的整体低甲基化优先靶向增强子区域)
发表期刊:Allergy
发表日期:2023年01月27日
影响因子:IF 12.4
技术:全基因组重亚硫酸盐测序(WGBS)等
样品及方法:
- 三个独立前瞻性出生队列的40名哮喘儿童与42名性别和年龄匹配对照的的全血样本进行全基因组重亚硫酸盐测序(WGBS)分析。
- 已鉴定的差异甲基化区域(DMRs)分为基因型相关、细胞类型依赖或产前预激。
- DMR相关基因的网络分析和随后的自然语言处理(natural language processing,NLP)在转录和蛋白质水平上表观遗传调控功能翻译的靶向分析得到补充。
摘要
本研究应用无偏好性的全基因组重亚硫酸盐测序(WGBS)来表征哮喘儿童与健康对照组相比的整体DNA甲基化谱。WGBS分析结果表明与对照组相比,哮喘儿童全血样本中共鉴定出158个DMR,其中37%与嗜酸性粒细胞含量相关。研究还鉴定出主要影响增强子区域并调控关键免疫基因(如IL4、IL5RA和EPX)的整体低甲基化。这些DMR在n=267个样本中得到验证可能与异常基因表达有关。在已建立表型中鉴定的158个DMR中有56个在出生时就已经受到影响,至少部分与产前影响有关(如产前烟草烟雾暴露或邻苯二甲酸盐暴露)。
图形摘要
全血的全基因组重亚硫酸盐测序鉴定出158个与儿童哮喘相关的DMR,主要靶向增强子区域,包括IL-4的增强子。哮喘表型中的整体DNA低甲基化调控关键免疫基因,如IL5RA或EPX。哮喘中35%的异常DNA甲基化存在于脐带血中,一定程度上与吸烟等不良产前影响有关。
缩写:
DMR:differentially methylated region, 差异甲基化区域
WGBS:whole-genome bisulfite sequencing,全基因组重亚硫酸盐测序
EPX:eosinophil peroxidase,嗜酸性粒细胞过氧化酶
IL5RA:interleukin 5 receptor subunit alpha,白细胞介素5受体亚基α
LINA:Lifestyle and environmental factors and their influence on newborns allergy risk,生活方式和环境因素及其对新生儿过敏风险的影响
LISA:Lifestyle-related factors on the immune system and the development of allergies in childhood;,生活方式相关因素对免疫系统和儿童过敏发展的影响
PASTURE:Protection against allergy: study in rural environments,抗过敏:农村环境研究
研究结果:
(1)儿童哮喘的全基因组DNA低甲基化
图1:研究设计。对三个队列的哮喘儿童患者和对照组儿童的血样进行WGBS,以鉴定哮喘相关的DMR。通过两种独立的DMR calling算法DSS和metilene来分析比较哮喘儿童和对照儿童的DMR。随后对哮喘相关DMR进行了分析。1基于LINA和PASTURE的脐带血样本。2基于LINA和LISA的可用全血样本。3基于LINA的可用血浆样品。
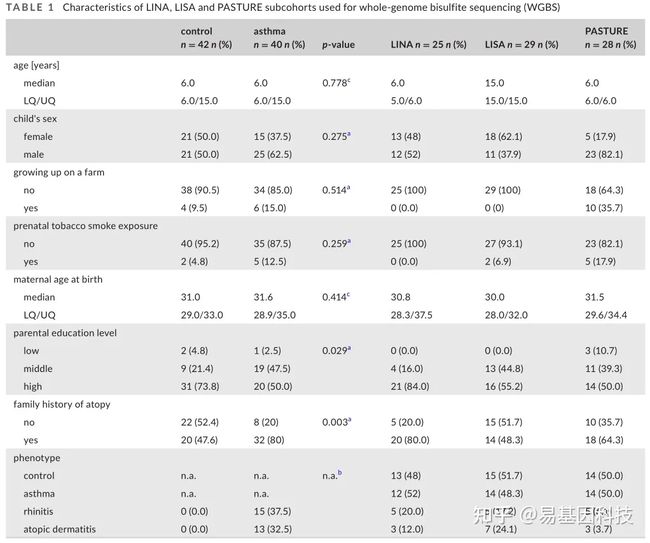
表1:全基因组亚硫酸氢盐测序 (WGBS) 的 LINA、LISA 和 PASTURE 亚队列特征
LQ=下四分位数,UQ=上四分位数。
a Fisher精确测试。
b对照组仅从非特应性儿童中选择。
cMann–Whitney U型测试。
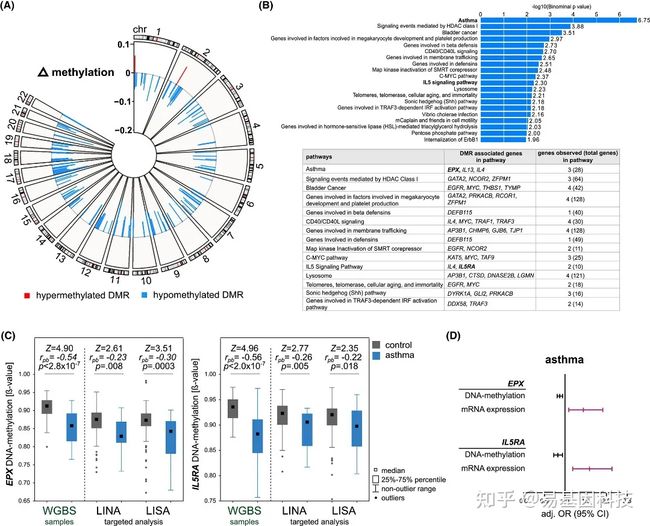
图2:DMR分布和下游分析。
(A) Circos图表示哮喘儿童与对照组中鉴定的158个差异甲基化区域(DMR)在所有常染色体中的分布。外圈显示22个常染色体。内圈条形图代表DMR及其染色体位置。高甲基化DMR用红色条表示,低甲基化DMRs用蓝色条表示。每个条的高度表示哮喘患者和对照组之间的DNA甲基化差异。
(B) 基于基因组位点的所有哮喘DMR的KEGG通路富集。
(C) 分别通过测序或MassARRAY检测哮喘儿童与WGBS样本(哮喘n=40,对照n=42)、LINA研究(哮喘n=19,对照n=108)和LISA研究(哮喘病n=25,对照n=115)中与EPX和IL5RA相关DMR的DNA甲基化差异(p值来自Mann–Whitney U-test,rpb:点双序列相关系数)。
(D) 结合LINA和LISA研究的meta分析中,EPX和IL5RA DNA甲基化(黑须)和转录(洋红色须)与哮喘结局相关性。(DNA甲基化:哮喘n=44,对照组n=223, EPX转录:哮喘n=44,对照组n = 220, IL5RA转录:哮喘n=44,对照组n=222)。考虑到儿童性别、队列、产前烟草烟雾暴露、特应性体质家族史、父母受教育程度和出生时母亲年龄等因素,采用ln-转化DNA甲基化值进行logistic回归校正后的OR值为+/-95%CI。
(2)遗传和细胞类型组成对哮喘相关DMR的影响
由于DNA甲基化水平可能强烈依赖于基因型或细胞类型组成,因此根据细胞类型依赖性和基因型相关性(gDMR)对哮喘相关DMR进行分类。根据这一分类,158个DMR中有38个(24.1%)与遗传背景有关,而其余120个DMR(75.9%)为非基因型相关DMR(ngDMR)。共鉴定出465个与38个gDMR相关的meQTL,其中没有一个在此前的全基因组关联研究中被描述为哮喘风险因子。包括GWAS目录中的所有表型性状,共有14个DMR至少与一个性状相关。对于其中8个DMR,该性状显示出与哮喘的松散表型关联,包括肺功能(rs645601和rs7700998)。五个SNPs与不同血细胞类型计数相关,其中RPN1-DMR上游的rs4328821和rs7646596与嗜酸性粒细胞计数相关。此外,与MAD1L1-DMR相关的rs12699415与特发性肺纤维化相关。
此外,研究表明哮喘儿童血液中嗜酸性粒细胞频率增加(Mann-Whitney U检验:Z=3.42,rpb=0.32,p=0.017),但其余细胞类型(B细胞、T细胞、单核细胞、NK细胞或中性粒细胞)没有增加。采用校正后的多元回归分析来检验不同的细胞类型频率是否对所确定的DMR的DNA甲基化水平有影响,结果显示37%的哮喘DMR(58个DMR)与嗜酸性粒细胞比例有关,只有三个DMR与B细胞、T细胞、单核细胞、NK细胞或中性粒细胞相关。然而,即使在校正后的多元回归模型中考虑了这些细胞类型后,哮喘仍然是所有细胞类型依赖性DMR DNA甲基化状态的重要影响因子。
(3)DNA甲基化变化模式与免疫调节异常相关
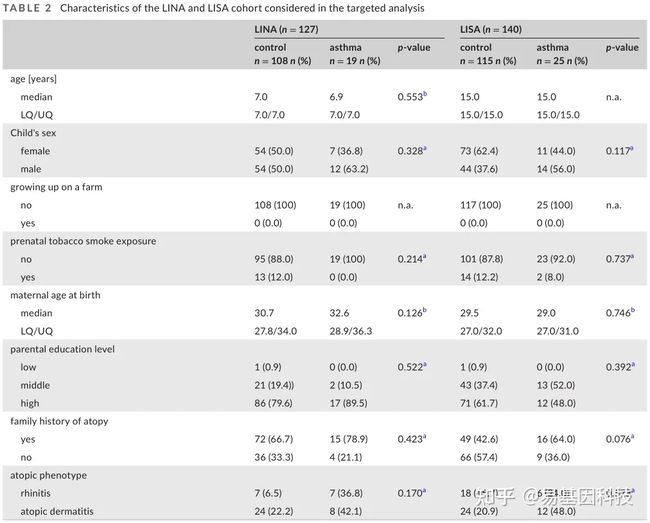
表2:目标分析中考虑的LINA和LISA队列的特征
(4)哮喘中DNA甲基化变化影响调控中枢
图3:哮喘相关DMR的基因组定位和IL4增强子低甲基化的功能翻译。
(A) 饼图表示受哮喘相关DMR影响的基因组区域比例分布。
(B) 在UCSC基因组浏览器中,IL4 DMR和MassARRAY分析的基因组位点和基因组区域。
(C) 散点图显示在LINA研究的6岁儿童中,IL4 DNA甲基化与IL4转录(n=112)和IL-4蛋白浓度(n=115)的关联,以及IL-4蛋白浓度与IL4逆转录(n=111)的关联。相关系数(ρ)和p值来自Spearman。(D) 6岁LINA儿童IL4增强子DNA甲基化、IL4转录和IL-4蛋白浓度关系的中介分析(n=111)。模型根据儿童性别、产前烟草烟雾暴露、特应性体质家族史、父母受教育程度和出生时的母亲年龄进行校正。在PHA刺激后检测IL-4蛋白浓度。蛋白质和DNA甲基化数据在分析前进行ln转换。间接路径效应大小以标准化β值表示(+/-95% CI)。通过偏差校正自举确定显著性。MA=MassARRAY,DMR=差异甲基化区域。
(5)受DNA甲基化变化调控基因在功能上相互关联
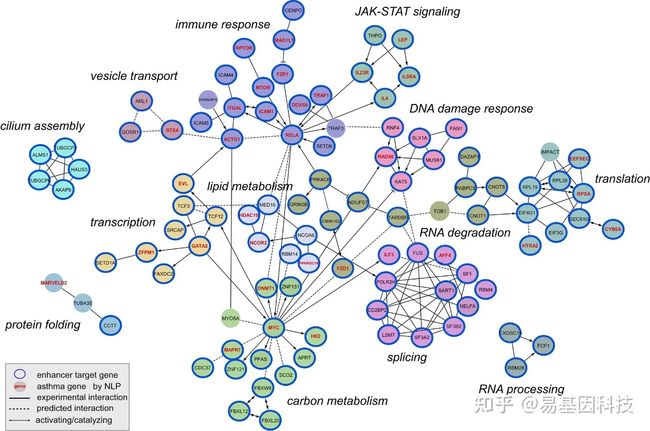
图4:哮喘DMR相关基因的网络模块分析。所有哮喘DMR相关基因显示出预测或基于实验的互作关系。仅显示具有多个连接的模块。受DMR调控的增强子靶基因由蓝色轮廓圆圈突出显示。由自然语言处理工具确定的哮喘相关基因或类似基因用红色字体表示。模块命名基于通路富集分析。
(6)哮喘的产前预激(Prenatal priming for asthma)
图5:脐带血哮喘DMR。
(A) 对匹配的脐带血样本进行WGBS测序,以确定对照组儿童与后来发展为哮喘的儿童相比,出生时哮喘相关DMR的DNA甲基化水平。
(B) 饼图表示部分基因型和非基因型相关DMR(g/ngDMR)以及那些已经在脐带血样本中差异甲基化的DMR(=脐带血DMR)。该表列出了之前与56个脐血哮喘相关DMR中包含的CPG相关的所有产前影响因素。红色字体突出显示通过自然语言处理鉴定的哮喘基因,以浅蓝色显示ngDMR,深蓝色表示gDMR。
增强子靶基因来源于GeneHancer,在没有GeneHancher注释的情况下,给出最接近的TSS基因。(C) 脐带血DMR相关基因的网络模块分析(左图)和仅存在于哮喘表型中的DMR相关基因网络模型分析(右图)。仅显示具有多个连接的模块。模块命名基于图4的网络。PFOS:perfluorooctane sulfonic acid,全氟辛烷磺酸,NLP:natural language processing,自然语言处理。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。
易基因科技提供全面的DNA甲基化研究整体解决方案。
参考文献:
Thürmann L, Klös M, Mackowiak SD, Bieg M, Bauer T, Ishaque N, Messingschlager M, Herrmann C, Röder S, Bauer M, Schäuble S, Faessler E, Hahn U, Weichenhan D, Mücke O, Plass C, Borte M, von Mutius E, Stangl GI, Lauener R, Karvonen AM, Divaret-Chauveau A, Riedler J, Heinrich J, Standl M, von Berg A, Schaaf B, Herberth G, Kabesch M, Eils R, Trump S, Lehmann I. Global hypomethylation in childhood asthma identified by genome-wide DNA-methylation sequencing preferentially affects enhancer regions. Allergy. 2023 Jun;78(6):1489-1506.
相关文献:
科研进展 | 全基因组DNA甲基化和转录组分析揭示绵羊羊毛类型变异的关键基因
项目文章 | WGBS等揭示SOX30甲基化在非梗阻性无精症中的表观遗传调控机制
项目文章|WGBS+RNA-seq揭示PM2.5引起男性生殖障碍的DNA甲基化调控机制
14种全基因组DNA甲基化测序(WGBS)标准分析比对软件的比较 | 生信专区