【生物信息学】使用谱聚类(Spectral Clustering)算法进行聚类分析
目录
一、实验介绍
二、实验环境
1. 配置虚拟环境
2. 库版本介绍
3. IDE
三、实验内容
0. 导入必要的工具
1. 生成测试数据
2. 绘制初始数据分布图
3. 循环尝试不同的参数组合并计算聚类效果
4. 输出最佳参数组合
5. 绘制最佳聚类结果图
6. 代码整合
一、实验介绍
本实验实现了使用谱聚类(Spectral Clustering)算法进行聚类分析
二、实验环境
本系列实验使用了PyTorch深度学习框架,相关操作如下(基于深度学习系列文章的环境):
1. 配置虚拟环境
深度学习系列文章的环境
conda create -n DL python=3.7 conda activate DLpip install torch==1.8.1+cu102 torchvision==0.9.1+cu102 torchaudio==0.8.1 -f https://download.pytorch.org/whl/torch_stable.html
conda install matplotlibconda install scikit-learn新增加
conda install pandasconda install seabornconda install networkxconda install statsmodelspip install pyHSICLasso注:本人的实验环境按照上述顺序安装各种库,若想尝试一起安装(天知道会不会出问题)
2. 库版本介绍
| 软件包 | 本实验版本 | 目前最新版 |
| matplotlib | 3.5.3 | 3.8.0 |
| numpy | 1.21.6 | 1.26.0 |
| python | 3.7.16 | |
| scikit-learn | 0.22.1 | 1.3.0 |
| torch | 1.8.1+cu102 | 2.0.1 |
| torchaudio | 0.8.1 | 2.0.2 |
| torchvision | 0.9.1+cu102 | 0.15.2 |
新增
| networkx | 2.6.3 | 3.1 |
| pandas | 1.2.3 | 2.1.1 |
| pyHSICLasso | 1.4.2 | 1.4.2 |
| seaborn | 0.12.2 | 0.13.0 |
| statsmodels | 0.13.5 | 0.14.0 |
3. IDE
建议使用Pycharm(其中,pyHSICLasso库在VScode出错,尚未找到解决办法……)
win11 安装 Anaconda(2022.10)+pycharm(2022.3/2023.1.4)+配置虚拟环境_QomolangmaH的博客-CSDN博客https://blog.csdn.net/m0_63834988/article/details/128693741https://blog.csdn.net/m0_63834988/article/details/128693741
三、实验内容
0. 导入必要的工具
import numpy as np
from sklearn.cluster import SpectralClustering
from sklearn.datasets import make_blobs
import matplotlib.pyplot as plt
from numpy import random
from sklearn import metrics1. 生成测试数据
random.seed(1)
x, y = make_blobs(n_samples=400, centers=4, cluster_std=1.5) 使用make_blobs方法生成了一个包含400个样本的数据集,共有4个聚类中心,每个聚类中心的标准偏差为1.5。
2. 绘制初始数据分布图
plt.scatter(x[:, 0], x[:, 1], c=y, label=len(np.unique(y)))
plt.title("Initial Data Distribution")
plt.show()将生成的数据集绘制成散点图,不同聚类的样本使用不同的颜色进行标记。
3. 循环尝试不同的参数组合并计算聚类效果
gamma_best = 0
k_cluster_best = 0
CH = 0
for index, gamma in enumerate((1, 1.5, 2, 2.5, 5)):
for index, k in enumerate((2, 3, 4, 5, 6)):
y_pred = SpectralClustering(n_clusters=k, gamma=gamma).fit_predict(x)
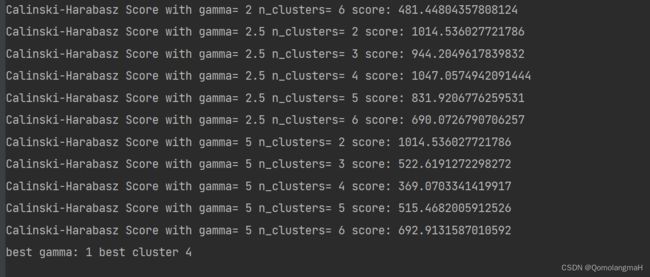
print("Calinski-Harabasz Score with gamma=", gamma, "n_clusters=", k, "score:",
metrics.calinski_harabasz_score(x, y_pred))
curr_CH = metrics.calinski_harabasz_score(x, y_pred)
if (curr_CH > CH):
gamma_best = gamma
k_cluster_best = k
CH = curr_CH- 使用嵌套的循环尝试不同的参数组合
- 其中
gamma代表谱聚类中的高斯核参数 k代表聚类的簇数。
- 其中
- 对于每一组参数,使用
SpectralClustering进行聚类,并计算聚类结果的 Calinski-Harabasz 得分(metrics.calinski_harabasz_score)。得分越高表示聚类效果越好。代码会记录得分最高的参数组合。
4. 输出最佳参数组合
print("best gamma:", gamma_best, "best cluster", k_cluster_best) 输出得分最高的参数组合(即最佳的 gamma 和 k)。
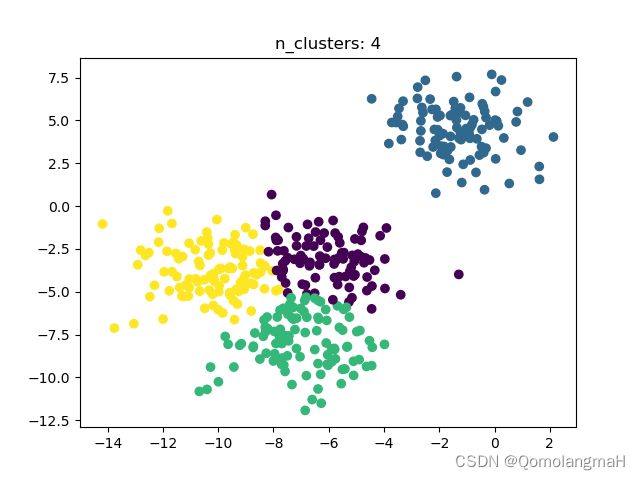
5. 绘制最佳聚类结 果图
f = plt.figure()
sc = SpectralClustering(n_clusters=k_cluster_best, gamma=gamma_best).fit_predict(x)
plt.scatter(x[:, 0], x[:, 1], c=sc)
plt.title("n_clusters: " + str(k_cluster_best))
plt.show()6. 代码整合
import numpy as np
from sklearn.cluster import SpectralClustering
from sklearn.datasets import make_blobs
import matplotlib.pyplot as plt
from numpy import random
from sklearn import metrics
SpectralClustering(affinity='rbf', coef0=1, degree=3, gamma=1.0,
kernel_params=None, n_clusters=4, n_init=10,
n_neighbors=10)
# scikit中的make_blobs方法常被用来生成聚类算法的测试数据,make_blobs会根据用户指定的特征数量、中心点数量、范围等来生成几类数据,这些数据可用于测试聚类算法的效果
random.seed(1)
# n_samples:样本数 n_features:int,可选(默认值= 2)centers:要生成的中心数或固定的中心位置 cluster_std: 聚类的标准偏差
x, y = make_blobs(n_samples=400, centers=4, cluster_std=1.5)
plt.scatter(x[:, 0], x[:, 1], c=y, label=len(np.unique(y)))
plt.title("Initial Data Distribution")
plt.show()
gamma_best = 0
k_cluster_best = 0
CH = 0
for index, gamma in enumerate((1, 1.5, 2, 2.5, 5)):
for index, k in enumerate((2, 3, 4, 5, 6)):
y_pred = SpectralClustering(n_clusters=k, gamma=gamma).fit_predict(x)
# 卡林斯基哈拉巴斯得分(Calinski Harabasz score),本质是簇间距离与簇内距离的比值,整体计算过程与方差计算方式类似,也称为方差比标准,
# 通过计算类内各点与类中心的距离平方和来度量类内的紧密度(类内距离),各个类中心点与数据集中心点距离平方和来度量数据集的分离度(类间距离),
# 较高的 Calinski Harabasz 分数意味着更好的聚类
print("Calinski-Harabasz Score with gamma=", gamma, "n_clusters=", k, "score:",
metrics.calinski_harabasz_score(x, y_pred))
curr_CH = metrics.calinski_harabasz_score(x, y_pred)
if (curr_CH > CH):
gamma_best = gamma
k_cluster_best = k
CH = curr_CH
print("best gamma:", gamma_best, "best cluster", k_cluster_best)
f = plt.figure()
sc = SpectralClustering(n_clusters=k_cluster_best, gamma=gamma_best).fit_predict(x)
plt.scatter(x[:, 0], x[:, 1], c=sc)
plt.title("n_clusters: " + str(k_cluster_best))
plt.show()
请详细介绍上述代码