医学图像分割模型:U-Net详解及实战
1 U-Net网络介绍
1.1 U-Net由来
2015年U-Net的出现使得原先需要数千个带注释的数据才能进行训练的深度学习神经网络大大减少了训练所需要的数据量,并且其针对神经网络在图像分割上的应用开创了先河。当时神经网络在图像分类任务上已经有了较好的成果,但在很多视觉的任务中由于输出需要进行定位,也就是每个像素需要分配一个类标签,这导致成千上万的训练图像在生物医学任务中通常难以获得,从而急需要一个神经网络,它不需要那么多的数据来进行训练却依旧有较好的效果,这就导致了U-Net的诞生。
U-Net几乎是当前segmentation项目中应用最广的模型。Unet能从更少的训练图像中进行学习,当它在少于40张图的生物医学数据集上训练时,IOU值仍能达到92%。
论文地址:https://arxiv.org/pdf/1505.04597.pdf
代码地址:https://github.com/milesial/Pytorch-UNet
1.2 U-Net网络
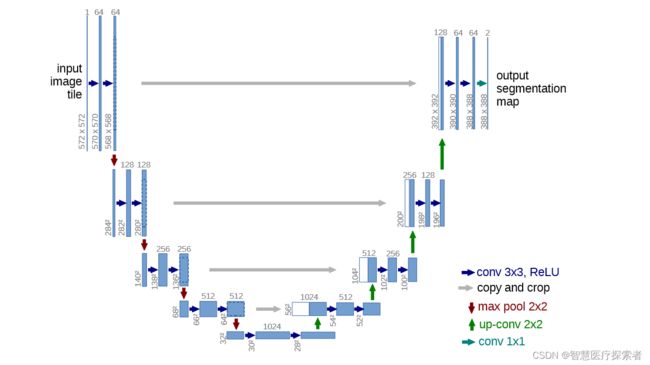
典型的encoder-decoder结构
-
左边是encoder,也就是提取特征和下采样的部分;右边decoder解码是一系列上采样,得到最终的一个分割图
-
图中每个长条的矩形对应的都是一个特征层,箭头都是一种操作
-
从输入开始看,输入是572x572的单通道的图片为例,首先进行一个卷积操作(步距为1,no padding)通过卷积层之后,高和宽都会减少,当时15年还没有BN。经过两个卷积层后进行下采样(max poooling),2x2,高和宽就会减半,channel不会变化,还是64。接着再进行两个3x3卷积层,(每次下采样后都会将channel进行翻倍,64-128)。右半部分:绿色的是上采样(其实是转置卷积,可回看视频,经过之后会将特征层的高和宽放大两倍,channel减半),1024-512,对应蓝色部分;灰色箭头是copy和crop,左边的高和宽是64x64,右边得到的是56x56大小的,无法进行直接拼接,所以对左边的特征层进行中心裁剪,和右边的蓝色部分进行拼接,拼接之后channel为1024。后面依次进行上采样。直到最后得到宽和高388x388,最后经过一个灰色的箭头卷积层,这个卷积核的个数和我们分类的类别个数是一致的,论文中只分割前景和背景。输出的结构就是388x388x2。(注意最后1x1的卷积的没有激活函数)
-
注意得到的分割图和原图并不一样,网络并不是完全对称,主要是因为用了 valid 卷积。除了收缩路径和扩张路径,中间还有一条拼接路径(skip connection),将左边的特征图(需要裁剪crop)拼接到右边。
-
反思:现在主流的实验方式并非严格按照论文中的方式去实现,而是在卷积层加上一个padding,即每次经过3x3卷积层,不会改变特征层的高和宽,并且会在卷积和ReLu之间加上一个BN(效果:在拼接的时候不要中心裁剪了,最后得到的高和宽和我们输入的高和宽保持一致)。
-
针对特别大(高分辨率的)图片,一般每次只分割一个patch,相邻两个预测区域之间一般会有一个重叠的部分,称为overlap,能够更好的分割边界区域。
-
pixel-wise loss的方案:细胞与细胞之间的背景取,赋予大的权重,对于大片的背景区域,施加小的权重
1.3 U-Net网络为何在医学图像分割表现好
UNet最早发表在2015的MICCAI上,短短3年,引用量目前已经达到了4070,足以见得其影响力。而后成为大多做医疗影像语义分割任务的baseline,也启发了大量研究者去思考U型语义分割网络。而如今在自然影像理解方面,也有越来越多的语义分割和目标检测SOTA模型开始关注和使用U型结构,比如语义分割Discriminative Feature Network(DFN)(CVPR2018),目标检测Feature Pyramid Networks for Object Detection(FPN)(CVPR 2017)等。
医疗影像的特点:
- 图像语义较为简单、结构较为固定。我们做脑的,就用脑CT和脑MRI,做胸片的只用胸片CT,做眼底的只用眼底OCT,都是一个固定的器官的成像,而不是全身的。由于器官本身结构固定和语义信息没有特别丰富,所以高级语义信息和低级特征都显得很重要(UNet的skip connection和U型结构就派上了用场)。
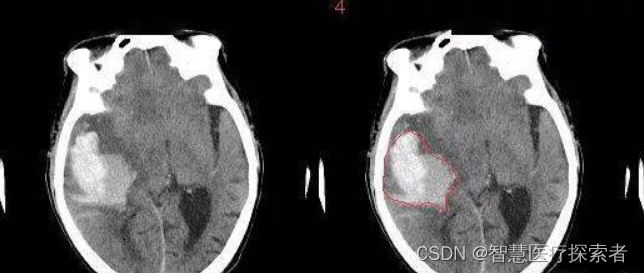
脑出血. 在CT影像上,高密度的区域就大概率是一块出血,如下图红色框区域。
眼底水肿。左图原图,右图标注(不同灰度值代表不同的水肿病变区域)。在OCT上,凸起或者凹陷的区域就大概率是一个水肿病变的区域。
- 数据量少。医学影像的数据获取相对难一些,很多比赛只提供不到100例数据。所以我们设计的模型不宜多大,参数过多,很容易导致过拟合。原始U-Net的参数量在28M左右(上采样带转置卷积的UNet参数量在31M左右),而如果把channel数成倍缩小,模型可以更小。缩小两倍后,UNet参数量在7.75M。缩小四倍,可以把模型参数量缩小至2M以内,非常轻量。个人尝试过使用Deeplab v3+和DRN等自然图像语义分割的SOTA网络在自己的项目上,发现效果和UNet差不多,但是参数量会大很多。
- 多模态。相比自然影像,医疗影像比较有趣和不同的一点是,医疗影像是具有多种模态的。以ISLES脑梗竞赛为例,其官方提供了CBF,MTT,CBV,TMAX,CTP等多种模态的数据。这就需要我们更好的设计网络去提取不同模态的特征feature。
- 可解释性重要。由于医疗影像最终是辅助医生的临床诊断,所以网络告诉医生一个3D的CT有没有病是远远不够的,医生还要进一步的想知道,病灶在哪一层,在哪一层的哪个位置,分割出来了吗,能求体积嘛?同时对于网络给出的分类和分割等结果,医生还想知道为什么,所以一些神经网络可解释性的trick就有用处了,比较常用的就是画activation map。看网络的哪些区域被激活了,如下图。
2 U-Net部署及实战
2.1 conda环境准备
conda环境准备详见:annoconda
2.2 运行环境构建
git clone https://github.com/milesial/Pytorch-UNet
cd Pytorch-UNet/
conda create -n unet python=3.8
conda activate unet
pip install torchvision==0.15.1
pip install -r requirements.txt
2.3 预训练模型下载
下载地址:https://github.com/milesial/Pytorch-UNet/releases/tag/v3.0
net = torch.hub.load('milesial/Pytorch-UNet', 'unet_carvana', pretrained=True, scale=0.5)2.4 训练数据下载
bash scripts/download_data.sh2.5 模型训练
python train.py --amp2.6 模型预测
python predict.py -i image.jpg -o output.jpg3 自己动手实现U-Net网络
import torch.nn as nn
import torch
# 编码器(论文中称之为收缩路径)的基本单元
def contracting_block(in_channels, out_channels):
block = torch.nn.Sequential(
# 这里的卷积操作没有使用padding,所以每次卷积后图像的尺寸都会减少2个像素大小
nn.Conv2d(kernel_size=(3, 3), in_channels=in_channels, out_channels=out_channels),
nn.BatchNorm2d(out_channels),
nn.ReLU(),
nn.Conv2d(kernel_size=(3, 3), in_channels=out_channels, out_channels=out_channels),
nn.BatchNorm2d(out_channels),
nn.ReLU()
)
return block
# 解码器(论文中称之为扩张路径)的基本单元
class expansive_block(nn.Module):
def __init__(self, in_channels, mid_channels, out_channels):
super(expansive_block, self).__init__()
# 每进行一次反卷积,通道数减半,尺寸扩大2倍
self.up = nn.ConvTranspose2d(in_channels, in_channels // 2, kernel_size=(3, 3), stride=2, padding=1,
output_padding=1)
self.block = nn.Sequential(
# 这里的卷积操作没有使用padding,所以每次卷积后图像的尺寸都会减少2个像素大小
nn.Conv2d(kernel_size=(3, 3), in_channels=in_channels, out_channels=mid_channels),
nn.BatchNorm2d(mid_channels),
nn.ReLU(),
nn.Conv2d(kernel_size=(3, 3), in_channels=mid_channels, out_channels=out_channels),
nn.BatchNorm2d(out_channels),
nn.ReLU()
)
def forward(self, e, d):
d = self.up(d)
# concat
# e是来自编码器部分的特征图,d是来自解码器部分的特征图,它们的形状都是[B,C,H,W]
diffY = e.size()[2] - d.size()[2]
diffX = e.size()[3] - d.size()[3]
# 裁剪时,先计算e与d在高和宽方向的差距diffY和diffX,然后对e高方向进行裁剪,具体方法是两边分别裁剪diffY的一半,
# 最后对e宽方向进行裁剪,具体方法是两边分别裁剪diffX的一半,
# 具体的裁剪过程见下图一
e = e[:, :, diffY // 2:e.size()[2] - diffY // 2, diffX // 2:e.size()[3] - diffX // 2]
cat = torch.cat([e, d], dim=1) # 在特征通道上进行拼接
out = self.block(cat)
return out
# 最后的输出卷积层
def final_block(in_channels, out_channels):
block = nn.Conv2d(kernel_size=(1, 1), in_channels=in_channels, out_channels=out_channels)
return block
class UNet(nn.Module):
def __init__(self, in_channel, out_channel):
super(UNet, self).__init__()
# 编码器 (Encode)
self.conv_encode1 = contracting_block(in_channels=in_channel, out_channels=64)
self.conv_pool1 = nn.MaxPool2d(kernel_size=2, stride=2)
self.conv_encode2 = contracting_block(in_channels=64, out_channels=128)
self.conv_pool2 = nn.MaxPool2d(kernel_size=2, stride=2)
self.conv_encode3 = contracting_block(in_channels=128, out_channels=256)
self.conv_pool3 = nn.MaxPool2d(kernel_size=2, stride=2)
self.conv_encode4 = contracting_block(in_channels=256, out_channels=512)
self.conv_pool4 = nn.MaxPool2d(kernel_size=2, stride=2)
# 编码器与解码器之间的过渡部分(Bottleneck)
self.bottleneck = nn.Sequential(
nn.Conv2d(kernel_size=(3, 3), in_channels=512, out_channels=1024),
nn.BatchNorm2d(1024),
nn.ReLU(),
nn.Conv2d(kernel_size=(3, 3), in_channels=1024, out_channels=1024),
nn.BatchNorm2d(1024),
nn.ReLU()
)
# 解码器(Decode)
self.conv_decode4 = expansive_block(1024, 512, 512)
self.conv_decode3 = expansive_block(512, 256, 256)
self.conv_decode2 = expansive_block(256, 128, 128)
self.conv_decode1 = expansive_block(128, 64, 64)
self.final_layer = final_block(64, out_channel)
def forward(self, x):
# Encode
encode_block1 = self.conv_encode1(x)
encode_pool1 = self.conv_pool1(encode_block1)
encode_block2 = self.conv_encode2(encode_pool1)
encode_pool2 = self.conv_pool2(encode_block2)
encode_block3 = self.conv_encode3(encode_pool2)
encode_pool3 = self.conv_pool3(encode_block3)
encode_block4 = self.conv_encode4(encode_pool3)
encode_pool4 = self.conv_pool4(encode_block4)
# Bottleneck
bottleneck = self.bottleneck(encode_pool4)
# Decode
decode_block4 = self.conv_decode4(encode_block4, bottleneck)
decode_block3 = self.conv_decode3(encode_block3, decode_block4)
decode_block2 = self.conv_decode2(encode_block2, decode_block3)
decode_block1 = self.conv_decode1(encode_block1, decode_block2)
final_layer = self.final_layer(decode_block1)
return final_layer
if __name__ == '__main__':
image = torch.rand((1, 3, 572, 572))
unet = UNet(in_channel=3, out_channel=2)
mask = unet(image)
print(mask.shape)
# 输出结果:
torch.Size([1, 2, 388, 388])