Clustering:Model-Based Algorithm
我们在前面学习过的Clustering算法模型有:
基于划分(Partitioning):K-Means及其扩展算法
基于层次(Hierarchical):Hierarchical Cluster算法
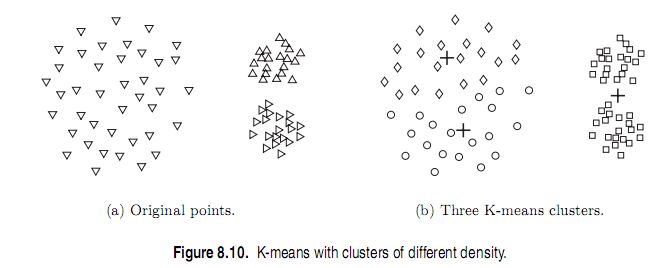
这两类算法能够在大多数常规数据空间中运行良好,但是其缺点也是比较明显。数据本身的特性,如欧式空间限制、初始值限制等,这些可以通过各种思路进行解决。现在有这么一些数据,其数据特性如下:
2:数据点在空间的分布形状不规则-非球形(Non-Globalar)
3:噪声影响
基于Partitioning、Hierarchical的Clustering算法在这些数据特征上表现不理想,如果认为通过增加簇的数目能解决这些问题的话请忽略这句话。对于这种情况,我们需要一种能够发现Density、Size、Outliar值的算法来解决这些问题,我们本次来学习基于空间数据Density来进行分簇的算法,这个实际上是基于Density-Model来进行分簇。
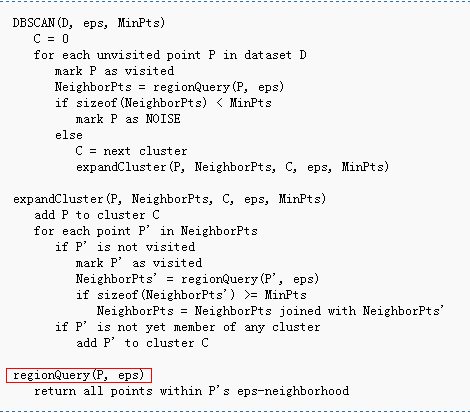
Density-Model Clustering的典型算法是DBSCAN(Density-Based Spatial Clustering of Application with Noise),由Martin Ester, Hans-Peter Kriegel, Jorg Sander, Xiaowei Xu等人在"A density-based algorithm for discovering clusters in large spatial databases with noise"中提出,算法的基本思路比较简单,采用Flood的方法来增加簇和更新簇内数据:
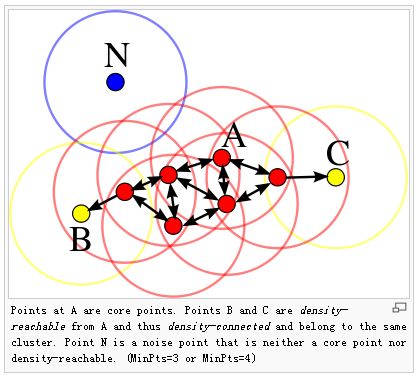
如果q离数据点P的距离小于给定的eps,就认为q是密度可达(Density-Reachable);如果数据点p周围有足够多(Sufficient)的数据点MinPts,就认为这些点应该归属于一个簇中。因此,归属于同一个簇的数据具有如下特性:
1:簇内所有点都是密度连通(Density-Connected)
2:如果一个数据点密度连通于簇中的任意一点,那么这个数据点一定在簇中。
从上面的描述来看,DBSCAN需要两个参数:eps和minPts。距离数据点eps距离之内的数据点个数满足至少minPts时,将这些数据点归属于同一个簇中。
算法执行流程的示意图可以参考这个:
复杂度分析:
从DBSCAN图中可以看到圈出来的地方,这个操作针对每个数据点都需要进行。通常情况下这个RangeQuery操作需要O(n)的时间复杂度,如果使用了索引结果(如K-D树)的话,可以将这个查找时间压缩到O(logn),所以最优的时间复杂度为O(n*logn)。为了减少重复计算,会将数据的Distance Matrix预先计算出来,这个需要O(n**2)的空间复杂度。
描述了DBSCAN的原理,可以看出来DBSCAN的最大优势是能够处理任意形状的簇,不用受到簇结构的影响;另外DBSCAN不需要像K-Means一样指定簇个数K,能够自动发现数据中的簇个数,这个也是个很大的好处;另外DBSCAN中有对于噪声能有识别,方便我们后期对这些噪声进行处理;另外对于数据集合内的数据顺序不敏感。最大的劣势是在RangeQuery中使用了距离的概念,那么对于纬度灾难问题一样没有办法;另外DBSCAN算法对于形状能够很好的解决,但是对于不同密度(Density),或者说密度差别太大的话,刚好是这个算法的软肋,没有办法通过不变的eps-minPts参数进行处理。
在github上有wiki上算法的流程实现:https://github.com/jessykate/DBScan/blob/master/dbscan.py,是python版本,这里还有个js版本的DBSCAN算法实现:https://github.com/bss/clustering.js/blob/master/clustering.js;我看了下还算是清楚。
wiki上有个Generalized DBSCAN(GDBSCAN)算法扩展,有兴趣的同学可以深入学习下。
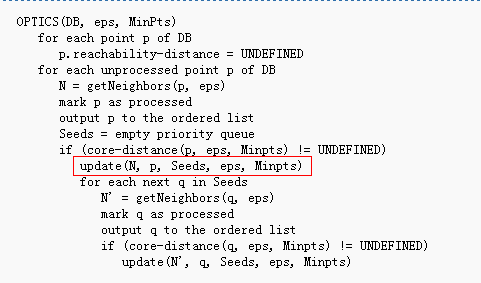
学习完DBSCAN算法,我们在上面看到了DBSCAN的缺点,初始化参数eps和MinPts需要用户手动输入,并且Clustering结果对这两个参数比较敏感,不同的取值会产生不同的结果;DBSCAN算法还不能很好处理不同密度(数据密度差距比较大)的数据集合,而OPTICS(Ordering Points to Identify the Clustering Structure)能解决这个问题。实际上OPTICS并不会产生结果类簇,而是为聚类分析生成一个增广的簇排序。这个排序代表了各样本点基于密度的聚类结果,如以可达距离为纵轴,样本点输出次序为横轴的坐标轴,包含的信息等价于从一个广泛的参数设置所获得的基于密度的聚类。换句话说,从这个排序中可以得到基于任何参数eps和MinPts的DBSCAN算法的聚类结果。
OPTICS引入两个额外的概念:
核心距离(Core Distance):对于数据点P的核心距离是指以P为核心对象的最小eps;如果P不是核心对象,这个核心距离没有意义。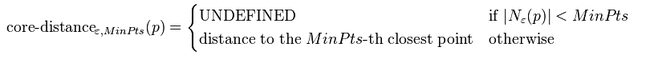
可达距离:数据点Q到数据点P的可达距离是指P的核心距离和P与Q之间的欧式距离之间的较大值;如果P不是核心对象,P和Q之间的可达距离没有什么意义。
这个流程非常重要,一定要仔细梳理这个流程,将过程仔细搞清楚。
这张OPTICS的截图来自这里:http://osl.iu.edu/~chemuell/projects/presentations/optics-v1.pdf 。从上图中也可以看出来,OPTICS的处理结果并不是单纯的Clustering,而是将数据点集合之间的距离关系很好的描述出来;而通过这些直观的距离关系又能够做许多额外的工作。
由于OPTICS能够得到数据的簇排序及数据集合之间的距离关系,那么基于OPTICS算法扩展就有很多,如OPTICS-OF来检测异常值(Outliar Detection);DeLi-Clu算法来消除eps参数,提供比OPTICS更好的性能指标;HiSC算法将其应用到Subspace Clustering;HiCO应用到相关聚类(Correlation Clustering)上;基于HiSC改进的DiSH能够找到更加复杂的簇结构。这些基于OPTICS的算法足以说明该算法的输出信息的多样性。ELKI框架实现了这些算法,对这些算法感兴趣的同学可以参考该框架对这些算法的实现,交叉学习验证。
OPTICS的算法实现可以参考这个JS实现:https://github.com/blauharley/OPTICS-Clustering-JavaScript,还是比较容易理解的。
本来还想写下Gaussian Mixture Model(GMM),发现文章已经够长了,这个GMM就等下次吧。