用DPM(Deformable Part Model,voc-release3.1)算法在INRIA数据集上训练自己的人体检测模型
我的环境
DPM源码版本:voc-release3.1
VOC开发包版本:VOC2007_devkit_08-Jun
Matlab版本:MatlabR2012b
c++编译器:VS2010
系统:Win7 32位
learn.exe迭代次数:5万次
数据集:INRIA 人体数据集,等
步骤一,首先要使voc-release3.1目标检测部分的代码在windows系统下跑起来:
在Windows下运行Felzenszwalb的Deformable Part Models(voc-release4.01)目标检测matlab源码
上文中用的4.01,3.1需要修改的地方是一样的,反而更简单。
步骤二,把训练部分代码跑通,在VOC数据集上进行测试,如下文:
在windows下运行Felzenszwalb的Deformable Part Model(DPM)源码voc-release3.1来训练自己的模型
步骤三,再之后就是使之能在其他的数据集上训练模型,比如INRIA人体数据集。
这一步中主要是修改pascal_data.m文件,这个文件的作用就是读取标注,为训练准备数据。此函数会返回两个数组,pos[]和neg[],
pos[]中是正样本信息,格式为:[imagePath x1 y1 x2 y2 ];
neg[]中是负样本信息,格式为:[imagePath] 。
先读取INRIA数据集的标注,保存为下面的格式:
然后在pascal_data.m中读取此文件,依次将标注信息保存到pos[]数组中,注意要将图片路径补全为绝对路径。
pos = []; % 存储正样本目标信息的数组,每个元素是一个结构,{im, x1, y1, x2, y2}
numpos = 0; % 正样本目标个数(一个图片中可能含有多个正样本目标)
% InriaPersonPos.txt是从Inria人体数据集获得的50个正样本的标注文件,格式为[x1 y1 x2 y2 RelativePath]
[a,b,c,d,p] = textread('InriaPersonPos.txt','%d %d %d %d %s'); % 注意:读取后p的类型时50*1的cell类型
% 遍历训练图片文件名数组ids
for i = 1:length(a);
if mod(i,10)==0
fprintf('%s: parsing positives: %d/%d\n', cls, i, length(a));
end;
numpos = numpos+1; % 正样本目标个数
pos(numpos).im = [VOCopts.datadir p{numpos}]; % 引用cell单元时要用{},引用矩阵单元时用()
pos(numpos).x1 = a(numpos);
pos(numpos).y1 = b(numpos);
pos(numpos).x2 = c(numpos);
pos(numpos).y2 = d(numpos);
endpos(numpos).im 中我也在相对路径前加了VOCopts的数据集目录datadir是因为我将INRIA数据集放在VOCdevkit目录下了。
这里要特别注意的是,不需要提前从INRIA数据集中根据标注文件手动裁出人体目标,而是将标注信息和正样本原图都告诉DPM算法,它自动会进行缩放、剪裁处理,对于有的标注信息超过图像边界的,也没关系,DPM中也会自己处理。
至于负样本就无所谓了,反正都是从不含人体的原图上随机裁取,还用VOC数据集中的就行。
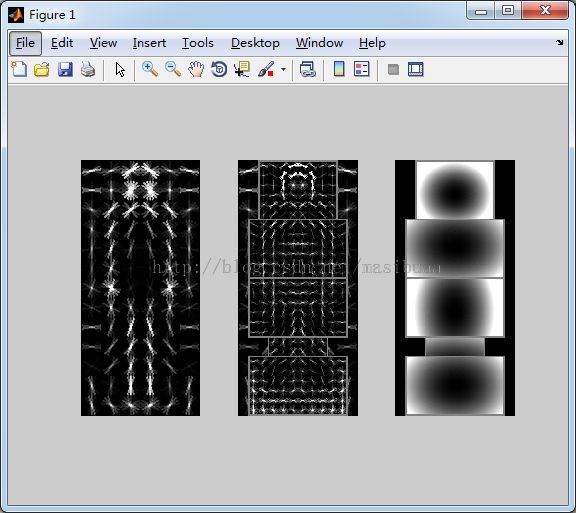
下面展示几个训练的模型,以及检测结果
(1)50个INRIA正样本目标,300个VOC负样本目标,单组件(component)模型,部件个数为6。
模型可视化图如下:
没想到仅用50个正样本,训练出的模型竟然很不错,这也跟INRIA人体数据集的质量很高有关。
检测结果如下:
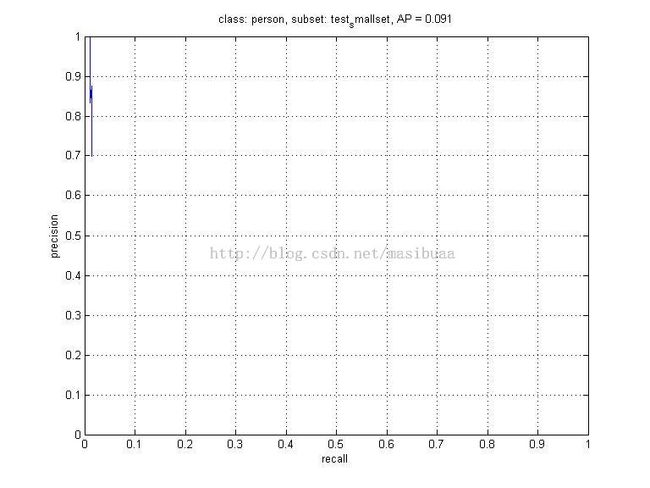
在500个VOC测试图上获得的平均精度AP=0.091
(2)537个Spinello RGBD数据集中的正样本目标,300个VOC负样本目标,单组件,6个部件。
模型可视化如下:
由于这537个正样本目标来自对单个人的跟踪结果,所以样本不太好,如下:
所以训练出来的模型根本检测不到任何人体目标。
(3)2396个Spinello RGBD数据集中的正样本目标,300个VOC负样本目标,单组件,6个部件。
模型可视化如下:
这次的数据源和(2)中相同,只不过这次正样本取自数据集中的所有34个人的跟踪结果,训练了一个晚上,结果还行。
检测结果如下:
在500个VOC测试图上获得的平均精度AP=0.091。带包围盒预测的精度-召回率(precision-recall)曲线如下: