基于遗传算法求解TSP问题(JAVA)
一、TSP问题
TSP问题(Travelling Salesman Problem)即旅行商问题,又译为旅行推销员问题、货郎担问题,是数学领域中著名问题之一。假设有一个旅行商人要拜访n个城市,他必须选择所要走的路径,路径的限制是每个城市只能拜访一次,而且最后要回到原来出发的城市。路径的选择目标是要求得的路径路程为所有路径之中的最小值。
TSP问题是一个组合优化问题。该问题可以被证明具有NPC计算复杂性。TSP问题可以分为两类,一类是对称TSP问题(Symmetric TSP),另一类是非对称问题(Asymmetric TSP)。所有的TSP问题都可以用一个图(Graph)来描述:
V={c1, c2, …, ci, …, cn},i = 1,2, …, n,是所有城市的集合.ci表示第i个城市,n为城市的数目;
E={(r, s): r,s∈ V}是所有城市之间连接的集合;
C = {crs: r,s∈ V}是所有城市之间连接的成本度量(一般为城市之间的距离);
如果crs = csr, 那么该TSP问题为对称的,否则为非对称的。
一个TSP问题可以表达为:
求解遍历图G = (V, E, C),所有的节点一次并且回到起始节点,使得连接这些节点的路径成本最低。
二、遗传算法
遗传算法(Genetic Algorithms )是基于生物进化理论的原理发展起来的一种广为应用的、高效的随机搜索与优化的方法。其主要特点是群体搜索策略和群体中个体之间的信息交换,搜索不依赖于梯度信息。它是在70年代初期由美国密西根( Michigan )大学的霍兰( Holland )教授发展起来的。1975年霍兰教授发表了第一本比较系统论述遗传算法的专著《自然系统与人工系统中的适应性》(《 Adaptationin Natural and Artificial Systems 》)。遗传算法最初被研究的出发点不是为专门解决最优化问题而设计的,它与进化策略、进化规划共同构成了进化算法的主要框架,都是为当时人工智能的发展服务的。迄今为止,遗传算法是进化算法中最广为人知的算法。
遗传火算法的实施步骤如下(以目标函数求最小为例)。
第一步:初始化 t←0进化代数计数器;T是最大进化代数;随机生成M个个体作为初始群体P(t);
第二步:个体评价 计算P(t)中各个个体的适应度;
第三步:选择运算 将选择算子作用于群体;
第四步:交叉运算 将交叉算子作用于群体;
第五步:变异运算 将变异算子作用于群体,并通过以上运算得到下一代群体P(t + 1);
第六步:终止条件判断 t≦T:t← t+1 转到步骤2;t>T:终止 输出解。
遗传算法应用步骤:
1)确定决策变量及各种约束条件,即个体的表现型X和问题的解空间;
2)建立优化模型 (目标函数最大OR 最小) 数学描述形式 量化方法;
3)染色体编码方法;
4)解码方法;
5)个体适应度的量化评价方法 F(x)
6)设计遗传算子;
7)确定有关运行参数。
三、遗传算法求解TSP问题
其实之前不大想写这个遗传算法求TSP,因为我之前已经有遗传算法求01背包了,但有些读者建议,加上确实tsp与01背包差别还是很大的,就再写一个。对于TSP之类的问题NP问题,使用启发式搜索算法是一个明智的选择,因为精确算发已经力不从心了。
使用遗传算法第一件事情就是确定染色编码方式,它根据不同的问题模型使用不同编码方式,有二进制编码也有整数编码和浮点数编码,面对TSP问题,我肯定选用整数编码,因为很简单,对于每个城市用一个整数来编号,例如有48个城市,就用0到47来标识每一个城市,然后一个路径就是一条染色体编码,染色体长度为48,如:0,1,2,3,4...47就是一个染色体,它表达的意思就是旅行者从0号城市出发,依次访问1,2,...47号城市再回到0号城市;遗传算法的第二个要点就是评价函数,TSP的评价函数很简单,就是染色体编码表达的路径总长度;最后很简单,其实在这个模型中就是将0到47这48个数进行全排列,从中找出最短的一条路径(想想48个数全排列,然后比较。。。)。清楚了解了这些,咱们就可以按照上面的遗传算法框架来进行编程了。
我们使用TSP问题依然来自于tsplib上的数据att48,这是一个对称TSP问题,城市规模为48,其最优值为10628.其距离计算方法下图所示:
具体代码如下:
package noah;
import java.io.BufferedReader;
import java.io.FileInputStream;
import java.io.IOException;
import java.io.InputStreamReader;
import java.util.Random;
public class GA {
private int scale;// 种群规模
private int cityNum; // 城市数量,染色体长度
private int MAX_GEN; // 运行代数
private int[][] distance; // 距离矩阵
private int bestT;// 最佳出现代数
private int bestLength; // 最佳长度
private int[] bestTour; // 最佳路径
// 初始种群,父代种群,行数表示种群规模,一行代表一个个体,即染色体,列表示染色体基因片段
private int[][] oldPopulation;
private int[][] newPopulation;// 新的种群,子代种群
private int[] fitness;// 种群适应度,表示种群中各个个体的适应度
private float[] Pi;// 种群中各个个体的累计概率
private float Pc;// 交叉概率
private float Pm;// 变异概率
private int t;// 当前代数
private Random random;
public GA() {
}
/**
* constructor of GA
*
* @param s
* 种群规模
* @param n
* 城市数量
* @param g
* 运行代数
* @param c
* 交叉率
* @param m
* 变异率
*
**/
public GA(int s, int n, int g, float c, float m) {
scale = s;
cityNum = n;
MAX_GEN = g;
Pc = c;
Pm = m;
}
// 给编译器一条指令,告诉它对被批注的代码元素内部的某些警告保持静默
@SuppressWarnings("resource")
/**
* 初始化GA算法类
* @param filename 数据文件名,该文件存储所有城市节点坐标数据
* @throws IOException
*/
private void init(String filename) throws IOException {
// 读取数据
int[] x;
int[] y;
String strbuff;
BufferedReader data = new BufferedReader(new InputStreamReader(
new FileInputStream(filename)));
distance = new int[cityNum][cityNum];
x = new int[cityNum];
y = new int[cityNum];
for (int i = 0; i < cityNum; i++) {
// 读取一行数据,数据格式1 6734 1453
strbuff = data.readLine();
// 字符分割
String[] strcol = strbuff.split(" ");
x[i] = Integer.valueOf(strcol[1]);// x坐标
y[i] = Integer.valueOf(strcol[2]);// y坐标
}
// 计算距离矩阵
// ,针对具体问题,距离计算方法也不一样,此处用的是att48作为案例,它有48个城市,距离计算方法为伪欧氏距离,最优值为10628
for (int i = 0; i < cityNum - 1; i++) {
distance[i][i] = 0; // 对角线为0
for (int j = i + 1; j < cityNum; j++) {
double rij = Math
.sqrt(((x[i] - x[j]) * (x[i] - x[j]) + (y[i] - y[j])
* (y[i] - y[j])) / 10.0);
// 四舍五入,取整
int tij = (int) Math.round(rij);
if (tij < rij) {
distance[i][j] = tij + 1;
distance[j][i] = distance[i][j];
} else {
distance[i][j] = tij;
distance[j][i] = distance[i][j];
}
}
}
distance[cityNum - 1][cityNum - 1] = 0;
bestLength = Integer.MAX_VALUE;
bestTour = new int[cityNum + 1];
bestT = 0;
t = 0;
newPopulation = new int[scale][cityNum];
oldPopulation = new int[scale][cityNum];
fitness = new int[scale];
Pi = new float[scale];
random = new Random(System.currentTimeMillis());
/*
* for(int i=0;i<cityNum;i++) { for(int j=0;j<cityNum;j++) {
* System.out.print(distance[i][j]+","); } System.out.println(); }
*/
// 初始化种群
}
// 初始化种群
void initGroup() {
int i, j, k;
// Random random = new Random(System.currentTimeMillis());
for (k = 0; k < scale; k++)// 种群数
{
oldPopulation[k][0] = random.nextInt(65535) % cityNum;
for (i = 1; i < cityNum;)// 染色体长度
{
oldPopulation[k][i] = random.nextInt(65535) % cityNum;
for (j = 0; j < i; j++) {
if (oldPopulation[k][i] == oldPopulation[k][j]) {
break;
}
}
if (j == i) {
i++;
}
}
}
/*
* for(i=0;i<scale;i++) { for(j=0;j<cityNum;j++) {
* System.out.print(oldPopulation[i][j]+","); } System.out.println(); }
*/
}
public int evaluate(int[] chromosome) {
// 0123
int len = 0;
// 染色体,起始城市,城市1,城市2...城市n
for (int i = 1; i < cityNum; i++) {
len += distance[chromosome[i - 1]][chromosome[i]];
}
// 城市n,起始城市
len += distance[chromosome[cityNum - 1]][chromosome[0]];
return len;
}
// 计算种群中各个个体的累积概率,前提是已经计算出各个个体的适应度fitness[max],作为赌轮选择策略一部分,Pi[max]
void countRate() {
int k;
double sumFitness = 0;// 适应度总和
double[] tempf = new double[scale];
for (k = 0; k < scale; k++) {
tempf[k] = 10.0 / fitness[k];
sumFitness += tempf[k];
}
Pi[0] = (float) (tempf[0] / sumFitness);
for (k = 1; k < scale; k++) {
Pi[k] = (float) (tempf[k] / sumFitness + Pi[k - 1]);
}
/*
* for(k=0;k<scale;k++) { System.out.println(fitness[k]+" "+Pi[k]); }
*/
}
// 挑选某代种群中适应度最高的个体,直接复制到子代中
// 前提是已经计算出各个个体的适应度Fitness[max]
public void selectBestGh() {
int k, i, maxid;
int maxevaluation;
maxid = 0;
maxevaluation = fitness[0];
for (k = 1; k < scale; k++) {
if (maxevaluation > fitness[k]) {
maxevaluation = fitness[k];
maxid = k;
}
}
if (bestLength > maxevaluation) {
bestLength = maxevaluation;
bestT = t;// 最好的染色体出现的代数;
for (i = 0; i < cityNum; i++) {
bestTour[i] = oldPopulation[maxid][i];
}
}
// System.out.println("代数 " + t + " " + maxevaluation);
// 复制染色体,k表示新染色体在种群中的位置,kk表示旧的染色体在种群中的位置
copyGh(0, maxid);// 将当代种群中适应度最高的染色体k复制到新种群中,排在第一位0
}
// 复制染色体,k表示新染色体在种群中的位置,kk表示旧的染色体在种群中的位置
public void copyGh(int k, int kk) {
int i;
for (i = 0; i < cityNum; i++) {
newPopulation[k][i] = oldPopulation[kk][i];
}
}
// 赌轮选择策略挑选
public void select() {
int k, i, selectId;
float ran1;
// Random random = new Random(System.currentTimeMillis());
for (k = 1; k < scale; k++) {
ran1 = (float) (random.nextInt(65535) % 1000 / 1000.0);
// System.out.println("概率"+ran1);
// 产生方式
for (i = 0; i < scale; i++) {
if (ran1 <= Pi[i]) {
break;
}
}
selectId = i;
// System.out.println("选中" + selectId);
copyGh(k, selectId);
}
}
//进化函数,正常交叉变异
public void evolution() {
int k;
// 挑选某代种群中适应度最高的个体
selectBestGh();
// 赌轮选择策略挑选scale-1个下一代个体
select();
// Random random = new Random(System.currentTimeMillis());
float r;
// 交叉方法
for (k = 0; k < scale; k = k + 2) {
r = random.nextFloat();// /产生概率
// System.out.println("交叉率..." + r);
if (r < Pc) {
// System.out.println(k + "与" + k + 1 + "进行交叉...");
//OXCross(k, k + 1);// 进行交叉
OXCross1(k, k + 1);
} else {
r = random.nextFloat();// /产生概率
// System.out.println("变异率1..." + r);
// 变异
if (r < Pm) {
// System.out.println(k + "变异...");
OnCVariation(k);
}
r = random.nextFloat();// /产生概率
// System.out.println("变异率2..." + r);
// 变异
if (r < Pm) {
// System.out.println(k + 1 + "变异...");
OnCVariation(k + 1);
}
}
}
}
//进化函数,保留最好染色体不进行交叉变异
public void evolution1() {
int k;
// 挑选某代种群中适应度最高的个体
selectBestGh();
// 赌轮选择策略挑选scale-1个下一代个体
select();
// Random random = new Random(System.currentTimeMillis());
float r;
for (k = 1; k + 1 < scale / 2; k = k + 2) {
r = random.nextFloat();// /产生概率
if (r < Pc) {
OXCross1(k, k + 1);// 进行交叉
//OXCross(k,k+1);//进行交叉
} else {
r = random.nextFloat();// /产生概率
// 变异
if (r < Pm) {
OnCVariation(k);
}
r = random.nextFloat();// /产生概率
// 变异
if (r < Pm) {
OnCVariation(k + 1);
}
}
}
if (k == scale / 2 - 1)// 剩最后一个染色体没有交叉L-1
{
r = random.nextFloat();// /产生概率
if (r < Pm) {
OnCVariation(k);
}
}
}
// 类OX交叉算子
void OXCross(int k1, int k2) {
int i, j, k, flag;
int ran1, ran2, temp;
int[] Gh1 = new int[cityNum];
int[] Gh2 = new int[cityNum];
// Random random = new Random(System.currentTimeMillis());
ran1 = random.nextInt(65535) % cityNum;
ran2 = random.nextInt(65535) % cityNum;
// System.out.println();
// System.out.println("-----------------------");
// System.out.println("----"+ran1+"----"+ran2);
while (ran1 == ran2) {
ran2 = random.nextInt(65535) % cityNum;
}
if (ran1 > ran2)// 确保ran1<ran2
{
temp = ran1;
ran1 = ran2;
ran2 = temp;
}
// System.out.println();
// System.out.println("-----------------------");
// System.out.println("----"+ran1+"----"+ran2);
// System.out.println("-----------------------");
// System.out.println();
flag = ran2 - ran1 + 1;// 删除重复基因前染色体长度
for (i = 0, j = ran1; i < flag; i++, j++) {
Gh1[i] = newPopulation[k2][j];
Gh2[i] = newPopulation[k1][j];
}
// 已近赋值i=ran2-ran1个基因
for (k = 0, j = flag; j < cityNum;)// 染色体长度
{
Gh1[j] = newPopulation[k1][k++];
for (i = 0; i < flag; i++) {
if (Gh1[i] == Gh1[j]) {
break;
}
}
if (i == flag) {
j++;
}
}
for (k = 0, j = flag; j < cityNum;)// 染色体长度
{
Gh2[j] = newPopulation[k2][k++];
for (i = 0; i < flag; i++) {
if (Gh2[i] == Gh2[j]) {
break;
}
}
if (i == flag) {
j++;
}
}
for (i = 0; i < cityNum; i++) {
newPopulation[k1][i] = Gh1[i];// 交叉完毕放回种群
newPopulation[k2][i] = Gh2[i];// 交叉完毕放回种群
}
// System.out.println("进行交叉--------------------------");
// System.out.println(k1+"交叉后...");
// for (i = 0; i < cityNum; i++) {
// System.out.print(newPopulation[k1][i] + "-");
// }
// System.out.println();
// System.out.println(k2+"交叉后...");
// for (i = 0; i < cityNum; i++) {
// System.out.print(newPopulation[k2][i] + "-");
// }
// System.out.println();
// System.out.println("交叉完毕--------------------------");
}
// 交叉算子,相同染色体交叉产生不同子代染色体
public void OXCross1(int k1, int k2) {
int i, j, k, flag;
int ran1, ran2, temp;
int[] Gh1 = new int[cityNum];
int[] Gh2 = new int[cityNum];
// Random random = new Random(System.currentTimeMillis());
ran1 = random.nextInt(65535) % cityNum;
ran2 = random.nextInt(65535) % cityNum;
while (ran1 == ran2) {
ran2 = random.nextInt(65535) % cityNum;
}
if (ran1 > ran2)// 确保ran1<ran2
{
temp = ran1;
ran1 = ran2;
ran2 = temp;
}
// 将染色体1中的第三部分移到染色体2的首部
for (i = 0, j = ran2; j < cityNum; i++, j++) {
Gh2[i] = newPopulation[k1][j];
}
flag = i;// 染色体2原基因开始位置
for (k = 0, j = flag; j < cityNum;)// 染色体长度
{
Gh2[j] = newPopulation[k2][k++];
for (i = 0; i < flag; i++) {
if (Gh2[i] == Gh2[j]) {
break;
}
}
if (i == flag) {
j++;
}
}
flag = ran1;
for (k = 0, j = 0; k < cityNum;)// 染色体长度
{
Gh1[j] = newPopulation[k1][k++];
for (i = 0; i < flag; i++) {
if (newPopulation[k2][i] == Gh1[j]) {
break;
}
}
if (i == flag) {
j++;
}
}
flag = cityNum - ran1;
for (i = 0, j = flag; j < cityNum; j++, i++) {
Gh1[j] = newPopulation[k2][i];
}
for (i = 0; i < cityNum; i++) {
newPopulation[k1][i] = Gh1[i];// 交叉完毕放回种群
newPopulation[k2][i] = Gh2[i];// 交叉完毕放回种群
}
}
// 多次对换变异算子
public void OnCVariation(int k) {
int ran1, ran2, temp;
int count;// 对换次数
// Random random = new Random(System.currentTimeMillis());
count = random.nextInt(65535) % cityNum;
for (int i = 0; i < count; i++) {
ran1 = random.nextInt(65535) % cityNum;
ran2 = random.nextInt(65535) % cityNum;
while (ran1 == ran2) {
ran2 = random.nextInt(65535) % cityNum;
}
temp = newPopulation[k][ran1];
newPopulation[k][ran1] = newPopulation[k][ran2];
newPopulation[k][ran2] = temp;
}
/*
* for(i=0;i<L;i++) { printf("%d ",newGroup[k][i]); } printf("\n");
*/
}
public void solve() {
int i;
int k;
// 初始化种群
initGroup();
// 计算初始化种群适应度,Fitness[max]
for (k = 0; k < scale; k++) {
fitness[k] = evaluate(oldPopulation[k]);
// System.out.println(fitness[k]);
}
// 计算初始化种群中各个个体的累积概率,Pi[max]
countRate();
System.out.println("初始种群...");
for (k = 0; k < scale; k++) {
for (i = 0; i < cityNum; i++) {
System.out.print(oldPopulation[k][i] + ",");
}
System.out.println();
System.out.println("----" + fitness[k] + " " + Pi[k]);
}
for (t = 0; t < MAX_GEN; t++) {
//evolution1();
evolution();
// 将新种群newGroup复制到旧种群oldGroup中,准备下一代进化
for (k = 0; k < scale; k++) {
for (i = 0; i < cityNum; i++) {
oldPopulation[k][i] = newPopulation[k][i];
}
}
// 计算种群适应度
for (k = 0; k < scale; k++) {
fitness[k] = evaluate(oldPopulation[k]);
}
// 计算种群中各个个体的累积概率
countRate();
}
System.out.println("最后种群...");
for (k = 0; k < scale; k++) {
for (i = 0; i < cityNum; i++) {
System.out.print(oldPopulation[k][i] + ",");
}
System.out.println();
System.out.println("---" + fitness[k] + " " + Pi[k]);
}
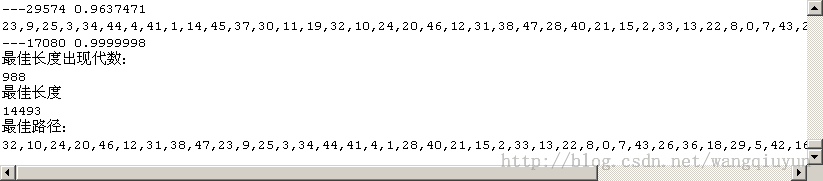
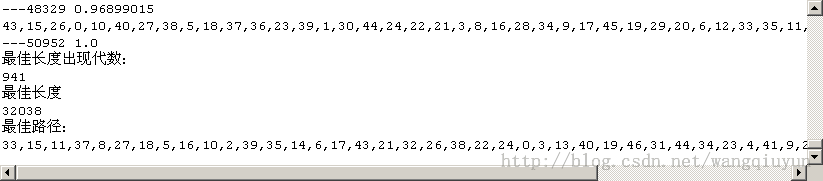
System.out.println("最佳长度出现代数:");
System.out.println(bestT);
System.out.println("最佳长度");
System.out.println(bestLength);
System.out.println("最佳路径:");
for (i = 0; i < cityNum; i++) {
System.out.print(bestTour[i] + ",");
}
}
/**
* @param args
* @throws IOException
*/
public static void main(String[] args) throws IOException {
System.out.println("Start....");
GA ga = new GA(30, 48, 1000, 0.8f, 0.9f);
ga.init("c://data.txt");
ga.solve();
}
}
运行结果截图:
四、总结
看到上面两个结果差别还是相当大的,其实我代码里提供了两个进化函数,两种交叉算子,需要提醒的是上面比较好的结果其实是使用了evolution1()这个进化函数,较差的是使用了evolution()这个函数,他们的最大区别就是前者保留最好染色体不进行交叉变异,后者则是按照基本遗传算法框架的完全交叉变异操作的,其区别之大各位可以深入琢磨,两种交叉算子的区别是一个就是简单的类OX交叉算子,另外一个交叉算子改进了一下,相同染色体交叉产生不同子代染色体,实际上它两对结果影响并不是很大,以上是我的一点点简单的总结,还是那句话,或许你觉得遗传算法效率其实也不怎样,实际上基本的遗传算法是有很多不足的,如容易选入局部收敛,全局搜索能力不够强,但是基本的遗传算法是有很多可以改进的地方,如交叉算子的设计、变异算子的设计、选择策略等等,有关遗传算法个人觉得作为一种智能启发式搜索算法它甚至比别的普通算法(如动态规划)理解起来还容易,而且它特别容易与别的算法相结合,设计新的混合算法,另外在基本遗传算法的基础上还衍生出了很多别的算法,如免疫遗传算法,这些都是一些比较高级的算法。