简单易学的机器学习算法——Metropolis-Hastings算法
在简单易学的机器学习算法——马尔可夫链蒙特卡罗方法MCMC中简单介绍了马尔可夫链蒙特卡罗MCMC方法的基本原理,介绍了Metropolis采样算法的基本过程,这一部分,主要介绍Metropolis-Hastings采样算法,Metropolis-Hastings采样算法也是基于MCMC的采样算法,是Metropolis采样算法的推广形式。
一、Metropolis-Hastings算法的基本原理
1、Metropolis-Hastings算法的基本原理
与Metropolis采样算法类似,假设需要从目标概率密度函数 p(θ) 中进行采样,同时, θ 满足 −∞<θ<∞ 。Metropolis-Hastings采样算法根据马尔可夫链去生成一个序列:
其中, θ(t) 表示的是马尔可夫链在第 t 代时的状态。
在Metropolis-Hastings采样算法的过程中,首先初始化状态值 θ(1) ,然后利用一个已知的分布 q(θ∣θ(t−1)) 生成一个新的候选状态 θ(∗) ,随后根据一定的概率选择接受这个新值,或者拒绝这个新值,与Metropolis采样算法不同的是,在Metropolis-Hastings采样算法中,概率为:
这样的过程一直持续到采样过程的收敛,当收敛以后,样本 θ(t) 即为目标分布 p(θ) 中的样本。
2、Metropolis-Hastings采样算法的流程
基于以上的分析,可以总结出如下的Metropolis-Hastings采样算法的流程:
- 初始化时间 t=1
- 设置 u 的值,并初始化初始状态 θ(t)=u
- 重复以下的过程:
- 令 t=t+1
- 从已知分布 q(θ∣θ(t−1)) 中生成一个候选状态 θ(∗)
- 计算接受的概率: α=min(1,p(θ(∗))p(θ(t−1))q(θ(t−1)∣θ(∗))q(θ(∗)∣θ(t−1)))
- 从均匀分布 Uniform(0,1) 生成一个随机值 a
- 如果 a⩽α ,接受新生成的值: θ(t)=θ(∗) ;否则: θ(t)=θ(t−1)
- 直到 t=T
3、Metropolis-Hastings采样算法的解释
与Metropolis采样算法类似,要证明Metropolis-Hastings采样算法的正确性,最重要的是要证明构造的马尔可夫过程满足如上的细致平稳条件,即:
对于上面所述的过程,分布为 p(θ) ,从状态 i 转移到状态 j 的转移概率为:
其中, Qi,j 为上述已知的分布, Qi,j 为:
在Metropolis-Hastings采样算法中,并不要求像Metropolis采样算法中的已知分布为对称的。
接下来,需要证明在Metropolis-Hastings采样算法中构造的马尔可夫链满足细致平稳条件。
因此,通过以上的方法构造出来的马尔可夫链是满足细致平稳条件的。
4、实验1
如前文的Metropolis采样算法,假设需要从柯西分布中采样数据,我们利用Metropolis-Hastings采样算法来生成样本,其中,柯西分布的概率密度函数为:
那么,根据上述的Metropolis-Hastings采样算法的流程,接受概率 α 的值为:
若已知的分布符合条件独立性,则
此时,与Metropolis采样算法一致。
二、多变量分布的采样
上述的过程中,都是针对的是单变量分布的采样,对于多变量的采样,Metropolis-Hastings采样算法通常有以下的两种策略:
- Blockwise Metropolis-Hastings采样
- Componentwise Metropolis-Hastings采样
1、Blockwise Metropolis-Hastings采样
对于BlockWise Metropolis-Hastings采样算法,在选择已知分布时,需要选择与目标分布具有相同维度的分布。针对上述的更新策略,在BlockWise Metropolis-Hastings采样算法中采用的是向量的更新策略,即: Θ=(θ1,θ2,⋯,θN) 。因此,算法流程为:
- 初始化时间 t=1
- 设置 u 的值,并初始化初始状态 Θ(t)=u
- 重复以下的过程:
- 令 t=t+1
- 从已知分布 q(Θ∣Θ(t−1)) 中生成一个候选状态 Θ(∗)
- 计算接受的概率: α=min(1,p(Θ(∗))p(Θ(t−1))q(Θ(t−1)∣Θ(∗))q(Θ(∗)∣Θ(t−1)))
- 从均匀分布 Uniform(0,1) 生成一个随机值 a
- 如果 a⩽α ,接受新生成的值: Θ(t)=Θ(∗) ;否则: Θ(t)=Θ(t−1)
- 直到 t=T
2、Componentwise Metropolis-Hastings采样
对于上述的BlockWise Metropolis-Hastings采样算法,有时很难找到与所要采样的分布具有相同维度的分布,因此可以采用Componentwise Metropolis-Hastings采样,该采样算法每次针对一维进行采样,其已知分布可以采用单变量的分布,算法流程为:
- 初始化时间 t=1
- 设置 u=(u1,u2,⋯,uN) 的值,并初始化初始状态 Θ(t)=u
- 重复以下的过程:
- 令 t=t+1
- 对每一维: i=1,2,⋯N
- 从已知分布 q(θi∣θ(t−1)i) 中生成一个候选状态 θ(∗)i ,假设没有更新之前的整个向量为: Θ(t−1)=(θ(t−1)1,⋯,θ(t−1)i,⋯,θ(t−1)N) ,更新之后的向量为: Θ=(θ(t−1)1,⋯,θ(∗)i,⋯,θ(t−1)N)
- 计算接受的概率: α=min(1,p(Θ)p(Θ(t−1))q(θ(t−1)i∣θ(∗)i)q(θ(∗)i∣θ(t−1)i))
- 从均匀分布 Uniform(0,1) 生成一个随机值 a
- 如果 a⩽α ,接受新生成的值: θ(t)i=θ(∗)i ;否则: θ(t)i=θ(t−1)i
- 直到 t=T
3、实验
假设希望从二元指数分布:
中进行采样,其中,假设 θ1 和 θ2 在区间 [0,8] , λ1=0.5 , λ2=0.1 , λ=0.01 , max(θ1,θ2)=8 。
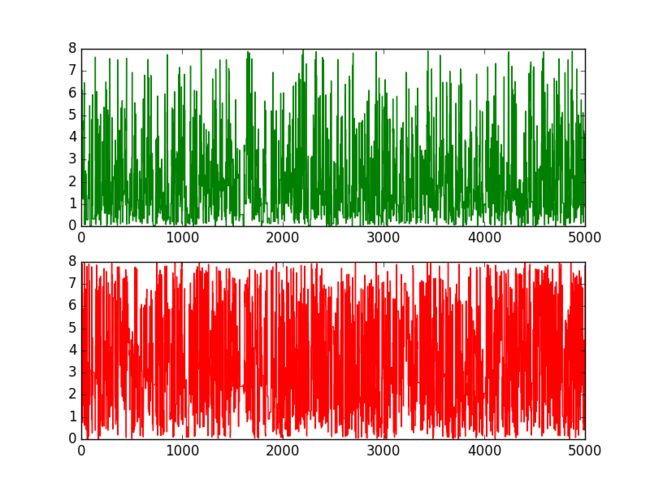
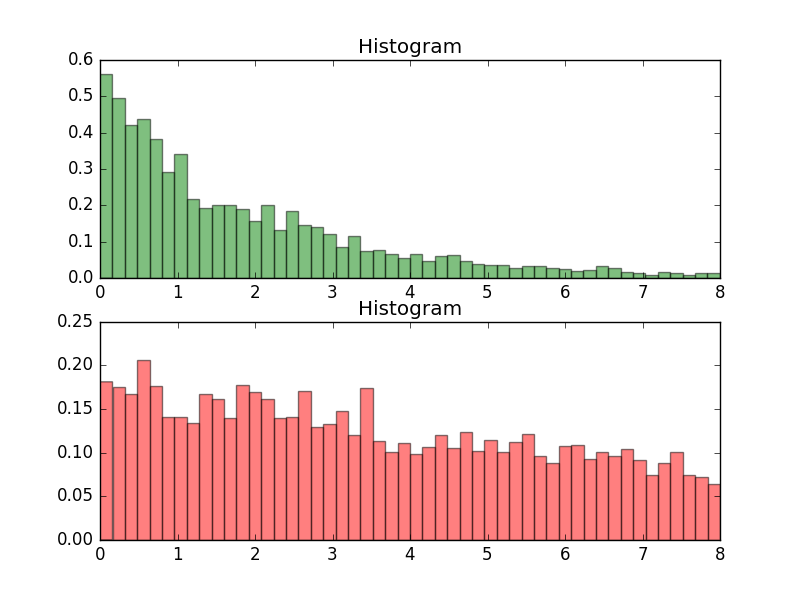
3.1、Blockwise
实验代码
''' Date:20160703 @author: zhaozhiyong '''
import random
import math
from scipy.stats import norm
import matplotlib.pyplot as plt
def bivexp(theta1, theta2):
lam1 = 0.5
lam2 = 0.1
lam = 0.01
maxval = 8
y = math.exp(-(lam1 + lam) * theta1 - (lam2 + lam) * theta2 - lam * maxval)
return y
T = 5000
sigma = 1
thetamin = 0
thetamax = 8
theta_1 = [0.0] * (T + 1)
theta_2 = [0.0] * (T + 1)
theta_1[0] = random.uniform(thetamin, thetamax)
theta_2[0] = random.uniform(thetamin, thetamax)
t = 0
while t < T:
t = t + 1
theta_star_0 = random.uniform(thetamin, thetamax)
theta_star_1 = random.uniform(thetamin, thetamax)
# print theta_star
alpha = min(1, (bivexp(theta_star_0, theta_star_1) / bivexp(theta_1[t - 1], theta_2[t - 1])))
u = random.uniform(0, 1)
if u <= alpha:
theta_1[t] = theta_star_0
theta_2[t] = theta_star_1
else:
theta_1[t] = theta_1[t - 1]
theta_2[t] = theta_2[t - 1]
plt.figure(1)
ax1 = plt.subplot(211)
ax2 = plt.subplot(212)
plt.ylim(thetamin, thetamax)
plt.sca(ax1)
plt.plot(range(T + 1), theta_1, 'g-', label="0")
plt.sca(ax2)
plt.plot(range(T + 1), theta_2, 'r-', label="1")
plt.show()
plt.figure(2)
ax1 = plt.subplot(211)
ax2 = plt.subplot(212)
num_bins = 50
plt.sca(ax1)
plt.hist(theta_1, num_bins, normed=1, facecolor='green', alpha=0.5)
plt.title('Histogram')
plt.sca(ax2)
plt.hist(theta_2, num_bins, normed=1, facecolor='red', alpha=0.5)
plt.title('Histogram')
plt.show()实验结果
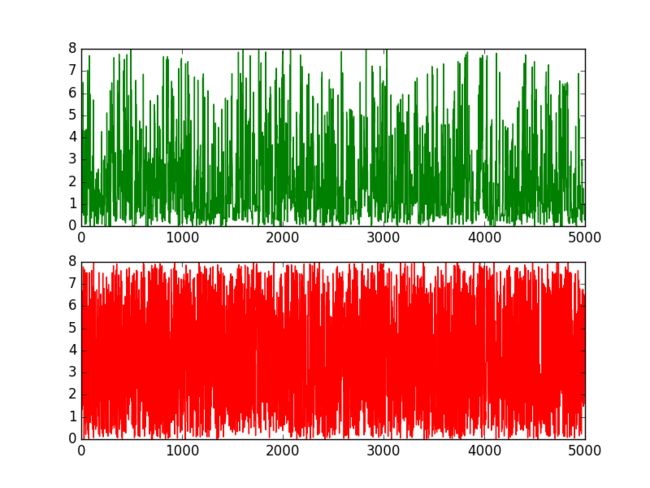
3.2、Componentwise
实验代码
''' Date:20160703 @author: zhaozhiyong '''
import random
import math
from scipy.stats import norm
import matplotlib.pyplot as plt
def bivexp(theta1, theta2):
lam1 = 0.5
lam2 = 0.1
lam = 0.01
maxval = 8
y = math.exp(-(lam1 + lam) * theta1 - (lam2 + lam) * theta2 - lam * maxval)
return y
T = 5000
sigma = 1
thetamin = 0
thetamax = 8
theta_1 = [0.0] * (T + 1)
theta_2 = [0.0] * (T + 1)
theta_1[0] = random.uniform(thetamin, thetamax)
theta_2[0] = random.uniform(thetamin, thetamax)
t = 0
while t < T:
t = t + 1
# step 1
theta_star_1 = random.uniform(thetamin, thetamax)
alpha = min(1, (bivexp(theta_star_1, theta_2[t - 1]) / bivexp(theta_1[t - 1], theta_2[t - 1])))
u = random.uniform(0, 1)
if u <= alpha:
theta_1[t] = theta_star_1
else:
theta_1[t] = theta_1[t - 1]
# step 2
theta_star_2 = random.uniform(thetamin, thetamax)
alpha = min(1, (bivexp(theta_1[t], theta_star_2) / bivexp(theta_1[t], theta_2[t - 1])))
u = random.uniform(0, 1)
if u <= alpha:
theta_2[t] = theta_star_2
else:
theta_2[t] = theta_2[t - 1]
plt.figure(1)
ax1 = plt.subplot(211)
ax2 = plt.subplot(212)
plt.ylim(thetamin, thetamax)
plt.sca(ax1)
plt.plot(range(T + 1), theta_1, 'g-', label="0")
plt.sca(ax2)
plt.plot(range(T + 1), theta_2, 'r-', label="1")
plt.show()
plt.figure(2)
ax1 = plt.subplot(211)
ax2 = plt.subplot(212)
num_bins = 50
plt.sca(ax1)
plt.hist(theta_1, num_bins, normed=1, facecolor='green', alpha=0.5)
plt.title('Histogram')
plt.sca(ax2)
plt.hist(theta_2, num_bins, normed=1, facecolor='red', alpha=0.5)
plt.title('Histogram')
plt.show()实验结果