遗传算法简单介绍与MATLAB实现(二)
遗传算法简单介绍与MATLAB实现(二)
引入题目一
上一篇文章中我们简单的介绍了一了一下遗传算法,其中提到了多元函数 f=f(x,y) ,所以在这里我们就定义一个二元函数,作为第一个练手的程序。
我们在这里令
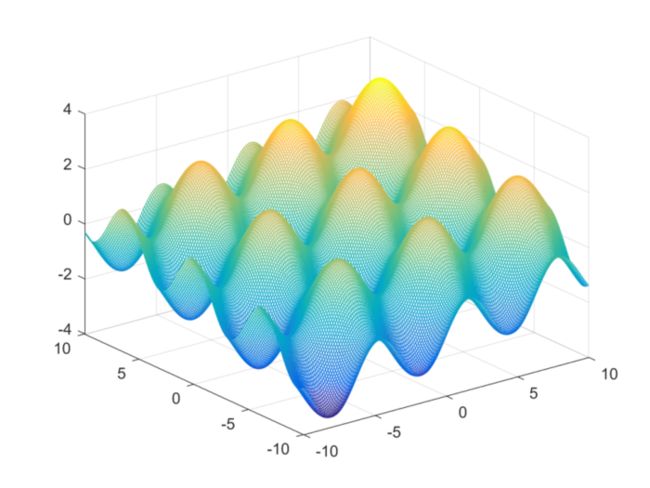
通过使用MATLAB作图可以看到下图
可以看出这是一个存在很多区域极大值,有很多“坑”。这正是遗传算法的缺陷之一——求出来的最优解可能并不是全局最优解而是局部最优解。
从图中可以看出全局最大值在 (10,10) 附近。所以接下来我们就开始编程解决这个问题。
遗传算法MATLAB程序
设定参数
首先我们要确定一系列参数
N = 100; %种群内个体数目
N_chrom = 2; %染色体节点数
iter = 2000; %迭代次数
mut = 0.2; %突变概率
acr = 0.2; %交叉概率其中突变概率指的是某个个体的染色体发生突变的概率。因为实际中并不可能每一个个体都会发生突变,而是存在一定的概率,因此我们在这里定义每一个个体突变发生的概率为0.2。
同理,交叉概率指的就是某个个体与另一个个体发生染色体交叉的概率。
迭代次数指的是整个种群迭代2000多次。
一般来说,并不是突变概率、交叉概率越小或者越大越好。比如说突变概率特别高的话,会让整个种群的最优适应度飘忽不定。如果特别小的话,会让种群迭代的速度特别慢,很难找到最优适应度。这里取0.2是我经过多次试验以后,觉得0.2是一个比较稳定不会乱飘且适应度收敛也不错的一个值。
迭代次数也同理,如果一个特别简单的模型去求最优解,可能五六次迭代就求出来了,那么2000次迭代纯属浪费时间。但是如果是一个特别复杂的模型,那么2000次迭代就有可能不够。具体迭代次数可以自己灵活决定。
chrom_range = [-10 -10; 10 10];%每个节点的值的区间
chrom = zeros(N, N_chrom);%存放染色体的矩阵
fitness = zeros(N, 1);%存放染色体的适应度
fitness_ave = zeros(1, iter);%存放每一代的平均适应度
fitness_best = zeros(1, iter);%存放每一代的最优适应度
chrom_best = zeros(1, N_chrom+1);%存放当前代的最优染色体与适应度其中的chrom_range指的是每个节点值的区间。在我们的这个模型中, x∈[−10,10],y∈[−10,10] 。第一行放的是两个变量的区间下限,第二行放的是区间上限。
chrom和fitness两个矩阵你们可以自己理解一下为什么这样子写。
fitness_ave用来存放每一代的平均适应度,主要是用来最后画图,观察种群在每一次迭代中适应度的变化情况。fitness_best同理。
chrom_best用来存放最优适应度对应的最优染色体与适应度,可以回想一下上一章染色体变异中需要最优适应度,优胜劣汰中需要最优染色体,这个矩阵作用就是如此。
初始化种群
接下来开始初始化参数
chrom = Initialize(N, N_chrom, chrom_range); %初始化染色体
fitness = CalFitness(chrom, N, N_chrom); %计算适应度
chrom_best = FindBest(chrom, fitness, N_chrom); %寻找最优染色体
fitness_best(1) = chrom_best(end); %将当前最优存入矩阵当中
fitness_ave(1) = CalAveFitness(fitness); %将当前平均适应度存入矩阵当中Initialize函数是我自定义的一个函数,用于初始化染色体。具体操作就是用一个for循环对每个个体的染色体进行随机赋值,并利用chrom_range将其限定在变量规定的区间之内。
function chrom_new = Initialize(N, N_chrom, chrom_range)
chrom_new = rand(N, N_chrom);
for i = 1:N_chrom %每一列乘上范围
chrom_new(:, i) = chrom_new(:, i)*(chrom_range(2, i)-chrom_range(1, i))+chrom_range(1, i);
endCalFitness函数则是计算chrom内每个个体的适应度,并将其保存在fitness中。这个函数很重要,请着重理解这个函数的用法。
function fitness = CalFitness(chrom, N, N_chrom)
fitness = zeros(N, 1);
%开始计算适应度
for i = 1:N
x = chrom(i, 1);
y = chrom(i, 2);
fitness(i) = sin(x)+cos(y)+0.1*x+0.1*y;
endFindBest函数寻找出当前种群中的最优染色体,并将其保存在chrom_best。
function chrom_best = FindBest(chrom, fitness, N_chrom)
chrom_best = zeros(1, N_chrom+1);
[maxNum, maxCorr] = max(fitness);
chrom_best(1:N_chrom) =chrom(maxCorr, :);
chrom_best(end) = maxNum;CalAveFitness函数用于计算当前种群中的平均适应度。
function fitness_ave = CalAveFitness(fitness)
[N ,~] = size(fitness);
fitness_ave = sum(fitness)/N;迭代开始
初始化完以后就开始迭代,为了保证迭代次数和iter一致,这里我们就直接令t从2开始迭代。
再一次迭代过程中,一个种群执行操作顺序为:染色体变异,染色体交叉,计算适应度,寻找最优染色体,替换当前存储的最优染色体,优胜劣汰。
需要注意的是这里替换当前最优染色体。因为我们将这个种群的个个个体的染色体重新变异、交叉过了,所以最优染色体也会变化。但是这一代的最优染色体不一定比上一次迭代的最优染色体好,所以需要判定是否将最优染色体这个位置给换掉。
整体的流程图为
因此迭代过程中的代码为
for t = 2:iter
chrom = MutChrom(chrom, mut, N, N_chrom, chrom_range, t, iter); %变异
chrom = AcrChrom(chrom, acr, N, N_chrom); %交叉
fitness = CalFitness(chrom, N, N_chrom); %计算适应度
chrom_best_temp = FindBest(chrom, fitness, N_chrom); %寻找最优染色体
if chrom_best_temp(end)>chrom_best(end) %替换掉当前储存的最优
chrom_best = chrom_best_temp;
end
%%替换掉最劣
[chrom, fitness] = ReplaceWorse(chrom, chrom_best, fitness);
fitness_best(t) = chrom_best(end); %将当前最优存入矩阵当中
fitness_ave(t) = CalAveFitness(fitness); %将当前平均适应度存入矩阵当中
end其中MutChrom函数用来对种群进行染色体变异操作。这里面的操作比较多,主要可以分为三步:
- 判断某节点是否变异;
- 判断变异是增加还是减少;
- 判断变异后的值是否越界。
用流程图表示为下图所示。
下面代码表示为
function chrom_new = MutChrom(chrom, mut, N, N_chrom, chrom_range, t, iter)
for i = 1:N
for j = 1:N_chrom
mut_rand = rand; %是否变异
if mut_rand <=mut
mut_pm = rand; %增加还是减少
mut_num = rand*(1-t/iter)^2;
if mut_pm<=0.5
chrom(i, j)= chrom(i, j)*(1-mut_num);
else
chrom(i, j)= chrom(i, j)*(1+mut_num);
end
chrom(i, j) = IfOut(chrom(i, j), chrom_range(:, j)); %检验是否越界
end
end
end
chrom_new = chrom;越界函数IfOut函数我们看它是左越界还是右越界,如果是左越界,则让节点值等于区间下限;如果是右越界,则让节点值等于区间上限。
function c_new = IfOut(c, range)
if c1) || c>range(2)
if abs(c-range(1))<abs(c-range(2))
c_new = range(1);
else
c_new = range(2);
end
else
c_new = c;
end AcrChrom函数是染色体交叉函数。逻辑相较染色体变异来说比较简单,大致可以分为以下几点
- 判断该节点是否交叉;
- 随机寻找一个与其交叉的节点;
- 对两个节点进行交叉。
function chrom_new = AcrChrom(chrom, acr, N, N_chrom)
for i = 1:N
acr_rand = rand;
if acr_rand%如果交叉
acr_chrom = floor((N-1)*rand+1); %要交叉的染色体
acr_node = floor((N_chrom-1)*rand+1); %要交叉的节点
%交叉开始
temp = chrom(i, acr_node);
chrom(i, acr_node) = chrom(acr_chrom, acr_node);
chrom(acr_chrom, acr_node) = temp;
end
end
chrom_new = chrom; ReplaceWorse函数则是优胜劣汰这个过程,将种群中的最劣染色体替换掉。这里是直接用最优染色体替换,并且替换掉的是最后几个染色体,目的是加快收敛速度,但可能会陷入局部最优解。
function [chrom_new, fitness_new] = ReplaceWorse(chrom, chrom_best, fitness)
max_num = max(fitness);
min_num = min(fitness);
limit = (max_num-min_num)*0.2+min_num;
replace_corr = fitnessones(replace_num, 1)*chrom_best(1:end-1);
fitness(replace_corr) = ones(replace_num, 1)*chrom_best(end);
chrom_new = chrom;
fitness_new = fitness;
end
输出遗传算法迭代图
在这里我们只输出一个标准图为迭代过程中种群的平均适应度与最优适应度。
在之前我们已经得到了每一代的平均适应度fitness_ave与最优适应度fitness_best,所以可以直接输出图像
figure(1)
plot(1:iter, fitness_ave, 'r', 1:iter, fitness_best, 'b')
grid on
legend('平均适应度', '最优适应度')之后我们还想要看得出的点在实际图像中哪个位置,所以我们可以增加一个用来作图的函数PlotModel
function y = PlotModel(chrom)
x = chrom(1);
y = chrom(2);
z = chrom(3);
figure(2)
scatter3(x, y, z, 'ko')
hold on
[X, Y] = meshgrid(-10:0.1:10);
Z =sin(X)+cos(Y)+0.1*X+0.1*Y;
mesh(X, Y, Z)
y=1;遗传算法主程序
上面就是所有程序片段,因此把主程序的代码联立起来为
clc, clear, close all
%%基础参数
N = 100; %种群内个体数目
N_chrom = 2; %染色体节点数
iter = 2000; %迭代次数
mut = 0.2; %突变概率
acr = 0.2; %交叉概率
best = 1;
chrom_range = [-10 -10;10 10];%每个节点的值的区间
chrom = zeros(N, N_chrom);%存放染色体的矩阵
fitness = zeros(N, 1);%存放染色体的适应度
fitness_ave = zeros(1, iter);%存放每一代的平均适应度
fitness_best = zeros(1, iter);%存放每一代的最优适应度
chrom_best = zeros(1, N_chrom+1);%存放当前代的最优染色体与适应度
%%初始化
chrom = Initialize(N, N_chrom, chrom_range); %初始化染色体
fitness = CalFitness(chrom, N, N_chrom); %计算适应度
chrom_best = FindBest(chrom, fitness, N_chrom); %寻找最优染色体
fitness_best(1) = chrom_best(end); %将当前最优存入矩阵当中
fitness_ave(1) = CalAveFitness(fitness); %将当前平均适应度存入矩阵当中
for t = 2:iter
chrom = MutChrom(chrom, mut, N, N_chrom, chrom_range, t, iter); %变异
chrom = AcrChrom(chrom, acr, N, N_chrom); %交叉
fitness = CalFitness(chrom, N, N_chrom); %计算适应度
chrom_best_temp = FindBest(chrom, fitness, N_chrom); %寻找最优染色体
if chrom_best_temp(end)>chrom_best(end) %替换掉当前储存的最优
chrom_best = chrom_best_temp;
end
%%替换掉最劣
[chrom, fitness] = ReplaceWorse(chrom, chrom_best, fitness);
fitness_best(t) = chrom_best(end); %将当前最优存入矩阵当中
fitness_ave(t) = CalAveFitness(fitness); %将当前平均适应度存入矩阵当中
end
%%作图
figure(1)
plot(1:iter, fitness_ave, 'r', 1:iter, fitness_best, 'b')
grid on
legend('平均适应度', '最优适应度')
e = PlotModel(chrom_best)
%%输出结果
disp(['最优染色体为', num2str(chrom_best(1:end-1))])
disp(['最优适应度为', num2str(chrom_best(end))])输出结果
我们运行一下, 可以得到最大点为 (7.9541,6.3834) ,最大值为3.4237。
可以做出最大值点在空间中的位置如图所示。证明我们的结论比较正确,起码没有落到周围的极大值的峰上。
下章预告
本章只是求解了一个二元函数,下一章我们将来解决一个实际模型,来看一下遗传算法怎么求解不同的模型。