(4)聚类算法之OPTICS算法
文章目录
- 1.引言
- 2.相关定义
- 2.1 `DBSCAN`相关定义
- 2.2 `OPTICS`相关定义
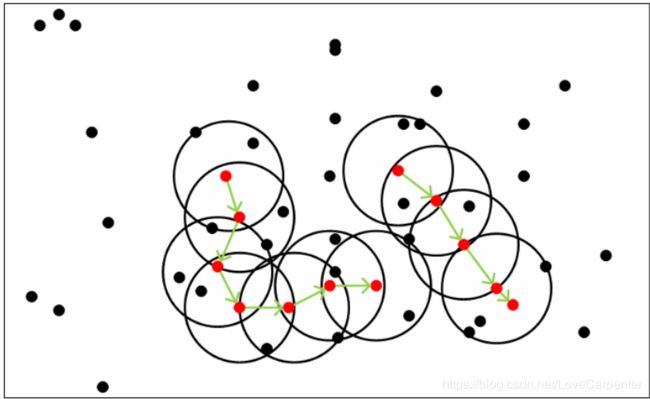
- 3.算法思想
- 3.1算法流程
- 3.2算法伪代码
- 4.算法实现
- 4.1使用`numpy`实现OPTICS算法
- 5.数据及代码下载地址
1.引言
OPTICS(Ordering points to identify the clustering structure)是一基于密度的聚类算法,OPTICS算法是DBSCAN的改进版本,因此OPTICS算法也是一种基于密度的聚类算法。在DBCSAN算法中需要输入两个参数: ϵ ϵ ϵ和 M i n P t s MinPts MinPts,选择不同的参数会导致最终聚类的结果千差万别,因此DBCSAN对于输入参数过于敏感。OPTICS算法的提出就是为了帮助DBSCAN算法选择合适的参数,降低输入参数的敏感度。OPTICS主要针对输入参数 ϵ ϵ ϵ过敏感做的改进,OPTICS和DBSCNA的输入参数一样( ϵ ϵ ϵ和 M i n P t s MinPts MinPts),虽然OPTICS算法中也需要两个输入参数,但该算法对 ϵ ϵ ϵ输入不敏感(一般将 ϵ ϵ ϵ固定为无穷大),同时该算法中并不显式的生成数据聚类,只是对数据集合中的对象进行排序,得到一个有序的对象列表,通过该有序列表,可以得到一个决策图,通过决策图可以不同 ϵ ϵ ϵ参数的数据集中检测簇集,即:先通过固定的 M i n P t s MinPts MinPts和无穷大的 ϵ ϵ ϵ得到有序列表,然后得到决策图,通过决策图可以知道当 ϵ ϵ ϵ取特定值时(比如 ϵ = 3 ϵ=3 ϵ=3)数据的聚类情况。
2.相关定义
由于OPTICS算法是DBSCAN算法的一种改进,因此有些概念是共用的,比如: ϵ ϵ ϵ-邻域,核心对象,密度直达,密度可达,密度相连等,下面是与OPTICS相关的定义(假设我的样本集是 X = ( x 1 , x 2 , . . . , x m ) X=(x_1,x_2,...,x_m) X=(x1,x2,...,xm)):
2.1 DBSCAN相关定义
- ϵ ϵ ϵ-邻域:对于 x j ∈ X x_j∈X xj∈X,其 ϵ ϵ ϵ-邻域包含样本集 X X X中与 x j x_j xj的距离不大于 ϵ ϵ ϵ的子样本集。 ϵ ϵ ϵ-邻域是一个集合,表示如下,这个集合的个数记为 ∣ N ϵ ( x j ) ∣ |N_ϵ(x_j)| ∣Nϵ(xj)∣。
N ϵ ( x j ) = { x i ∈ X ∣ d i s t a n c e ( x i , x j ) ≤ ϵ } N_ϵ(x_j)=\{x_i∈X \mid distance(x_i,x_j)≤ϵ\} Nϵ(xj)={xi∈X∣distance(xi,xj)≤ϵ} - 核心对象:对于任一样本 x j ∈ X x_j∈X xj∈X,如果其 ϵ ϵ ϵ-邻域对应的 N ϵ ( x j ) N_ϵ(x_j) Nϵ(xj)至少包含 M i n P t s MinPts MinPts个样本,即如果 ∣ N ϵ ( x j ) ∣ ≥ M i n P t s |N_ϵ(x_j)|≥MinPts ∣Nϵ(xj)∣≥MinPts,则 x j x_j xj是核心对象。
- 密度直达:如果 x i x_i xi位于 x j x_j xj的 ϵ ϵ ϵ-邻域中,且 x j x_j xj是核心对象,则称 x i x_i xi由 x j x_j xj密度直达。反之不一定成立,即此时不能说 x j x_j xj由 x i x_i xi密度直达, 除非且 x i x_i xi也是核心对象,即密度直达不满足对称性
- 密度可达:对于 x i x_i xi和 x j x_j xj,如果存在样本样本序列 p 1 , p 2 , . . . , p T p_1,p_2,...,p_T p1,p2,...,pT,满足 p 1 = x i , p T = x j p1=x_i,p_T=x_j p1=xi,pT=xj, 且 p t + 1 p_{t+1} pt+1由 p t p_t pt密度直达,则称 x j x_j xj由 x i x_i xi密度可达。也就是说,密度可达满足传递性。此时序列中的传递样本 p 1 , p 2 , . . . , p T − 1 p_1,p_2,...,p_{T−1} p1,p2,...,pT−1均为核心对象,因为只有核心对象才能使其他样本密度直达。 密度可达也不满足对称性,这个可以由密度直达的不对称性得出。
- 密度相连:对于 x i x_i xi和 x j x_j xj,如果存在核心对象样本 x k x_k xk,使** x i x_i xi和 x j x_j xj均由 x k x_k xk密度可达**,则称 x i x_i xi和 x j x_j xj密度相连。密度相连关系满足对称性。
2.2 OPTICS相关定义
在上述DBSCAN定义的基础上,OPTICS在引入了两个算法需要的定义:
- 核心距离(core-distance):样本 x ∈ X x∈X x∈X,对于给定的 ϵ ϵ ϵ和 M i n P t s MinPts MinPts,使得 x x x成为核心点的最小邻域半径称为 x x x的核心距离,其数学表达如下, N ϵ i ( x ) N_ϵ^{i}(x) Nϵi(x)代表集合 N ϵ ( x ) N_ϵ(x) Nϵ(x)中与节点 x x x第 i i i近邻的节点,如 N ϵ 1 ( x ) N_ϵ^{1}(x) Nϵ1(x)表示 N ϵ ( x ) N_ϵ(x) Nϵ(x)中与 x x x最近的节点
c d ( x ) = { u n d e f i n e d ∣ N ϵ ( x ) ∣ < M i n P t s d ( x , N ϵ M i n P t s ( x ) ) ∣ N ϵ ( x ) ∣ > = M i n P t s cd(x)=\begin{cases} undefined & |N_ϵ(x)| <MinPts \\ d(x,N_ϵ^{MinPts}(x) ) & |N_ϵ(x)| >=MinPts \end{cases} cd(x)={undefinedd(x,NϵMinPts(x))∣Nϵ(x)∣<MinPts∣Nϵ(x)∣>=MinPts
- 可达距离(reachability-distance):设 x , y ∈ X x,y∈X x,y∈X,对于给定的 ϵ ϵ ϵ和 M i n P t s MinPts MinPts, y y y关于 x x x的可达距离定义为:
r d ( y , x ) = { u n d e f i n e d ∣ N ϵ ( x ) ∣ < M i n P t s m a x { c d ( x ) , d ( x , y ) } ∣ N ϵ ( x ) ∣ > = M i n P t s rd(y,x)=\begin{cases} undefined & |N_ϵ(x)| <MinPts \\ max\{ cd(x),d(x,y) \} & |N_ϵ(x)| >=MinPts \end{cases} rd(y,x)={undefinedmax{cd(x),d(x,y)}∣Nϵ(x)∣<MinPts∣Nϵ(x)∣>=MinPts
特别的,当 x x x为核心点时(相应的参数为 ϵ ϵ ϵ和 M i n P t s MinPts MinPts),可按照下式来理解 r d ( y , x ) rd(y,x) rd(y,x):
r d ( y , x ) = m i n { η : y ∈ N η ( x ) 且 ∣ N η ( x ) ∣ ≥ M i n P t s } rd(y,x)=min\{ \eta: y ∈ N_{\eta}(x) 且 | N_{\eta}(x) | \ge MinPts\} rd(y,x)=min{η:y∈Nη(x)且∣Nη(x)∣≥MinPts}
即 r d ( y , x ) rd(y,x) rd(y,x)表示 使得“ x x x成为核心点”,“ y y y可以从 x x x直接密度可达” 同时成立的最小邻域半径。
可达距离这里可能不太好理解,先记住一点,每一个点都有两个新属性:可达距离,核心距离
3.算法思想
假设我们的数据集为 X = ( x 1 , x 2 , . . . , x m ) X=(x_1,x_2,...,x_m) X=(x1,x2,...,xm),OPTICS算法的目标是输出一个有序排列,以及每个元素的两个属性值:核心距离,可达距离。为此引入如下的数据结构:
- p i , i = 1 , 2 , . . . , N p_i,i=1,2,...,N pi,i=1,2,...,N:OPTICS算法的输出有序列表,例如 p = { 10 , 100 , 4 , . . . } p=\{10,100,4,...\} p={10,100,4,...}表示:在集合X中的数据,第10号节点首先被处理,然后第100号节点被处理,然后第4号节点被处理(即节点被处理的顺序列表)
- c i , i = 1 , 2 , . . . , N c_i,i=1,2,...,N ci,i=1,2,...,N:第 i i i号节点的核心距离,例如 c = { 1.2 , 1.4 , 4.5 , . . . } c=\{1.2,1.4,4.5,...\} c={1.2,1.4,4.5,...}表示:在集合X中的数据,第1号节点的核心距离为1.2,第1号节点的核心距离为1.4,第1号节点的核心距离为4.5
- r i , i = 1 , 2 , . . . , N r_i,i=1,2,...,N ri,i=1,2,...,N:第 i i i号节点的可达距离,例如 r = { 3.4 , 3.1.4 , 4.5 , . . . } r=\{3.4,3.1.4,4.5,...\} r={3.4,3.1.4,4.5,...}表示:在集合X中的数据,第1号节点的可达距离为3.4,第1号节点的可达距离为3.1,第1号节点的可达距离为4.5
3.1算法流程
输入:样本集 X = ( x 1 , x 2 , . . . , x m ) X=(x_1,x_2,...,x_m) X=(x1,x2,...,xm),邻域参数 ( ϵ , M i n P t s ) (ϵ,MinPts) (ϵ,MinPts)
- 初始化核心对象集合 Ω = ∅ Ω=∅ Ω=∅
- 遍历 X X X的元素,如果是核心对象,则将其加入到核心对象集合 Ω Ω Ω中
- 如果核心对象集合 Ω Ω Ω中元素都已经被处理,则算法结束,否则转入步骤4.
- 在核心对象集合 Ω Ω Ω中,随机选择一个未处理的核心对象 o o o,首先将 o o o标记为已处理,同时将 o o o压入到有序列表 p p p中,最后将 o o o的 ϵ ϵ ϵ-邻域中未访问的点,根据可达距离的大小(计算未访问的邻居点到 o o o点的可达距离)依次存放到种子集合 s e e d s seeds seeds中。
- 如果种子集合 s e e d s = ∅ seeds=∅ seeds=∅,跳转到3,否则,从种子集合 s e e d s seeds seeds中挑选可达距离最近的种子点 s e e d seed seed,首先将其标记为已访问,首先将 s e e d seed seed标记为已处理,同时将 s e e d seed seed压入到有序列表 p p p中,然后判断 s e e d seed seed是否为核心对象,如果是将 s e e d seed seed中未访问的邻居点加入到种子集合中,重新计算可达距离。(计算种子集合中距离 s e e d seed seed点的可达距离)跳转到5。
说明:
- 第一点,第一个被处理的对象是不存在可达距离的 (因为没有被计算过),只有进入过 s e e d s seeds seeds的点才能计算可达距离
3.2算法伪代码
OPTICS算法伪代码
update算法伪代码
4.算法实现
4.1使用numpy实现OPTICS算法
import numpy as np
import matplotlib.pyplot as plt
import time
import operator
from scipy.spatial.distance import pdist
from scipy.spatial.distance import squareform
def compute_squared_EDM(X):
return squareform(pdist(X,metric='euclidean'))
# 显示决策图
def plotReachability(data,eps):
plt.figure()
plt.plot(range(0,len(data)), data)
plt.plot([0, len(data)], [eps, eps])
plt.show()
# 显示分类的类别
def plotFeature(data,labels):
clusterNum = len(set(labels))
fig = plt.figure()
scatterColors = ['black', 'blue', 'green', 'yellow', 'red', 'purple', 'orange', 'brown']
ax = fig.add_subplot(111)
for i in range(-1, clusterNum):
colorSytle = scatterColors[i % len(scatterColors)]
subCluster = data[np.where(labels == i)]
ax.scatter(subCluster[:, 0], subCluster[:, 1], c=colorSytle, s=12)
plt.show()
def updateSeeds(seeds,core_PointId,neighbours,core_dists,reach_dists,disMat,isProcess):
# 获得核心点core_PointId的核心距离
core_dist=core_dists[core_PointId]
# 遍历core_PointId 的每一个邻居点
for neighbour in neighbours:
# 如果neighbour没有被处理过,计算该核心距离
if(isProcess[neighbour]==-1):
# 首先计算改点的针对core_PointId的可达距离
new_reach_dist = max(core_dist, disMat[core_PointId][neighbour])
# 如果可达距离没有被计算过,将计算的可达距离赋予
if(np.isnan(reach_dists[neighbour])):
reach_dists[neighbour]=new_reach_dist
seeds[neighbour] = new_reach_dist
# 如果可达距离已经被计算过,判读是否要进行修改
elif(new_reach_dist<reach_dists[neighbour]):
reach_dists[neighbour] = new_reach_dist
seeds[neighbour] = new_reach_dist
return seeds
def OPTICS(data,eps=np.inf,minPts=15):
# 获得距离矩阵
orders = []
disMat = compute_squared_EDM(data)
# 获得数据的行和列(一共有n条数据)
n, m = data.shape
# np.argsort(disMat)[:,minPts-1] 按照距离进行 行排序 找第minPts个元素的索引
# disMat[np.arange(0,n),np.argsort(disMat)[:,minPts-1]] 计算minPts个元素的索引的距离
temp_core_distances = disMat[np.arange(0,n),np.argsort(disMat)[:,minPts-1]]
# 计算核心距离
core_dists = np.where(temp_core_distances <= eps, temp_core_distances, -1)
# 将每一个点的可达距离未定义
reach_dists= np.full((n,), np.nan)
# 将矩阵的中小于minPts的数赋予1,大于minPts的数赋予零,然后1代表对每一行求和,然后求核心点坐标的索引
core_points_index = np.where(np.sum(np.where(disMat <= eps, 1, 0), axis=1) >= minPts)[0]
# 用于标识是否被处理,没有被处理,设置为-1
isProcess = np.full((n,), -1)
# 遍历所有的核心点
for pointId in core_points_index:
# 如果核心点未被分类,将其作为的种子点,开始寻找相应簇集
if (isProcess[pointId] == -1):
# 将点pointId标记为当前类别(即标识为已操作)
isProcess[pointId] = 1
orders.append(pointId)
# 寻找种子点的eps邻域且没有被分类的点,将其放入种子集合
neighbours = np.where((disMat[:, pointId] <= eps) & (disMat[:, pointId] > 0) & (isProcess == -1))[0]
seeds = dict()
seeds=updateSeeds(seeds,pointId,neighbours,core_dists,reach_dists,disMat,isProcess)
while len(seeds)>0:
nextId = sorted(seeds.items(), key=operator.itemgetter(1))[0][0]
del seeds[nextId]
isProcess[nextId] = 1
orders.append(nextId)
# 寻找newPoint种子点eps邻域(包含自己)
# 这里没有加约束isProcess == -1,是因为如果加了,本是核心点的,可能就变成了非和核心点
queryResults = np.where(disMat[:, nextId] <= eps)[0]
if len(queryResults) >= minPts:
seeds=updateSeeds(seeds,nextId,queryResults,core_dists,reach_dists,disMat,isProcess)
# 簇集生长完毕,寻找到一个类别
# 返回数据集中的可达列表,及其可达距离
return orders,reach_dists
def extract_dbscan(data,orders, reach_dists, eps):
# 获得原始数据的行和列
n,m=data.shape
# reach_dists[orders] 将每个点的可达距离,按照有序列表排序(即输出顺序)
# np.where(reach_dists[orders] <= eps)[0],找到有序列表中小于eps的点的索引,即对应有序列表的索引
reach_distIds=np.where(reach_dists[orders] <= eps)[0]
# 正常来说:current的值的值应该比pre的值多一个索引。如果大于一个索引就说明不是一个类别
pre=reach_distIds[0]-1
clusterId=0
labels=np.full((n,),-1)
for current in reach_distIds:
# 正常来说:current的值的值应该比pre的值多一个索引。如果大于一个索引就说明不是一个类别
if(current-pre!=1):
# 类别+1
clusterId=clusterId+1
labels[orders[current]]=clusterId
pre=current
return labels
data = np.loadtxt("cluster2.csv", delimiter=",")
start = time.clock()
orders,reach_dists=OPTICS(data,np.inf,30)
end = time.clock()
print('finish all in %s' % str(end - start))
labels=extract_dbscan(data,orders,reach_dists,3)
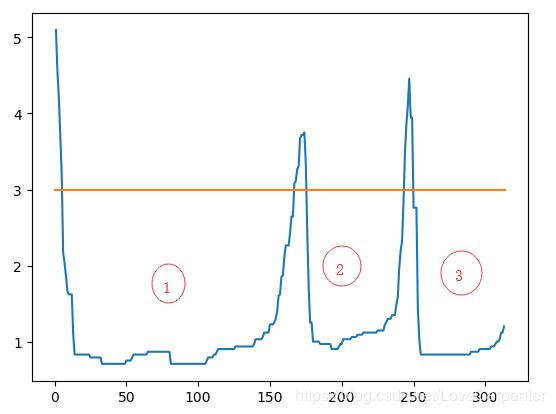
plotReachability(reach_dists[orders],3)
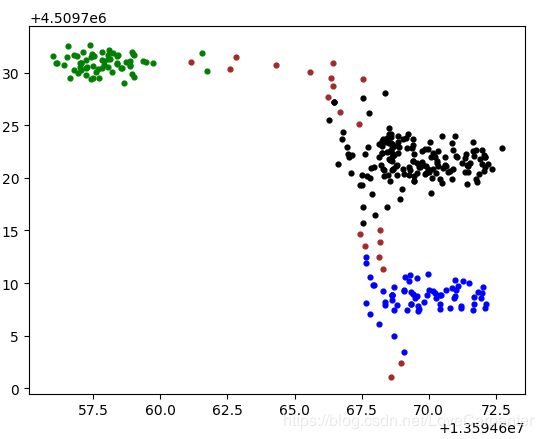
plotFeature(data,labels)
- 有序列表决策图(横坐标是处理顺序,纵坐标是该点的可达距离),举个例子,横坐标为: [ 1 , 2 , 3 ] [1,2,3] [1,2,3],纵坐标为: [ 5.5 , 3.6 , 8.4 ] [5.5,3.6,8.4] [5.5,3.6,8.4]。说明:第一个被处理的点的可达距离为5.5,第二个被处理的点的可达距离为3.6,第三个被处理的点的可达距离为8.4。同时在该图中可以看出,当eps取3时,原数据集可以被分为3个类别(决策图有一个凹槽).
- 聚类结果可视化图(棕色是离群点)
5.数据及代码下载地址
- GitHub的数据及代码下载地址为:GitHub的数据及代码下载链接(如果从GitHub下载代码,麻烦给小
Demo一个Star,您的支持是我最大的动力)