主要内容
需要熟悉:UTF-8, GBK编码使用方式
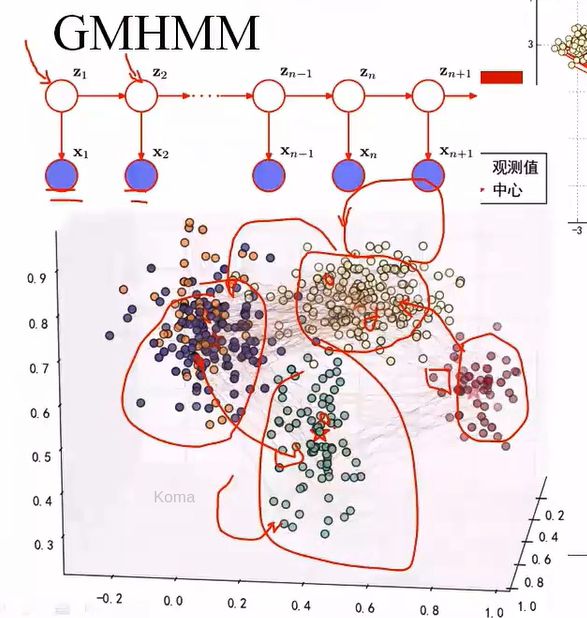
如果需要使用隐马尔科夫模型,首先状态一定是离散的,观测值不一定要求是离散的,可以做观测值是连续的隐马尔科夫模型
中文分词
如下图:A矩阵是4x4的矩阵(Begin Middle End Single), B矩阵是4x65536的矩阵,π是4个元素的矩阵
用Viterbi算法做文档的切词,现在比较常用是直接用jieba组件做分词
import jieba.posseg
if __name__ == "__main__":
f = open('.\\novel.txt', encoding='utf-8')
str = f.read()#.decode('utf-8')
f.close()
seg = jieba.posseg.cut(str)
for word, pos in seg:
print(word, pos, '|', end=' ')
分词结果:
Hmmlearn的安装
# pip install hmmlearn
Looking in indexes: https://pypi.tuna.tsinghua.edu.cn/simple
Requirement already satisfied: hmmlearn in c:\python3.6\lib\site-packages (0.2.1)
Requirement already satisfied: numpy in c:\python3.6\lib\site-packages (from hmmlearn) (1.16.2+mkl)
Requirement already satisfied: scikit-learn>=0.16 in c:\python3.6\lib\site-packages (from hmmlearn) (0.20.3)
Requirement already satisfied: scipy>=0.13.3 in c:\python3.6\lib\site-packages (from scikit-learn>=0.16->hmmlearn) (1.0.0)
hmmlearn实现了三种HMM模型类,按照观测状态是连续状态还是离散状态,可以分为两类。GaussianHMM和GMMHMM是连续观测状态的HMM模型,而MultinomialHMM是离散观测状态的模型,也是我们在HMM原理系列篇里面使用的模型。
对于MultinomialHMM的模型,使用比较简单,"startprob_"参数对应我们的隐藏状态初始分布Π, "transmat_"对应我们的状态转移矩阵A, "emissionprob_"对应我们的观测状态概率矩阵B。
对于连续观测状态的HMM模型,GaussianHMM类假设观测状态符合高斯分布,而GMMHMM类则假设观测状态符合混合高斯分布。一般情况下我们使用GaussianHMM即高斯分布的观测状态即可。以下对于连续观测状态的HMM模型,我们只讨论GaussianHMM类。
在GaussianHMM类中,"startprob_"参数对应我们的隐藏状态初始分布Π, "transmat_"对应我们的状态转移矩阵A, 比较特殊的是观测状态概率的表示方法,此时由于观测状态是连续值,我们无法像MultinomialHMM一样直接给出矩阵B。而是采用给出各个隐藏状态对应的观测状态高斯分布的概率密度函数的参数。
如果观测序列是一维的,则观测状态的概率密度函数是一维的普通高斯分布。如果观测序列是N维的,则隐藏状态对应的观测状态的概率密度函数是N维高斯分布。高斯分布的概率密度函数参数可以用μ表示高斯分布的期望向量,Σ表示高斯分布的协方差矩阵。在GaussianHMM类中,“means”用来表示各个隐藏状态对应的高斯分布期望向量μ形成的矩阵,而“covars”用来表示各个隐藏状态对应的高斯分布协方差矩阵Σ形成的三维张量。
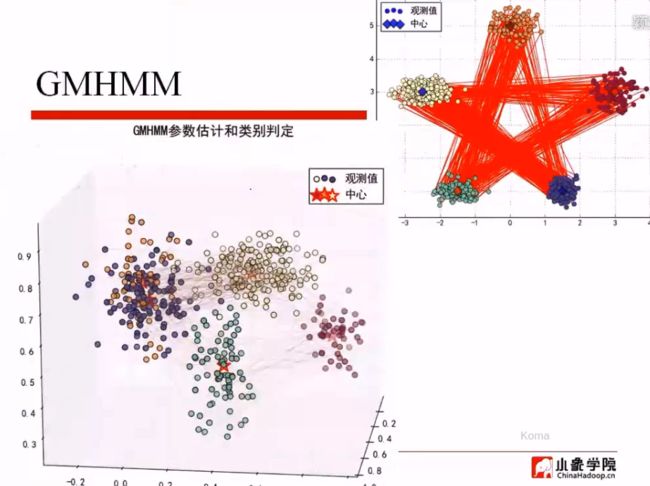
GMHMM
这个例子,给定任何一个样本点,将其作为高斯分布的采样,即直接使用隐马尔科夫模型。从5个高斯分布,随机选出一个高斯分布,并从中选择一个样本来,通过高斯分布,以某种状态转移概率矩阵方式,转移到另外的可能的高斯分布上面去。再在这个高斯分布采取一个样本点,然后继续转移。。。不断做状态转移,以及选择样本点,只把采样的状态样本值取出来,这个即是高斯混合模型的样子。
代码如下:
# !/usr/bin/python
import numpy as np
from hmmlearn import hmm
import matplotlib.pyplot as plt
import matplotlib as mpl
from mpl_toolkits.mplot3d import Axes3D
from matplotlib import cm
from sklearn.metrics.pairwise import pairwise_distances_argmin
import warnings
def expand(a, b):
return 1.05*a-0.05*b, 1.05*b-0.05*a
if __name__ == "__main__":
warnings.filterwarnings("ignore") # hmmlearn(0.2.0) < sklearn(0.18)
np.random.seed(0)
n = 5 # 隐状态数目
n_samples = 500
pi = np.random.rand(n)
pi /= pi.sum()
print('初始概率:', pi)
A = np.random.rand(n, n)
mask = np.zeros((n, n), dtype=np.bool)
mask[0][1] = mask[0][4] = True
mask[1][0] = mask[1][2] = True
mask[2][1] = mask[2][3] = True
mask[3][2] = mask[3][4] = True
mask[4][0] = mask[4][3] = True
A[mask] = 0
for i in range(n):
A[i] /= A[i].sum()
print('转移概率:\n', A)
means = np.array(((30, 30, 30), (0, 50, 20), (-25, 30, 10), (-15, 0, 25), (15, 0, 40)), dtype=np.float)
# means = np.random.rand(5, 3)
print(means)
for i in range(n):
means[i,:] /= np.sqrt(np.sum(means ** 2, axis=1))[i]
print('均值:\n', means)
covars = np.empty((n, 3, 3))
for i in range(n):
# covars[i] = np.diag(np.random.randint(1, 5, size=2))
covars[i] = np.diag(np.random.rand(3)*0.03+0.001) # np.random.rand ∈[0,1)
print('方差:\n', covars)
model = hmm.GaussianHMM(n_components=n, covariance_type='full')
model.startprob_ = pi
model.transmat_ = A
model.means_ = means
model.covars_ = covars
sample, labels = model.sample(n_samples=n_samples, random_state=0)
# 估计参数
model = hmm.GaussianHMM(n_components=n, covariance_type='spherical', n_iter=10)
model.fit(sample)
y = model.predict(sample)
np.set_printoptions(suppress=True)
print('##估计初始概率:', model.startprob_)
print('##估计转移概率:\n', model.transmat_)
print('##估计均值:\n', model.means_)
print('##估计方差:\n', model.covars_)
# 类别
order = pairwise_distances_argmin(means, model.means_, metric='euclidean')
print(order)
pi_hat = model.startprob_[order]
A_hat = model.transmat_[order]
A_hat = A_hat[:, order]
means_hat = model.means_[order]
covars_hat = model.covars_[order]
change = np.empty((n, n_samples), dtype=np.bool)
for i in range(n):
change[i] = y == order[i]
for i in range(n):
y[change[i]] = i
print('估计初始概率:', pi_hat)
print('估计转移概率:\n', A_hat)
print('估计均值:\n', means_hat)
print('估计方差:\n', covars_hat)
print(labels)
print(y)
acc = np.mean(labels == y) * 100
print('准确率:%.2f%%' % acc)
mpl.rcParams['font.sans-serif'] = ['SimHei']
mpl.rcParams['axes.unicode_minus'] = False
fig = plt.figure(figsize=(8, 8), facecolor='w')
ax = fig.add_subplot(111, projection='3d')
colors = plt.cm.Spectral(np.linspace(0,1,n))
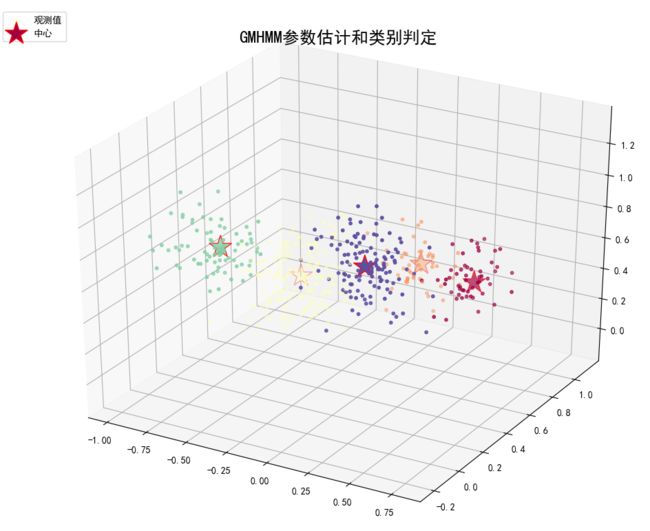
ax.scatter(sample[:, 0], sample[:, 1], sample[:, 2], s=10, c=labels, cmap=plt.cm.Spectral, marker='o', label='观测值', depthshade=True)
# plt.plot(sample[:, 0], sample[:, 1], sample[:, 2], lw=0.4, color='#A07070')
colors = plt.cm.Spectral(np.linspace(0, 1, n))
ax.scatter(means[:, 0], means[:, 1], means[:, 2], s=600, c=colors, edgecolor='r', linewidths=1, marker='*', label='中心')
x_min, y_min, z_min = sample.min(axis=0)
x_max, y_max, z_max = sample.max(axis=0)
x_min, x_max = expand(x_min, x_max)
y_min, y_max = expand(y_min, y_max)
z_min, z_max = expand(z_min, z_max)
ax.set_xlim((x_min, x_max))
ax.set_ylim((y_min, y_max))
ax.set_zlim((z_min, z_max))
plt.legend(loc='upper left')
plt.grid(True)
plt.tight_layout(1)
plt.title('GMHMM参数估计和类别判定', fontsize=18)
plt.show()
问:
问题:π中的M, E概率不是应该为0么,取对数以后应该是一一个很大的负数,老师的pi.txt里面好像不是这样的么?
答:是的,可能代码有bug