功能基因组学方法可以克服阻碍癌症药物开发的局限性,如缺乏确定可靠的靶点以及临床疗效差强人意。在这里,我们对来自30种癌症类型的324个人类癌细胞系进行了基因组规模的CRISPR-Cas9筛选,并开发了一个数据驱动框架,以优先选择癌症治疗方案。我们将细胞适应度效应与基因组生物标志物和药物开发的靶标易用性结合起来,系统地优先考虑已定义的组织和基因型中的新靶标。我们证实了我们最有希望的依赖之一,沃纳综合性ATP依赖解旋酶(Werner syndrome ATP-dependent helicase),作为多个癌症类型与微卫星不稳定的一个关键靶标。我们的分析提供了癌症依赖关系的资源,生成了一个框架来优先考虑癌症药物靶标,并提出了具体的新靶标。本研究中描述的原理可以为药物开发的初始阶段提供信息,为新的、多样化和更有效的癌症药物靶标组合做出贡献
患者肿瘤的分子特征影响临床反应,可用于指导治疗促进更有效的治疗和降低毒性。然而,大多数患者并没有得益于这种靶向治疗,部分原因是对候选靶点的了解有限。在癌症药物的开发中,缺乏疗效是由于90%的损耗率造成的,而且用于新靶点的分子药物依旧有限。有效识别和优先考虑肿瘤靶点的无偏策略可以扩大靶点的范围,提高成功率,并加快新癌症疗法的开发。
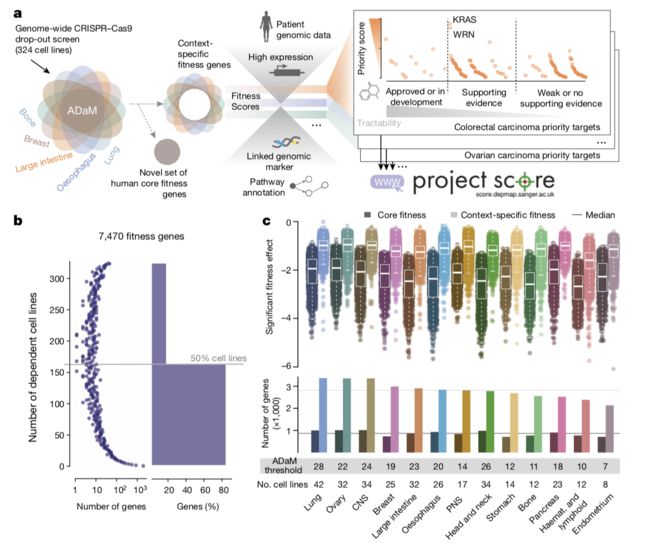
利用sgRNA文库的CRISPR-Cas9筛选已被用于研究基因功能及其在细胞适应性中的作用。基于crispr - cas9的基因组编辑具有很高的特异性,可以生成空等位基因,从而改变外显表型。在这里,我们提出了324个癌细胞系的基因组规模的CRISPR-Cas9适应度筛选和一项综合分析,使候选癌症治疗靶点的优先化成为可能(图1a),我们通过识别沃纳综合征ATP依赖解旋酶(WRN)作为微卫星不稳定性(MSI)肿瘤的靶点来说明这一点。
基因组规模的CRISPR-Cas9在癌细胞系中筛选
为了全面分类肿瘤细胞适配性(全文的适应性都是这个意思:为细胞生长或生存所需的基因),我们对339个癌细胞系进行了941个CRISPR-Cas9适应度筛选,目标基因为18009个。遵循严格的质量控制,最终的分析包括324细胞系来自30个不同的癌症类型,在19个不同的组织。
这些细胞系是高度基因组注释细胞系的细胞模型集合的一部分,广泛代表患者肿瘤的分子特征,包括常见的癌症(如肺癌,结肠癌和乳腺癌)和特定未满足临床需求的癌症( 如肺癌和胰腺癌)。对来自这324个细胞系的筛选数据的分析表明,在分类必需和非必需基因时具有高灵敏度,特异性和精确性,并且结果不受实验因素的偏倚。
定义核心和特定背景的适应度基因
在特定分子或组织学环境中细胞适应性所需的基因(这个就是文中说的背景依赖性核心基因)可能编码有利的药物靶标,因为在健康组织中诱导毒性作用的可能性降低。 相反,大多数测试细胞系常见或在癌症类型中常见的适合度基因(分别称为泛癌或癌症类型特异性核心适应性基因)可能参与细胞的基本过程并具有更大毒性。 因此,重要的是区分特定背景的适应性基因和核心适应性基因。
我们在每个细胞系中鉴定了1,459个适合度基因。 总共有41%(n = 7,470)的所有靶基因在一种或多种细胞系中诱导了适应性效应,并且这些基因的大多数(83%)在不到50%的测试细胞系中诱导了依赖性(图1b)。 为了识别核心适应性基因,我们开发了一种统计方法,即自适应最优模型(ADaM),以自适应地确定基因被分类为核心适合度基因所需的最小数量的依赖细胞系(图1c)。 被定义为13种癌症类型中至少12种(也是适应性确定的)的核心适应性的基因被归类为泛癌核心适应性基因。 这产生了866个癌症类型特异性和553个泛癌症核心适应性基因。
在使用ADaM鉴定的泛癌核心适应性基因中,399先前被定义为必需基因,125是参与必需细胞过程的基因。其余132(24%)个基因是新发现的,并且在细胞管家基因和途径中也显着富集。与先前鉴定的参考核心适合度基因组相比。我们的泛癌核心适应度基因组显示更多基因过程中涉及的基因(中位数= 67%,相对于之前发表的基因组分别为28%和51%),而背景依赖性基因具有相似的假设发现率(FDRs)(取自先前的研究)。血癌细胞系具有最独特的核心适应性基因谱(31个独有的核心适应度基因)。癌症类型特异性核心适应性基因通常在匹配的健康组织中高度表达,与其在基本细胞过程中预测的作用一致,并表明如果用作靶标它们显示出潜在的毒性。值得注意的是,五种基因在单一癌症类型中是核心适应性,并且在匹配的正常组织中基础水平低或不表达,这表明它们可以在这些组织中诱导癌细胞特异性依赖性。
总的来说,使用统计学方法,我们改进和扩展了我们对人类核心适应性基因的现有知识,并鉴定了具有高毒性可能性的基因,因此代表了不太有利的治疗靶点。 此外,由于拥有大规模的数据集,我们现在可以定义背景依赖适应性基因(每个癌症类型的中位数n = 2,813个基因),其中许多具有与核心适应度基因相似或更强的失去适应性效应。
目标优先次序的量化框架
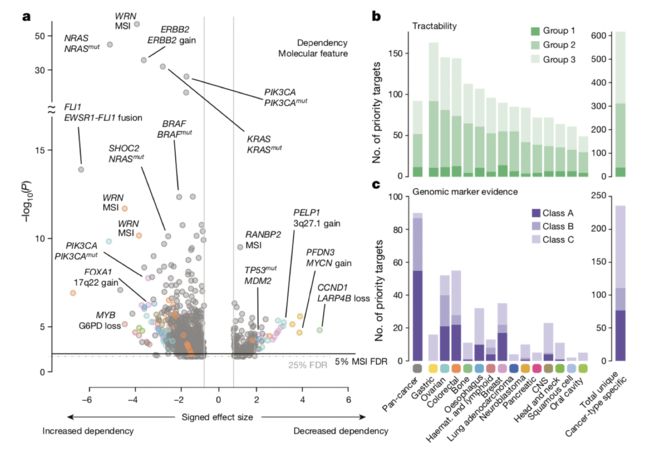
为了从我们的特定背景特定适应度基因列表中提名有希望的治疗目标,我们开发了一个计算框架,它整合了多种证据,为每个基因分配了一个目标优先级分数,范围0-100,并生成了候选的候选列表对于个体癌症类型或泛癌症候选者。为了排除由于潜在毒性而可能是不良靶标的基因,核心适合度基因被评为“0”。对于每个基因,70%的优先级得分来自CRISPR-Cas9实验证据,并根据适应度效应大小,适应性缺陷的显著性,目标基因表达,目标突变状态和其他证据,对依赖细胞系进行均值评估。其余30%的优先级分数是基于与靶标依赖性相关的遗传生物标记物的证据以及靶标在患者肿瘤中被体细胞改变的频率。对于生物标志物分析,我们进行了方差分析(ANOVA)(图2)以测试适合度基因与484种癌症驱动事件(151种单核苷酸变体和333种拷贝数变异体)或MSI之间的关联,在每种癌症类型中都有足够的大样本(n≥10细胞系)。我们基于针对具有批准或临床前癌症化合物的目标计算的得分(扩展数据图5c和补充表5)得出优先级评分阈值(分别为泛癌和癌症类型特异性分析的55和41)。
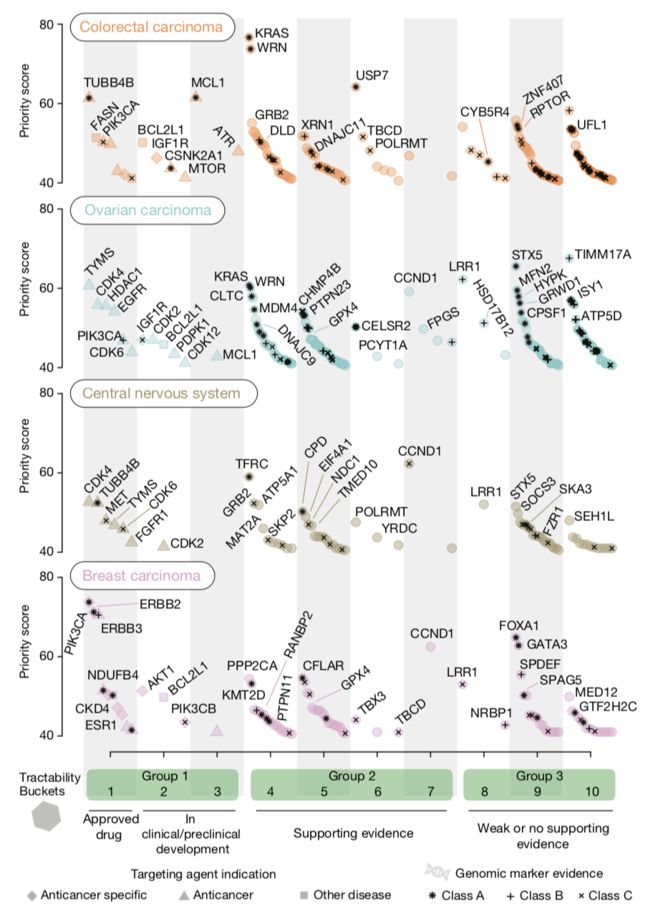
我们共确定了628个独特的优先目标,包括92个泛癌和617个癌症类型特定目标。 在癌症类型中,优先目标的数量变化大约三倍,中位数为88个目标。 大多数癌症类型的优先目标(n = 457,74%)仅在一种(56%)或两种(18%)癌症类型中被识别,强调了它们的背景特异性。 在癌症类型特异性分析中也发现了最优先的泛癌靶点(88%)。 仅在泛癌症分析中确定的11个优先目标通常包括在来自多种癌症类型(例如,CREBBP和JUP)的一小部分细胞系中发生的依赖性或在有限数量的癌症类型中发生的依赖性。 可用细胞系阻止进行癌症类型特异性分析(例如,黑素瘤中的SOX10)。
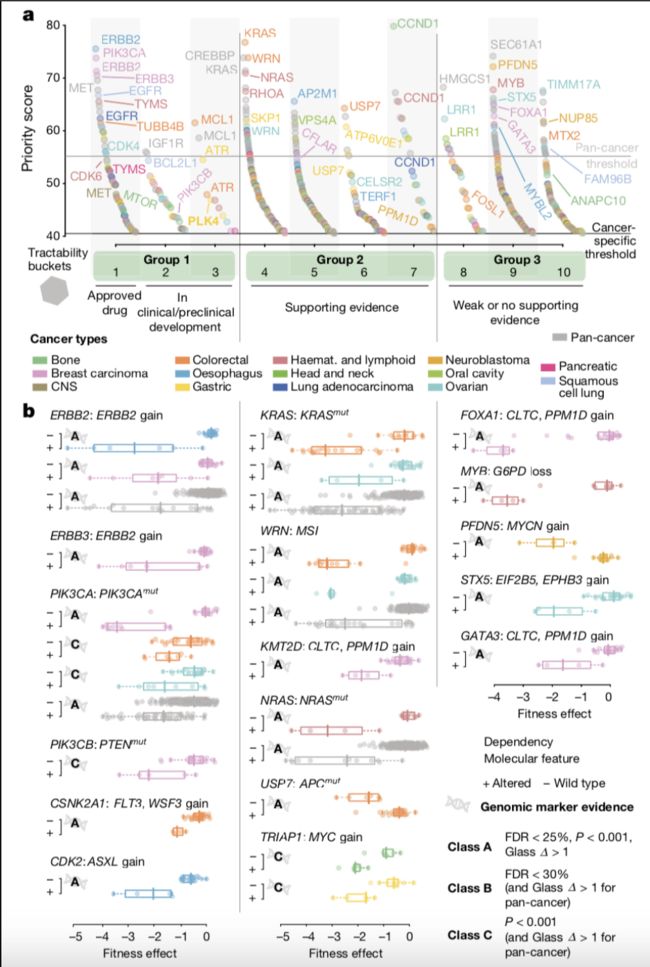
在628个优先目标中,120个(19%)与使用具有高显着性和大效应大小(定义为A类靶标)的ANOVA鉴定的至少一种生物标志物相关,因此这些蛋白质对于药物开发特别有利。 例如,PIK3CA是乳腺癌,食道癌,结肠直肠癌和卵巢癌中的A类靶标; PI3K抑制剂正在临床开发用于PIK3CA13突变的癌症。 使用渐进不那么严格的显着性阈值扩展了目标,其中至少一个生物标志物关联由ANOVA确定,其被定义为B类(n = 61,10%),其次是C类(n = 117,19%)目标,一些 在多种癌症类型中鉴定出来的。 总之,这些结果强调了数据驱动的定量框架通过组合来自多种细胞系的CRISPR-Cas9筛选数据和相关基因组特征来确定靶标优先级的潜力。
优先目标的易用性评估
在目前的药物开发策略的基础上,目标在药物干预的适用性方面各不相同,这为目标选择提供了依据。 使用目标易处理性评估来开发小分子和抗体,我们以前将每个基因分配到10个易处理桶中的1个(1表示最高的易处理性)。 我们将628个优先目标与其可控性交叉引用,并将它们分为三个易处理组。
可追踪性组1(桶1-3)包括临床或临床前开发中批准的抗癌药物或化合物的靶标,并包括40个独特的优先目标,例如乳腺癌中的ERBB2,ERBB3,CDK4,AKT1,ESR1,TYMS和PIK3CB以及PIK3CA。 ,IGF1R,MTOR和ATR在结直肠癌中的表达。在这40个优先目标中,20个具有至少一种针对癌症类型开发的药物,其中靶标被确定为优先,而其余20个靶标具有已经用于或开发用于治疗其他癌症类型的药物,提供重新利用这些药物的机会。第1组中的三分之一优先目标具有A类生物标志物,表明它们是非常理想的目标。一个例子是CSNK2A1,它由结直肠癌细胞系中高度显着的适合度基因CSNK2A1编码,扩增含有FLT3和WASF3的染色体片段(P = 6.65×10-6,玻璃△> 2.9)并被靶向silmasertib。具有标记的第1组中的其他优先目标显示ERBB2扩增时存在ERBB2或ERBB3依赖性,ASXL扩增食管癌细胞系中CDK2依赖性,PIK3CA突变存在时PIK3CA依赖性和PTEN乳腺癌细胞系中PIK3CB依赖性突变。
可追踪性组2(桶4-7)在临床开发中包含277个没有药物的优先目标,但有证据支持目标易处理性。其中,18%具有A类生物标志物,包括KRAS依赖于KRAS突变细胞系,USP7依赖于APC野生型结肠直肠细胞系,KMT2D依赖于乳腺癌细胞系,扩增含有PPM1D和CLTC的染色体片段和MYI扩增的骨和胃癌细胞系中的TRIAP1依赖性。值得注意的是,我们观察到具有MSI和泛癌的结肠直肠和卵巢细胞系中的A类生物标记物依赖于WRN。在第2组中与生物标志物无关的优先目标中,GPX4是多种癌症类型的靶标。对GPX4抑制的敏感性与上皮 - 间充质转变相关,并且我们观察到与GPX4依赖性细胞系中的上皮 - 间质转化相关的标志物的差异表达。这表明我们的目标优先级方案的未来改进如何能够捕获与扩展的分子特征集相关的优先目标,包括基因表达,染色质修饰和分化状态。
最后,第3组(第8-10栏)包括311个优先目标,这些目标没有任何支持或缺乏可以告知可行性的信息; 该组显着富含转录因子。 具有A类生物标志物的组3中的优先目标的实例包括乳腺癌中的FOXA1和GATA3,血液学和淋巴癌中的MYB,卵巢癌中的STX5和神经母细胞瘤细胞系中的PFDN5。
易处理性组1中的优先目标富含蛋白激酶,突出了针对这类目标的药物开发的主要焦点,与第2组和第3组相比,其包括功能更多的多样化靶组。 第2组中的目标最有可能通过常规方式新颖且易于处理,因此代表了药物开发的良好候选者。 较新的治疗方式,如蛋白水解 - 靶向嵌合体,可能会增加适合药物干预的蛋白质的范围,以包括第3组中的目标。总体而言,我们的框架提供了数据驱动的优先治疗目标列表,这些目标将是 癌症药物的发展。
WRN是MSI癌症的目标
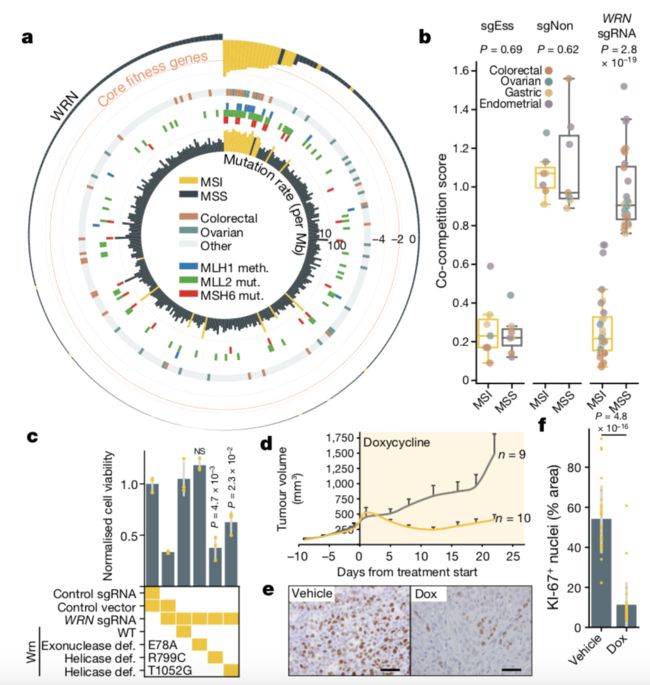
为了证实我们的目标优先级策略,我们研究了WRN解旋酶作为MSI癌症的有希望的靶标。 WRN是五种RecQ家族DNA解旋酶之一,是唯一一种同时具有解旋酶和外切核酸酶结构域,并且在DNA修复,复制,转录和端粒维持中具有不同的作用。 MSI表型是由于MMR途径基因的沉默或失活导致的DNA错配修复(MMR)受损引起的。 MSI与高突变负荷相关,发生在20多种肿瘤类型中,常见于结肠癌,卵巢癌,子宫内膜癌和胃癌(3-28%)。
WRN的依赖性与泛癌ANOVA中的MSI以及结肠癌和卵巢癌细胞系的分析密切相关。 大多数MSI的子宫内膜和胃癌细胞系依赖于WRN; 然而,由于样本量小,与MSI的关联不显着(对于胃)或未进行测试。 MSI在许多其他肿瘤类型中是罕见的(<1%),例如肾,黑素瘤和前列腺癌,并且大多数(测试的5个中的4个)来自这些组织的MSI细胞系不依赖于WRN。 其他经过测试的RecQ家族成员(BLM,RECQL和RECQL5)与MSI细胞系中的适应性基因无关。 对MMR途径基因的非同义突变,启动子甲基化和纯合缺失的集中分析证实了WRN依赖性与MLH1启动子的高甲基化或MSH6突变之间的显着关联; 以及表观遗传调节因子MLL2(也称为KMT2D)的突变。
为了进一步验证WRN,我们进行了基于CRISPR的共竞争测定,其中比较了WRN敲除与野生型细胞的相对适应度。与来自结肠癌,卵巢癌,子宫内膜癌和胃癌的六种MSI细胞系中的野生型细胞相比,使用四种单独sgRNA的WRN敲除降低了WRN敲除的适应性。相比之下,来自这四种组织的所有微卫星稳定细胞系没有差异。一致地,WRN在克隆形成测定中对MSI细胞具有选择性的基础。值得注意的是,WRN敲除对细胞适应性具有有效影响,其效应大小与核心适应度基因相似。此外,我们从系统RNA干扰筛选中挖掘数据并确认MSI癌细胞系中的WRN依赖性,并证实通过RNA干扰的WRN下调强烈地损害了MSI HCT116细胞中的生长,从而在正交实验系统中提供了验证。尽管MMR缺乏与WRN依赖性之间存在很强的相关性,但在微卫星稳定的SW620细胞系中敲除MLH1并未诱导WRN依赖性;相反,HCT116细胞与含有MLH1和/或MSH3的染色体互补(以恢复其表达和纠正MMR缺陷) - 并不能恢复WRN敲除的效果。
为了确定适应性损失效应是否对WRN具有选择性并确定药物靶向的潜在策略,我们使用野生型或亚型Wrn的亚型(对我们使用的WRN sgRNA具有抗性)进行功能性拯救实验 )外切核酸酶(E78A)或解旋酶(R799C或T1052G)结构域中的突变会损害蛋白质功能。 野生型或核酸外切酶缺陷型Wrn的表达拯救了MSN细胞中WRN的敲除,而解旋酶缺陷型Wrn的表达导致无(R799C)或弱(T1052G)拯救。 因此,WRN的解旋酶活性是必需的,并且是可用于治疗靶向的重要结构域。
为了评估MSI细胞对WRN消耗的体内敏感性,我们在HCT116细胞中开发了多西环素诱导型WRN sgRNA系统。 在小鼠中皮下移植表达WRN sgRNA的HCT116细胞后,用强力霉素治疗导致已建立肿瘤的显着生长抑制和增殖细胞数量的减少。 这些发现证实WRN是维持MSI结肠直肠癌细胞体内生长所必需的。
全文总结:
- 作者采用的主要方法是CRISPR-cas9和大规模生物信息学分析;
- 作者进行了迄今为止最大规模的癌症相关基因敲除筛选;
- 定义了不同的分类阈值,全部采用定量的方法进行分类;
- 采用细胞适应度效应与基因组生物标志物和药物开发的靶标易用性相结合的综合性评估绘制了人类癌症靶向图谱;
- 确定了WRN在微卫星干扰类癌症中重要作用,并验证了其作为靶向位点的可行性
参考文献:
Behan F M, Iorio F, Picco G, et al. Prioritization of cancer therapeutic targets using CRISPR–Cas9 screens[J]. Nature, 2019, 568(7753): 511.