一些计算机视觉该有的常识
K-means
已知样本集![]() ,其中每一个观测都是d-维实向量 ,K-means要把这m个样本划分到k个集合中(k ≤ m),使得组内平方和(WCSS)最小。
,其中每一个观测都是d-维实向量 ,K-means要把这m个样本划分到k个集合中(k ≤ m),使得组内平方和(WCSS)最小。
标准算法
最常用的算法使用了迭代优化的技术。它被称为K-means而广为使用,有时也被称为Lloyd算法(尤其在计算机科学领域)。已知初始的k个聚类质心点![]() ,算法按照下面两个步骤交替进行。
,算法按照下面两个步骤交替进行。
- 分配(Assignment):将每个样本i分配到聚类中,使得组内平方和(WCSS)达到最小。
![]()
- 更新(Update):对于上一步得到的每一个聚类,以聚类中样本值的图心,作为新的均值点。
![]()
K-means面对的第一个问题是如何保证收敛,首先可以固定每个类的质心 ![]() ,调整每个样本的所属类别
,调整每个样本的所属类别 ![]() 来让WCSS减小;同样固定
来让WCSS减小;同样固定 ![]() ,调整每个类的质心
,调整每个类的质心 ![]() 也可以让WCSS减小,这两个过程就是内循环中使J单调递减的过程。当WCSS递减到最小时,
也可以让WCSS减小,这两个过程就是内循环中使J单调递减的过程。当WCSS递减到最小时,![]() 和c也同时收敛。(在理论上,可以有多组不同的
和c也同时收敛。(在理论上,可以有多组不同的![]() 和c值能够使得J取得最小值,但这种现象实际上很少见)。
和c值能够使得J取得最小值,但这种现象实际上很少见)。
iris
我们用非常著名的iris数据集。
from sklearn import datasets
from matplotlib import pyplot as plt
import numpy as np
iris = datasets.load_iris()
X, y = iris.data, iris.targetdata = X[:,[1,3]] # 为了便于可视化,只取两个维度
fig, plt.scatter(data[:,0],data[:,1])
plt.show()欧式距离
def distance(p1,p2):
tmp = np.sum((p1-p2)**2)
return np.sqrt(tmp)随机质心
def rand_center(data, k):
"""Generate k center within the range of data set."""
n = data.shape[1]
centroids = np.zeros((k,n)) # k个质心,每个质心的维度和样本维度相同
for i in range(n):
dmin, dmax = np.min(data[:, i]), np.max(data[:, i])
centroids[:, i] = dmin + (dmax - dmin) * np.random.rand(k)
return centroids
centroids = rand_center(data,2)
centroidsarray([[ 2.15198267, 2.42476808],
[ 2.77985426, 0.57839675]])完整代码
kmeans有个缺点,就是可能陷入局部最小值,有改进的方法,比如二分k均值;也可以多计算几次,取效果好的结果。
def kmeans(data,k=2):
def _distance(p1,p2):
tmp = np.sum((p1-p2)**2)
return np.sqrt(tmp)
def _rand_center(data,k):
"""Generate k center within the range of data set."""
n = data.shape[1] # features
centroids = np.zeros((k,n))
for i in range(n):
dmin, dmax = np.min(data[:,i]), np.max(data[:,i])
centroids[:,i] = dmin + (dmax - dmin) * np.random.rand(k)
return centroids
def _converged(centroids1, centroids2):
# if centroids not changed, we say 'converged'
set1 = set([tuple(c) for c in centroids1])
set2 = set([tuple(c) for c in centroids2])
return (set1 == set2)
m = data.shape[0] # number of entries
centroids = _rand_center(data,k)
c = np.zeros(m, dtype=np.int) # track the nearest centroid
assement = np.zeros(m) # for the assement of our model
converged = False
while not converged:
old_centroids = np.copy(centroids)
for i in range(m):
# determine the nearest centroid and track it with label
min_dist, min_index = np.inf, -1
for j in range(k):
dist = _distance(data[i],centroids[j])
if dist < min_dist:
min_dist, min_index = dist, j
c[i] = j
assement[i] = _distance(data[i], centroids[c[i]])**2
# update centroid
for j in range(k):
centroids[j] = np.mean(data[c==j], axis=0)
converged = _converged(old_centroids, centroids)
return centroids, c, np.sum(assement)由于算法可能局部收敛的问题,随机多运行几次,取最优值
best_assement = np.inf
best_centroids = None
best_c = None
for i in range(10):
centroids, c, assement = kmeans(data,2)
if assement < best_assement:
best_assement = assement
best_centroids = centroids
best_c = c
data0 = data[best_c==0]
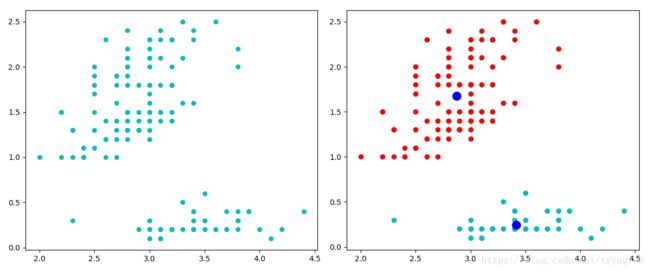
data1 = data[best_c==1]如下图,我们把数据分为两簇:
fig, (ax1,ax2) = plt.subplots(1,2,figsize=(12,5))
ax1.scatter(data[:,0],data[:,1],c='c',s=30,marker='o')
ax2.scatter(data0[:,0],data0[:,1],c='r')
ax2.scatter(data1[:,0],data1[:,1],c='c')
ax2.scatter(centroids[:,0],centroids[:,1],c='b',s=120,marker='o')
plt.show()最大似然估计
最大似然估计是一种确定模型参数值的方法。确定参数值的过程,是找到能最大化模型产生真实观察数据可能性的那一组参数。同时我们将找到的参数值称为最大似然估计(maximum likelihood estimates,MLE)。
最大似然估计总是能精确地得到解吗?
简单来说,不能。更有可能的是,在真实的场景中,对数似然函数的导数仍然是难以解析的(也就是说,很难甚至不可能人工对函数求微分)。因此,一般采用期望最大化(EM)算法等迭代方法为参数估计找到数值解,但总体思路还是一样的。
为什么叫「最大似然(最大可能性)」,而不是「最大概率」呢?
我们先来定义 P(data; μ, σ) 它的意思是「在模型参数μ、σ条件下,观察到数据 data 的概率」。值得注意的是,我们可以将其推广到任意数量的参数和任何分布。
另一方面,L(μ, σ; data) 的意思是「我们在观察到一组数据 data 之后,参数μ、σ取特定的值的似然度。」
上面的公式表示,给定参数后数据的概率等于给定数据后参数的似然度。但是,尽管这两个值是相等的,但是似然度和概率从根本上是提出了两个不同的问题——一个是关于数据的,另一个是关于参数值的。这就是为什么这种方法被称为最大似然法(极大可能性),而不是最大概率。
什么时候最小二乘参数估计和最大似然估计结果相同?
结果表明,当模型被假设为高斯分布时,MLE 的估计等价于最小二乘法。
Kullback–Leibler divergence
相对熵(relative entropy)又称为KL散度(Kullback–Leibler divergence,简称KLD),信息散度(information divergence),信息增益(information gain)。
KL散度是两个概率分布P和Q差别的非对称性的度量。 KL散度是用来 度量使用基于Q的编码来编码来自P的样本平均所需的额外的位元数。 典型情况下,P表示数据的真实分布,Q表示数据的理论分布,模型分布,或P的近似分布。
定义
对于离散随机变量,其概率分布P和Q的KL散度可按下式定义为
对于连续随机变量,其概率分布P和Q可按积分方式定义为
流形(manifolds)
流形(英语:Manifolds),是局部具有欧几里得空间性质的空间,是欧几里得空间中的曲线、曲面等概念的推广。欧几里得空间就是最简单的流形的实例。地球表面这样的球面则是一个稍微复杂的例子。一般的流形可以通过把许多平直的片折弯并粘连而成。
任何一个流形都可以嵌入到足够高维度的欧氏空间中 (Whitney嵌入定理)。
流形的特殊性质: