Matrix的用法详解
稀释矩阵顾名思义是非零值分布较少的矩阵,处理稀释矩阵一般用到三个包,Matrix,slam and glmnet包,前两个用于存储与处理稀释矩阵,后者是用于稀释矩阵的广义线性模型的分析。
Matrix 包
Doug Bates 的Matrix包能够非常方便地将矩阵在全矩阵和稀释矩阵间转换
举个例子,用系统自带的matrix函数生成1000*1000的数值为零的矩阵,和用Matrix函数生成同样的矩阵,如下所示:
| 1 2 3 4 5 6 7 8 9 |
library('Matrix')
m1 <- matrix(0, nrow =1000, ncol =1000) m2 <- Matrix(0, nrow =1000, ncol =1000, sparse = TRUE)
object.size(m1) # 8000200 bytes object.size(m2) # 5632 bytes |
你能看到,matrix生成的矩阵占有的内存(RAM)比Matrix高1500倍以上,但是假如两个矩阵用的是非零矩阵,效果会如何呢?
| 1 2 3 4 5 6 7 |
m1[500,500]<-1 m2[500,500]<-1
object.size(m1) # 8000200 bytes object.size(m2) # 5648 bytes |
全矩阵并未改变内存值,因为所有零值一开始就已经被展示出来了,而稀释矩阵在非零值增加后,其内存值就发生了改变。测试发现,稀释矩阵增加一个非零值,其内存增加16BYTES,因此将所有零值替换为非零值,稀释矩阵将增加5632+16*1000*1000BYTES,即16005632BYTES,差不多是用matrix函数创建的矩阵所占内存的两倍。
因此,假如你的矩阵是稀释的,那么用Matrix会相当高效,而非稀释的话,则相对低效。
除了初始化和指定任务作业上,我们也能用Matrix包做很多其它操作,如向量相乘、相加、相减和转换:
| 1 2 3 4 |
m2 %*% rnorm(1000) m2 + m2 m2 - m2 t(m2) |
同时,也能用cBind和rBind进行合并工作;
| 1 2 3 4 5 6 7 |
m3 <- cBind(m2, m2) nrow(m3) ncol(m3)
m4 <- rBind(m2, m2) nrow(m4) ncol(m4) |
slam 包
Slam包是由Kurt Hornik设计的,使用这个包生成的稀释矩阵远比用Matrix包要小的多。
比如,生成一个上述第一个这样的稀释矩阵只要1032BYTES:
| 1 2 3 4 5 6 7 8 9 |
library('slam')
m1 <- matrix(0, nrow =1000, ncol =1000) m2 <- simple_triplet_zero_matrix(nrow =1000, ncol =1000)
object.size(m1) # 8000200 bytes object.size(m2) # 1032 bytes |
当然,我们也要看看每增加一个非零值,内存增量是多少(结果可以自己运算比较一下):
| 1 2 3 4 5 6 |
m1[500,500]<-1 m2[500,500]<-1
object.size(m1) object.size(m2) |
最后,所有对矩阵的运算方式和Matrix一样:
| 1 2 3 4 |
m2 %*% rnorm(1000) m2 + m2 m2 - m2 t(m2) |
glmnet 包
当然,以上所有处理的目的是用于最终分析的,不然就无降低内存的意义;GLM是分析该数据的一种通用性较广的方法,多数时候用线性和逻辑回归就足以,当然需要加上部分规则以优化模型。
Glmnet包可被用于处理稀释矩阵而无需转换成如数据框的形式:
| 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 |
library('Matrix') library('glmnet')
n <-10000 p <-500
x <- matrix(rnorm(n * p), n, p) iz <- sample(1:(n * p), size = n * p *0.85, replace = FALSE) x[iz]<-0
object.size(x)
sx <- Matrix(x, sparse = TRUE)
object.size(sx)
beta <- rnorm(p)
y <- x %*% beta + rnorm(n)
glmnet.fit<- glmnet(x, y) |
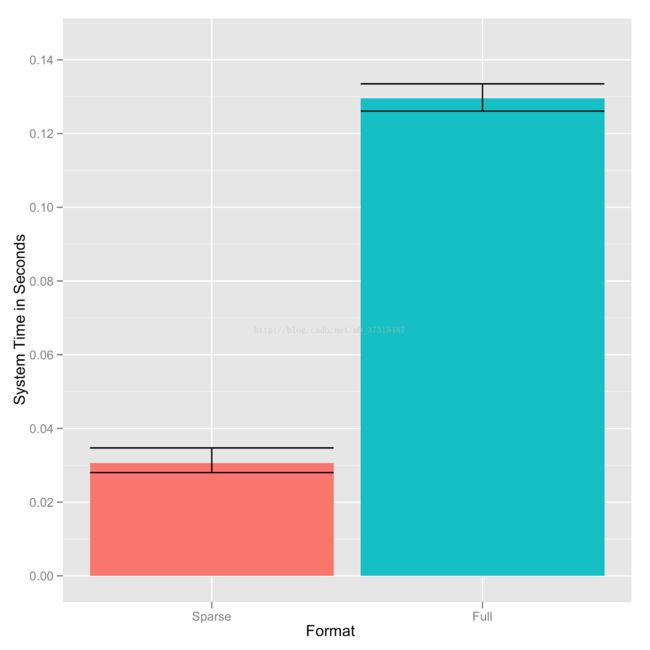
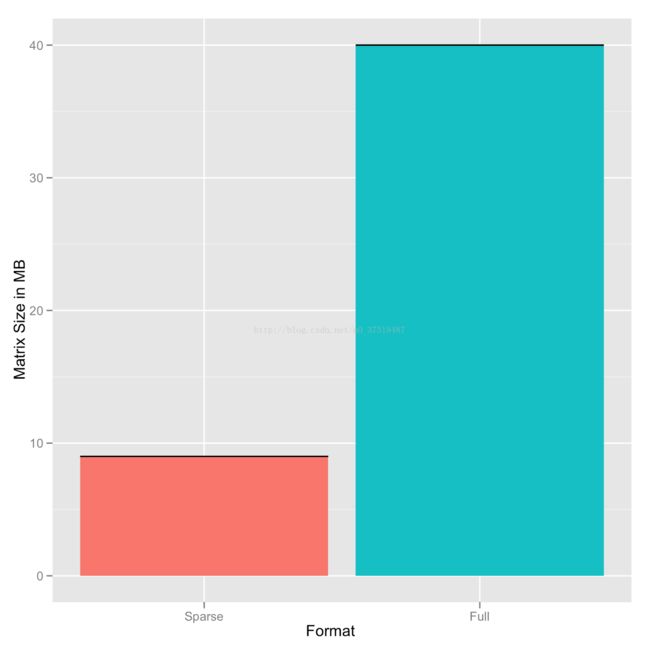
使用稀释矩阵有多高效?下面用GLMNET包对稀释矩阵和全矩阵的处理速度作了对比
| 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 |
library('Matrix') library('glmnet')
set.seed(1) performance <- data.frame()
for(sim in 1:10) { n <-10000 p <-500
nzc <- trunc(p /10)
x <- matrix(rnorm(n * p), n, p) iz <- sample(1:(n * p), size = n * p *0.85, replace = FALSE) x[iz]<-0 sx <- Matrix(x, sparse = TRUE)
beta <- rnorm(nzc) fx <- x[, seq(nzc)]%*% beta
eps <- rnorm(n) y <- fx + eps
sparse.times<- system.time(fit1 <- glmnet(sx, y)) full.times<- system.time(fit2 <- glmnet(x, y)) sparse.size<- as.numeric(object.size(sx)) full.size<- as.numeric(object.size(x))
performance <- rbind(performance, data.frame(Format ='Sparse', UserTime = sparse.times[1], SystemTime = sparse.times[2], ElapsedTime = sparse.times[3], Size = sparse.size)) performance <- rbind(performance, data.frame(Format ='Full', UserTime = full.times[1], SystemTime = full.times[2], ElapsedTime = full.times[3], Size = full.size)) }
ggplot(performance, aes(x = Format, y = UserTime, fill = Format))+ stat_summary(fun.data='mean_cl_boot', geom ='bar')+ stat_summary(fun.data='mean_cl_boot', geom ='errorbar')+ ylab('User Time in Seconds')+ opts(legend.position='none') ggsave('sparse_vs_full_user_time.pdf')
ggplot(performance, aes(x = Format, y = SystemTime, fill = Format))+ stat_summary(fun.data='mean_cl_boot', geom ='bar')+ stat_summary(fun.data='mean_cl_boot', geom ='errorbar')+ ylab('System Time in Seconds')+ opts(legend.position='none') ggsave('sparse_vs_full_system_time.pdf')
ggplot(performance, aes(x = Format, y = ElapsedTime, fill = Format))+ stat_summary(fun.data='mean_cl_boot', geom ='bar')+ stat_summary(fun.data='mean_cl_boot', geom ='errorbar')+ ylab('Elapsed Time in Seconds')+ opts(legend.position='none') ggsave('sparse_vs_full_elapsed_time.pdf')
ggplot(performance, aes(x = Format, y = Size /1000000, fill = Format))+ stat_summary(fun.data='mean_cl_boot', geom ='bar')+ stat_summary(fun.data='mean_cl_boot', geom ='errorbar')+ ylab('Matrix Size in MB')+ opts(legend.position='none') ggsave('sparse_vs_full_memory.pdf') |
以下是结果图:
########################################################################
欢迎微信交流:左码为作者微信,右码为作者公众号(博客文章会同步于公众号发布)
![]()
![]()