制作自己的COCO数据集,训练mask R-CNN
先占个坑,国庆假期的时候来写。
10月16号,终于打开了CSDN,想起了还有一篇没写的博客
本文适合一些小白看,比较简单,稍微改一下别人的示例代码就行。
之前做了点肿瘤检测的工作,就是在图上把肿瘤的位置用边界框标出来,用自己的数据做成了VOC格式,然后训练了faster R-CNN,结果还行。下面要做的工作是在之前的基础上,还要把肿瘤勾画出来,所以就要用到mask R-CNN了,这时候数据就成了问题。用VOC的公开数据集训练mask R-CNN的时候就不太顺利,更不用说训练自己的数据了。于是直接转战COCO格式的数据,毕竟R-CNN一系列的很多模型都是用COCO数据集预训练的。
那么问题来了,该怎么制作自己的COCO数据集,看了很多博客,都没看懂,千辛万苦下,终于找到了一个有用的教程!!
参考https://patrickwasp.com/create-your-own-coco-style-dataset/
可以下载pycococreator-master这个python工具包。
这里展示了一个关于形状数据集的例子,是一个三分类的数据集,分别为三角,圆形,正方形。需要准备的就是原图和对应的mask,如图所示: shapes_train2018放的是原图,这里有两张图,annotations存放的就是对应的mask。
1001.jpeg里面有四个实例,所以它对应的mask就有四幅图。1001.jpeg和对应的mask如下所示:
然后图像的命名也有要求,1001.jpeg的mask的文件名中必须有1001,后面的circle,square就是类别的名字。准备好后,代码会根据你的命名来寻找对应图像的标签文件。
准备好自己的数据以及对应的标签后,就要运行工具包中的 shapes_to_coco.py 文件了
#!/usr/bin/env python3
import datetime
import json
import os
import re
import fnmatch
from PIL import Image
import numpy as np
from pycococreatortools import pycococreatortools
# 这里设置一些文件路径
ROOT_DIR = 'train' # 根目录
IMAGE_DIR = os.path.join(ROOT_DIR, "shapes_train2018") # 根目录下存放你原图的文件夹
ANNOTATION_DIR = os.path.join(ROOT_DIR, "annotations") # 根目录下存放mask标签的文件夹
# 这里就是填一些有关你数据集的信息
INFO = {
"description": "Example Dataset",
"url": "https://github.com/waspinator/pycococreator",
"version": "0.1.0",
"year": 2018,
"contributor": "waspinator",
"date_created": datetime.datetime.utcnow().isoformat(' ')
}
LICENSES = [
{
"id": 1,
"name": "Attribution-NonCommercial-ShareAlike License",
"url": "http://creativecommons.org/licenses/by-nc-sa/2.0/"
}
]
# 这里是你数据集的类别,这里有三个分类,就是square, circle, triangle。制作自己的数据集主要改这里就行了
CATEGORIES = [
{
'id': 1,
'name': 'square',
'supercategory': 'shape',
},
{
'id': 2,
'name': 'circle',
'supercategory': 'shape',
},
{
'id': 3,
'name': 'triangle',
'supercategory': 'shape',
},
]
def filter_for_jpeg(root, files):
file_types = ['*.jpeg', '*.jpg']
file_types = r'|'.join([fnmatch.translate(x) for x in file_types])
files = [os.path.join(root, f) for f in files]
files = [f for f in files if re.match(file_types, f)]
return files
def filter_for_annotations(root, files, image_filename):
file_types = ['*.png']
file_types = r'|'.join([fnmatch.translate(x) for x in file_types])
basename_no_extension = os.path.splitext(os.path.basename(image_filename))[0]
file_name_prefix = basename_no_extension + '.*'
files = [os.path.join(root, f) for f in files]
files = [f for f in files if re.match(file_types, f)]
files = [f for f in files if re.match(file_name_prefix, os.path.splitext(os.path.basename(f))[0])]
return files
def main():
coco_output = {
"info": INFO,
"licenses": LICENSES,
"categories": CATEGORIES,
"images": [],
"annotations": []
}
image_id = 1
segmentation_id = 1
# filter for jpeg images
for root, _, files in os.walk(IMAGE_DIR):
image_files = filter_for_jpeg(root, files)
# go through each image
for image_filename in image_files:
image = Image.open(image_filename)
image_info = pycococreatortools.create_image_info(
image_id, os.path.basename(image_filename), image.size)
coco_output["images"].append(image_info)
# filter for associated png annotations
for root, _, files in os.walk(ANNOTATION_DIR):
annotation_files = filter_for_annotations(root, files, image_filename)
# go through each associated annotation
for annotation_filename in annotation_files:
print(annotation_filename)
class_id = [x['id'] for x in CATEGORIES if x['name'] in annotation_filename][0]
category_info = {'id': class_id, 'is_crowd': 'crowd' in image_filename}
binary_mask = np.asarray(Image.open(annotation_filename)
.convert('1')).astype(np.uint8)
annotation_info = pycococreatortools.create_annotation_info(
segmentation_id, image_id, category_info, binary_mask,
image.size, tolerance=2)
if annotation_info is not None:
coco_output["annotations"].append(annotation_info)
segmentation_id = segmentation_id + 1
image_id = image_id + 1
with open('{}/instances_shape_train2018.json'.format(ROOT_DIR), 'w') as output_json_file:
json.dump(coco_output, output_json_file)
if __name__ == "__main__":
main()
最终生成的训练数据的json文件就是上面代码中的instances_shape_train2018.json,制作自己的数据集的时候,可以先准备一个空的.json文件放在那,程序执行完,就能把你的标注信息都写进去了。
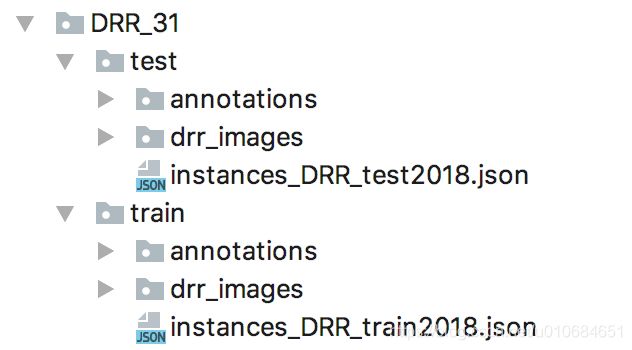
然后我也展示一下自己的数据吧。文件结构如图:
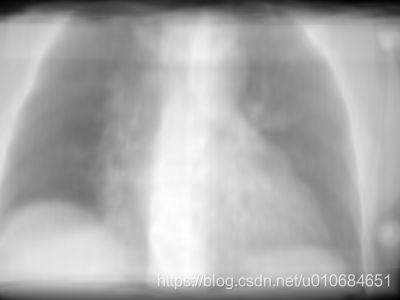
训练数据和测试数据都生成了json文件。我的原图是81-1.png,mask文件是81-1tumor.png,因为我的图只有tumor一个类别,并且每一幅图中都只有一个tumor,所以对应的mask文件也只有一个。
下面是我的原图和标签图,左边是肺部的图,右边是肿瘤的mask。
看到生成的json文件里这些密密麻麻的坐标有木有很开心!mask R-CNN终于可以训练起来了。
另外:我也刚开始用医学图像做目标检测相关的工作,把dcm转成png做的,有做相关方向的同学可以一起讨论。