R语言样条曲线,泊松回归对患者准化死亡率 (SMR)分析
泊松建模标准化发病率或死亡率(SIR/SMR),即计算标准化率的间接方法。 SIR 是观察到的和预期的案例的比率。 预期病例数是通过将特定阶层的人口率乘以队列中相应的人年得出的。
我们继续使用我们的女性直肠癌数据(公众号回复:SMR2可以获得数据)
首先我们导入R包和数据
library(popEpi)
library(Epi)
library(splines)
bc<-read.csv("E:/r/test/smr2.csv",sep=',',header=TRUE)
导入数据后,我们来看看数据的构成,sex表示性别,bi_date为出生日期,dg_date为诊断日期,ex_date为退出日期,其实就是截止日期,status为是否死亡,我们的结局变量,dg_age为诊断时的年龄,这个指标其实是多余的,可以通过诊断日期减去出生日期得出来。

这个数据记录1993-2012年女性直肠癌的死亡数据,假如我们想了解每个年龄段患者的死亡情况。我们先对status这个变量进行处理,让它变成0和1两种状态
bc$status<-ifelse(bc$status==0,0,1)
把关于时间的数据进行格式转换
bc$ex_date<-as.Date(bc$ex_date)
bc$dg_date<-as.Date(bc$dg_date)
bc$bi_date<-as.Date(bc$bi_date)
处理好以后我们使用popEpi包的lexpand函数生成一个一个列联表,这个函数我来解释一下,status,birth,exit,entry这几个参数很好理解,都是一些状态和时间参数,breaks这个参数是对数据进行一个隔断,生成一个表,我们取1993年到2013年,然后年龄取1-100岁之间,fot这个是随访时间,我们这里选开始(0年),10年,20年3个时间点
c<- lexpand(bc, status = status, birth = bi_date, exit = ex_date, entry = dg_date,
breaks = list(per = 1993:2013, age = 1:100, fot = c(0,10,20,Inf)),
aggre = list(fot, agegroup = age, year = per, sex) )
下图显示删除了16条信息,这些是已进入就退出组的。
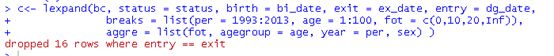
下图为生成的表格
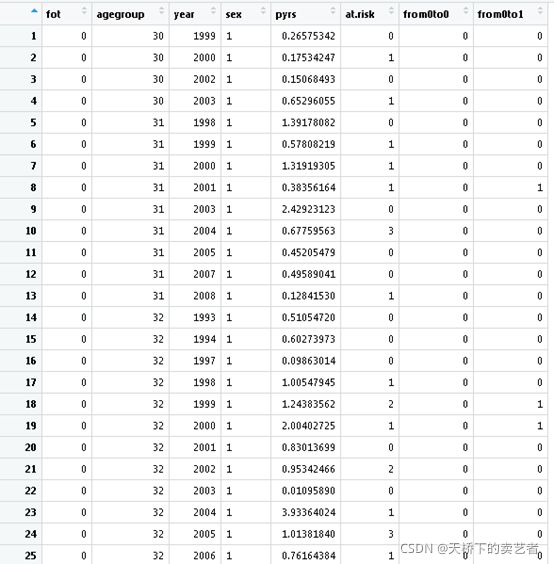
表格很清楚的展示了各项数据fot只是展示0和10年两个时间点,为什么没有20呢?因为1993年到2013年没有20年,所以20这个节点的数据没有。agegroup是按我们之前的设置通过每一年进行分组,pyrs为人年, from0to0为生存人数,from0to1为死亡人数。
得出数据表后我们就可以用过sir函数得出SMR,sir函数需要一个参考的人年死亡率,这里以popEpi包的popmort数据集的haz指标做参考。
se <- sir( coh.data = c, coh.obs = 'from0to1', coh.pyrs = 'pyrs',
ref.data = popmort, ref.rate = 'haz',
adjust = c('agegroup','year','sex'), print ='fot')

总的SMR和0年,10年的SMR都求出来了,还可以进一步作图表示
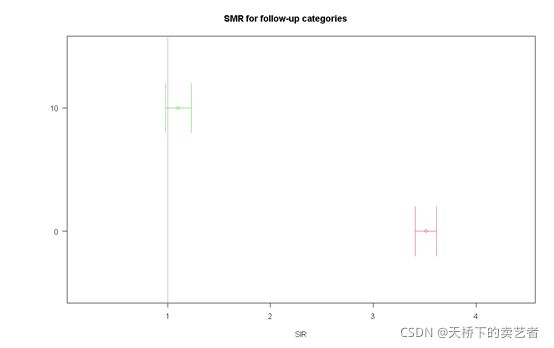
由图形可以看出10年的SMR明显低于开始(0年的)。
假设我们想查看每一个年的数据情况,我们可以设置为,
c1<- lexpand( bc, status = status, birth = bi_date, exit = ex_date, entry = dg_date,
breaks = list(per = 1950:2013, age = 1:100, fot = 0:50),
aggre = list(fot, agegroup = age, year = per, sex) )
sf1 <- sirspline( coh.data = c1, coh.obs = 'from0to1', coh.pyrs = 'pyrs',
ref.data = popmort, ref.rate = 'haz',
adjust = c('agegroup','year','sex'),
spline = c('fot'), dependent.splines=FALSE)
plot(sf1, col=2, log=TRUE)

可以看出来,随着时间延长SMR不断降低,年龄也是连续变量我们也可以查看年龄和SMR关系
sf2 <- sirspline( coh.data = c1, coh.obs = 'from0to1', coh.pyrs = 'pyrs',
ref.data = popmort, ref.rate = 'haz',
adjust = c('agegroup','year','sex'),
spline = c('agegroup','fot'), dependent.splines=FALSE)
plot(sf1, col=2, log=TRUE)
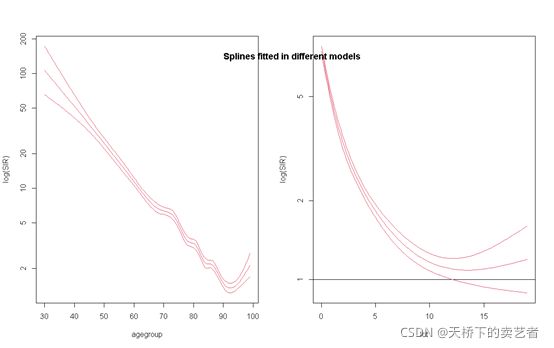
可以发现随着时间和年龄增长,SMR下降,这和我们刚开始选取0年和10年的节点的结论是一样的。
我们现在选取的时间节点(也就是参考点)是以fot=0为基础的,我们还可以换其他的参考点,假设我们想以2012年前后作为参考点
c$year.cat <- ifelse(c$year < 2002, 1, 2)
sy <- sirspline( coh.data = c1, coh.obs = 'from0to1', coh.pyrs = 'pyrs',
ref.data = popmort, ref.rate = 'haz',
adjust = c('agegroup','year','sex'),
spline = c('agegroup'), print = 'year.cat')
得出数据sy后,查看一下数据

我们把spline.est.A这个数据表提出来
be<-sy$spline.est.A
be$x<-sy$spline.seq.A
be$i<-factor(be$i)
names(be)
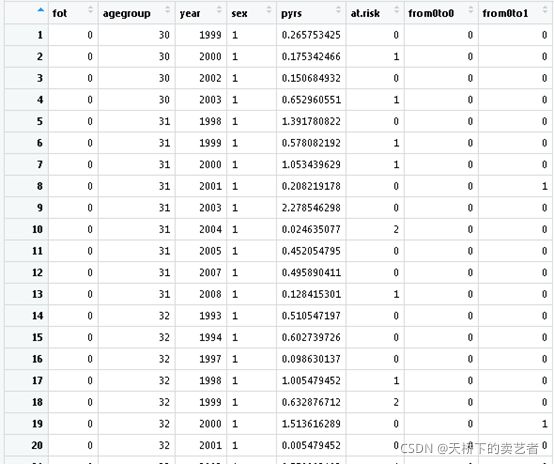
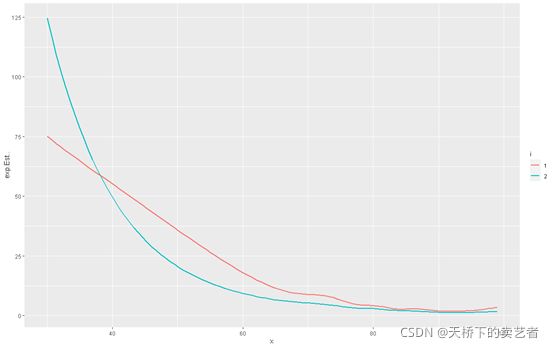
ggplot(be,aes(x=x,y=exp.Est..))+
geom_line(aes(col=i),size=1)
ggplot(be,aes(x=x,y=exp.Est..))+
geom_ribbon(aes(ymin=X2.5.,ymax=X97.5.,fill=i),alpha=0.2)+
geom_line(aes(col=i),size=1)
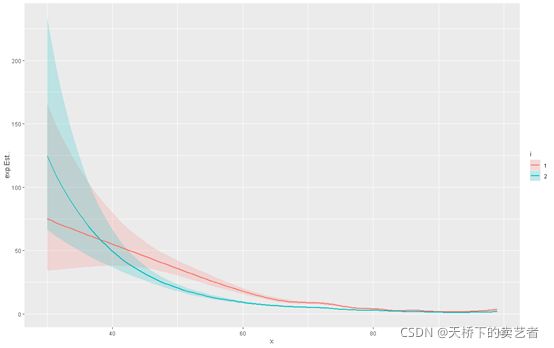
print(sy)

对于 2002 年之前的类别,50 岁之后的 SMR 似乎更高。此外,p 值 (<0.0001) 表明 2002 年之前和之后的年龄组趋势存在差异。P 值是似然比检验比较样条拟合在一起和单独拟合的模型。