【菜菜的sklearn课堂笔记】逻辑回归与评分卡-逻辑回归中的特征工程
视频作者:菜菜TsaiTsai
链接:【技术干货】菜菜的机器学习sklearn【全85集】Python进阶_哔哩哔哩_bilibili
当特征的数量很多的时候,我们出于业务考虑,也出于计算量的考虑,希望对逻辑回归进行特征选择来降维。比如,在判断一个人是否会患乳腺癌的时候,医生如果看5~8个指标来确诊,会比需要看30个指标来确诊容易得多。
业务选择
说到降维和特征选择,首先要想到的是利用自己的业务能力进行选择,肉眼可见明显和标签有关的特征就是需要留下的。或者,可以让算法先帮助我们筛选过一遍特征,然后在少量的特征中,我们再根据业务常识来选择更少量的特征。
PCA和SVD一般不用
说到降维,我们首先想到的是之前提过的高效降维算法,PCA和SVD,遗憾的是,这两种方法大多数时候不适用于逻辑回归。
逻辑回归是由线性回归演变而来,线性回归的一个核心目的是通过求解参数来探究特征X与标签y之间的关系,而逻辑回归也传承了这个性质,我们常常希望通过逻辑回归的结果,来判断什么样的特征与分类结果相关,因此我们希望保留特征的原貌。PCA和SVD的降维结果是不可解释的,因此一旦降维后,我们就无法解释特征和标签之间的关系了。
当然,在不需要探究特征与标签之间关系的线性数据上,降维算法PCA和SVD也是可以使用的。
统计方法可以使用,但不是非常必要
既然降维算法不能使用,我们要用的就是特征选择方法。
逻辑回归对数据的要求低于线性回归,由于我们不是使用最小二乘法来求解,所以逻辑回归对数据的总体分布和方差没有要求,也不需要排除特征之间的共线性,但如果我们确实希望使用一些统计方法,比如方差,卡方,互信息等方法来做特征选择,也并没有问题。过滤法中所有的方法,都可以用在逻辑回归上。
在一些博客中有这样的观点:多重共线性会影响线性模型的效果。对于线性回归来说,多重共线性会影响比较大,所以我们需要使用方差过滤和方差膨胀因子VIF(variance inflation factor)来消除共线性。但是对于逻辑回归,其实不是非常必要,甚至有时候,我们还需要多一些相互关联的特征来增强模型的表现。当然,如果我们无法通过其他方式提升模型表现,并且你感觉到模型中的共线性影响了模型效果,那懂得统计学的你可以试试看用VIF消除共线性的方法,遗憾的是现在sklearn中并没有提供VIF的功能(新版好像有了)。
R vs Python,统计学 vs 机器学习
统计学的思路是一种“先验”的思路,不管做什么都要先”检验“,先”满足条件“,事后也要各种”检验“,以确保各种数学假设被满足,不然的话,理论上就无法得出好结果。而机器学习是一种”后验“的思路,不管三七二十一,我先让模型跑一跑,效果不好我再想办法,如果模型效果好,我完全不在意什么共线性,残差不满足正态分布,没有哑变量之类的细节,模型效果好大过天!
高效的嵌入法Embedded
我感觉这里视频的代码是有点问题的,所以部分代码有少许区别,执行结果也因此不同
但是更有效的方法,毫无疑问会是我们的embedded嵌入法。我们已经说明了,由于L1正则化会使得部分特征对应的参数为0,因此L1正则化可以用来做特征选择,结合嵌入法的模块SelectFromModel,我们可以很容易就筛选出让模型十分高效的特征。
注意,此时我们的目的是,尽量保留原数据上的信息,让模型在降维后的数据上的拟合效果保持优秀,因此我们不考虑训练集测试集的问题,把所有的数据都放入模型进行降维。
也就是说我们是把特征选择后的 X e m b e d d e d X_{embedded} Xembedded代入用 X , y X,y X,y建出来的逻辑回归模型中,然后看 X e m b e d d e d , y X_{embedded},y Xembedded,y交叉验证的分数
from sklearn.linear_model import LogisticRegression as LR
from sklearn.datasets import load_breast_cancer
import numpy as np
import matplotlib.pyplot as plt
from sklearn.model_selection import cross_val_score
from sklearn.feature_selection import SelectFromModel
data = load_breast_cancer()
X = data.data
y = data.target
LR_ = LR(solver='liblinear',penalty='l1',max_iter=1000,C=0.8,random_state=420)
# C是根据上一节学习曲线选择的最佳参数值
cross_val_score(LR_,X,y,cv=10).mean()
---
0.9508998790078644
X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(X,y)
# norm_order=1:使用L1范式,模型会删除在L1范式下不重要的特征
# 由于使用了L1范式,因此我们可以不使用threshold,即默认为None,那么此时不重要的特征就是coef_中值为0对应的特征
说一下我不太明白视频里的一个地方
SelectFromModel(LR(solver='liblinear',penalty='l2',max_iter=1000,C=0.8,random_state=420),norm_order=1) # 这里我们建模和特征选择的时候使用不同的范数 # 其实我不太理解为什么这么做,个人感觉还是二者同一比较好吧,但也确实不会报错 # 也就是下面的penalty='l2'但norm_order=1 --- SelectFromModel(estimator=LogisticRegression(C=0.8, class_weight=None, dual=False, fit_intercept=True, intercept_scaling=1, max_iter=1000, multi_class='warn', n_jobs=None, penalty='l2', random_state=420, solver='liblinear', tol=0.0001, verbose=0, warm_start=False), max_features=None, norm_order=1, prefit=False, threshold=None)
X_embedded.shape
---
(569, 10)
(LR_.fit(X,y).coef_ != 0).sum() # 非零的特征有十个,符合我们上面说的norm_order=1的意义
---
10
cross_val_score(LR_,X_embedded,y,cv=10).mean()
---
0.9368323826808401
看看结果,特征数量被减小到个位数,并且模型的效果却没有下降太多,如果我们要求不高,在这里其实就可以停下了。
但是,能否让模型的拟合效果更好呢?在这里,我们有两种调整方式:
threshold
调节SelectFromModel这个类中的参数threshold,这是嵌入法的阈值,表示删除所有参数的绝对值低于这个阈值的特征。现在threshold默认为None,选择了所有L1正则化后参数不为0的特征。我们此时,只要调整threshold的值(画出threshold的学习曲线),就可以观察不同的threshold下模型的效果如何变化。一旦调整threshold,就不是在使用L1正则化选择特征,而是使用模型的属性.coef_中生成的各个特征的系数来选择(同样的我们指定norm_order=2也是用threshold来选择特征,但没太看懂norm_order=2的时候阈值默认是啥,反正肯定不是0,而且此时coef_有正有负,用的绝对值来衡量重要性)。
coef_虽然返回的是特征的系数,但是系数的大小和决策树中的feature_ importances_以及降维算法中的可解释性方差explained_vairance_概念相似,其实都是衡量特征的重要程度和贡献度的,因此SelectFromModel中的参数threshold可以设置为coef_的阈值,即可以剔除系数小于threshold中输入的数字的所有特征。
fullx = []
fsx = []
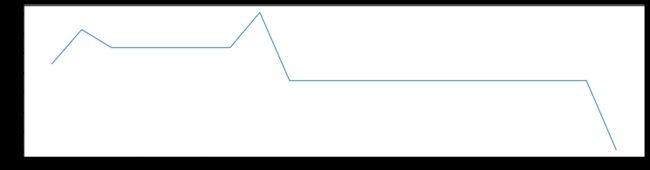
threshold = np.linspace(0,abs(LR_.fit(X,y).coef_).max(),20)
k = 0
for i in threshold:
X_embedded = SelectFromModel(LR_,threshold=i).fit_transform(X,y)
# 这里的threshold就是对于L1范数下coef_返回的列表进行选择,因为默认norm_order=1,上面我们建模的时候LR_也用的L1范数
fullx.append(cross_val_score(LR_,X,y,cv=5).mean())
fsx.append(cross_val_score(LR_,X_embedded,y,cv=5).mean())
print((threshold[k],X_embedded.shape[1]))
print((abs(LR_.fit(X,y).coef_).ravel() >= threshold[k]).sum())
# 如果执行上面这一行,我们可以发现
# (abs(LR_.fit(X,y).coef_).ravel() > threshold[k]).sum() == X_embedded.shape[1]
# 也就是说,如果嵌入法选择特征的时候不指定norm_order,指定threshold
# 那么选择出来的特征是根据coef_的绝对值是否大于threshold来决定的(feature_importances未验证)
# 但如果threshold过大,甚至大于最大的特征对应的coef_,SelectFromModel则会报错
k = k + 1
---
(0.0, 30)
30
(0.23220654667112106, 4)
4
(0.4644130933422421, 3)
3
……
(3.947511293409058, 1)
1
(4.179717840080179, 1)
1
(4.4119243867513, 1)
1
plt.figure(figsize=(20,5))
plt.plot(threshold,fullx,label="full")
plt.plot(threshold,fsx,label='feature selection')
plt.xticks(threshold)
plt.legend()
plt.show()
我们可以进一步细化,因为从30个特征到4个特征太快了
fullx = []
fsx = []
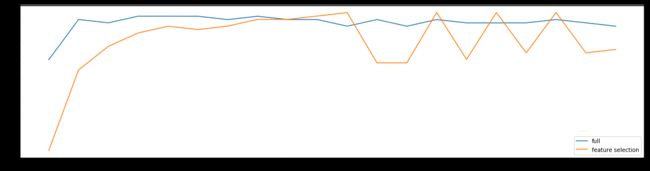
threshold = np.linspace(0,0.23220654667112106,20)
k = 0
for i in threshold:
X_embedded = SelectFromModel(LR_,threshold=i).fit_transform(X,y)
# fullx.append(cross_val_score(LR_,X,y,cv=5).mean())
fsx.append(cross_val_score(LR_,X_embedded,y,cv=5).mean())
print((threshold[k],X_embedded.shape[1]))
k = k + 1
plt.figure(figsize=(20,5))
plt.plot(threshold,fsx,label='feature selection')
plt.xticks(threshold)
plt.show()
# ConvergenceWarning: Liblinear failed to converge, increase the number of iterations.
# 迭代次数不够导致的,调节max_iter即可
---
(0.0, 30)
(0.012221397193216898, 10)
(0.024442794386433795, 8)
(0.036664191579650694, 8)
(0.04888558877286759, 8)
……
(0.20776375228468727, 5)
(0.21998514947790415, 5)
(0.23220654667112106, 4)
这里我们发现依旧从30个特征直接掉到10个特征,但是要注意
(LR_.fit(X,y).coef_ != 0).sum()
---
10
也就是说,只要threshold存在,就是最多10个特征,因此曲线已经够细化了
这里如果我们一直用的是L2范数(用L2范数建模,用L2范数来筛选特征)
嵌入法其实是比较无效的,大家可以用学习曲线来跑一跑:当threshold越来越大,被删除的特征越来越多,模型的效果也越来越差,模型效果最好的情况下需要保证有17个以上的特征。实际上我画了细化的学习曲线,如果要保证模型的效果比降维前更好,我们需要保留25个特征,这对于现实情况来说,是一种无效的降维:需要30个指标来判断病情,和需要25个指标来判断病情,对医生来说区别不大。
调整逻辑回归建模时的C
fullx = []
fsx = []
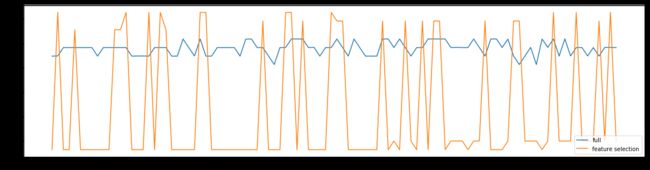
C = np.arange(0.01,10.01,0.5)
# 换L2范数了,因为我们之前说过一般先用L2范数来建模,上面不用是因为效果不好,也就是引用中说的话
for i in C:
LR_ = LR(solver='liblinear',C=i,random_state=420)
X_embedded = SelectFromModel(LR_,norm_order=2).fit_transform(X,y)
fullx.append(cross_val_score(LR_,X,y,cv=10).mean())
fsx.append(cross_val_score(LR_,X_embedded,y,cv=10).mean())
print(max(fsx),C[fsx.index(max(fsx))])
---
0.9545620516809263 6.51
plt.figure(figsize=(20,5))
plt.plot(C,fullx,label='full')
plt.plot(C,fsx,label='feature selection')
plt.xticks(C)
plt.legend()
plt.show()
继续细化学习曲线:
fullx = []
fsx = []
C = np.arange(6.01,7.01,0.01)
for i in C:
LR_ = LR(solver='liblinear',C=i,random_state=420)
X_embedded = SelectFromModel(LR_,norm_order=2).fit_transform(X,y)
fullx.append(cross_val_score(LR_,X,y,cv=10).mean())
fsx.append(cross_val_score(LR_,X_embedded,y,cv=10).mean())
print(max(fsx),C[fsx.index(max(fsx))])
---
0.9563164376458386 6.02
plt.figure(figsize=(20,5))
plt.plot(C,fullx,label='full')
plt.plot(C,fsx,label='feature selection')
plt.legend(loc=4)
plt.show()
这里要注意,我们现在得到的C是为了筛选特征而选出的C,之前C=0.8是在不筛选特征的情况下最佳的C值,二者含义是不同的
验证一下模型
LR_ = LR(solver='liblinear',C=6.02,random_state=420)
cross_val_score(LR_,X,y,cv=10).mean()
---
0.9473911070780398
X_embedded = SelectFromModel(LR_,norm_order=2).fit_transform(X,y)
cross_val_score(LR_,X_embedded,y,cv=10).mean()
---
0.9563164376458386
# 也就是上面的print(max(fsx),C[fsx.index(max(fsx))])的结果
X_embedded.shape
---
(569, 10)
这样我们就实现了在特征选择的前提下,保持模型拟合的高效,现在,如果有一位医生可以来为我们指点迷津,看看剩下的这些特征中,有哪些是对针对病情来说特别重要的,也许我们还可以继续降维。
当然,除了嵌入法,系数累加法或者包装法也是可以使用的。
上面我们使用的嵌入法都是先执行然后知道有多少个特征剩余,但是包装法可以指定我们需要多少个特征,这是一个优于嵌入法的方面
比较麻烦的系数累加法
系数累加法的原理非常简单。在PCA中,我们通过绘制累积可解释方差贡献率曲线来选择超参数,在逻辑回归中我们可以使用系数coef_来这样做,并且我们选择特征个数的逻辑也是类似的:找出曲线由锐利变平滑的转折点,转折点之前被累加的特征都是我们需要的,转折点之后的我们都不需要。
不过这种方法相对比较麻烦,因为我们要先对特征系数进行从大到小的排序,还要确保我们知道排序后的每个系数对应的原始特征的位置,才能够正确找出那些重要的特征。如果要使用这样的方法,不如直接使用嵌入法来得方便。
突然想起来自己之前关于sort和sorted的使用问题,正好记录一下
- sort是应用在list上的方法,而sorted可以对所有可迭代的对象进行排序操作
- sort是对原有列表进行操作,而sorted返回的是一个新的可迭代对象,不会改变原来的对象
- sort使用方法为list.sort(), 而sorted的使用方法为sorted(list)
作者:蛋炒饭之神
链接:python中sort()与sorted()的区别_蛋炒饭之神的博客-CSDN博客_python sort和sorted的区别a = [4,3,2,1] a.sort() # 没有任何返回值 a # 直接改变a --- [1, 2, 3, 4] b = [4,2,3,1] sorted(b) --- [1, 2, 3, 4] sorted(b,reverse=True) # 两种方法都能reverse降序排列 --- [4, 3, 2, 1] import numpy as np import pandas as pd sorted(np.array(b)) sorted(pd.Series(b,index=[7,8,6,5])) --- [1, 2, 3, 4] sorted(pd.DataFrame(np.random.randint(0,10,(4,5)),columns=[1,3,2,5,4],index=[3,2,4,1])) # 对于DataFrame来说排的是columns,没啥意义了 --- [1, 2, 3, 4, 5]
简单快速的包装法
相对的,包装法可以直接设定我们需要的特征个数,逻辑回归在现实中运用时,可能会有”需要5~8个变量”这种需求,包装法此时就非常方便了。不过逻辑回归的包装法的使用和其他算法一样,并不具有特别之处,因此在这里就不在赘述。