英特尔oneAPI—开发生物序列聚类工具
摘要
本文介绍了基于oneAPI平台开发的生物序列聚类应用,文章结构如下。
应用背景:这里介绍聚类应用nGIA,介绍了为什么开发这个应用,以及它解决了什么问题。这部分非生信领域的同学可以跳过。
如何入门oneAPI:结合我自己的经历,介绍了一条从零开始学习oneAPI的路径。
CUDA代码移植与原生代码开发:结合开发应用的经验,介绍了从CUDA移植到oneAPI时候踩的一些坑,以及为什么推荐写原生的代码。
oneAPI的跨平台特性:用实际的应用,把oneAPI代码跑在nvidia的GPU上。展示了oneAPI的跨平台特性,介绍了开发跨平台代码的时候需要避免哪些问题。
应用背景
首先介绍一下生物序列聚类。序列聚类也就是序列去冗余工作,比较著名的应用有CD-HIT,Uclust,Linclust等。相关领域的同学应该都知道,聚类是生信领域基础且重要的工具。那些被广泛使用的聚类工具,谷歌学术上可以看到,文章的引用都是过千甚至过万的,这也显示了聚类工具的重要和普遍。
目前的聚类工具得到的都是近似结果。Holm[1]在1998年提出了一种贪婪增量的聚类方法,这是可以产生金标准的方法。但是Holm的方法计算量太大,以至于运行时间长到无法接受,因此研究者们提出了各种改进算法,通过牺牲精度的方法换取计算速度的提升。我实际测试发现,速度越快的聚类工具,聚类结果的精度越低。
nGIA是一个能够进行准确聚类,且速度足够快的聚类工具集。Holm的方法能够得到聚类金标准,受限于计算量巨大而无法实现,随着GPU的发展目前算力已经足够充足了。其他聚类工具都是基于CPU平台的,所以可用算力很少。nGIA是基因超算平台的,利用GPU加速,通过MPI支持多节点,因此可用算力比其他应用高出几个数量级。通过充分利用超算的巨大算力,nGIA可以得到精确的聚类结果,且运行速度很快。同时nGIA也有可以运行在普通电脑上的单节点版本,利用GPU进行加速,即使用普通电脑依然比其他工具运行速度更快。nGIA支持蛋白序列和基因序列数据集的聚类,支持单节点和多节点,支持cuda和oneAPI。
具体算法可以参考相关论文,具体实现可以参考代码。
[1]Holm L, Sander C. Removing near-neighbour redundancy from large protein sequence collections[J]. Bioinformatics (Oxford, England), 1998, 14(5): 423-429.
如何入门oneAPI
这部分我想结合我自己的经历,推荐给大家一条从零开始学习oneAPI的路径。最初我是听了oneAPI的线上讲座,开始对oneAPI产生兴趣。之后开始自己写代码,然后遇到问题就从零零碎碎的渠道一点一点学习。回头来看,我走了一些弯路,初始的学习也是不成体系的。现在整理了一下我走过的路,然后按照循序渐进的顺序推荐给大家,希望有帮助。
1. 首先关于oneAPI的介绍,我觉得有一篇博客写的很好,如果是想了解oneAPI的同学可以参考一下。其中的代码部分看不懂可以不用看,后面会进行系统的学习。之后可以申请一个英特尔的云账号,方便后面学习和测试。
2. 看过介绍之后想来大家都会写hello world了,但是这显然是不够的,想更多了解一些oneAPI的知识,可以做一个编程游戏。通过以上学习,应该可以写一些简单的代码了,可以在英特尔的云上测试自己的代码。
3. 初期大家最需要解决的工作很可能不是开发新代码,而是把已有的cuda代码移植到oneAPI平台。这部分工作比较简单,可以参考intel的官方文档。需要注意的是工具自动移植后的代码通常是有bug的,不过经过步骤2的学习,这些bug大家都能修复。
4. 这时候就可以关注更深入的内容,比如程序运行背后的机制,如何提升代码的性能等等。可以参考Data Parallel C++。这本书很厚,但是不用通读,遇到问题的时候当作手册进行查询就好了。
CUDA代码移植与原生代码开发
到这里相信大家都已经比较熟悉oneAPI了,就不写太细节的内容了。结合我开发nGIA的过程,聊一聊踩的一些坑。
nGIA最初的版本是用CUDA开发的,接触了oneAPI之后,就把cuda代码用工具移植到oneAPI了。工具是比较好用的,但是需要注意以下两点:
1. 移植后的代码会为每个函数生成一个队列,严重影响性能。接下来举个例子:
__global__ void kernel_sayHello() {
printf("hello\n");
}
void sayHello1() {
kernel_sayHello<<<1,1>>>();
cudaDeviceSynchronize();
}
void sayHello2() {
kernel_sayHello<<<1,1>>>();
cudaDeviceSynchronize();
}
int main() {
sayHello1();
sayHello2();
}以上是cuda的代码,核函数输出hello。两个子函数分别调用一次和函数,主函数调用两个子函数。
#include
#include
void kernel_sayHello(const sycl::stream &stream_ct1) {
stream_ct1 << "hello\n";
}
void sayHello1() {
dpct::get_default_queue().submit([&](sycl::handler &cgh) {
sycl::stream stream_ct1(64 * 1024, 80, cgh);
cgh.parallel_for(
sycl::nd_range<3>(sycl::range<3>(1, 1, 1), sycl::range<3>(1, 1, 1)),
[=](sycl::nd_item<3> item_ct1) {
kernel_sayHello(stream_ct1);
});
});
dpct::get_current_device().queues_wait_and_throw();
}
void sayHello2() {
dpct::get_default_queue().submit([&](sycl::handler &cgh) {
sycl::stream stream_ct1(64 * 1024, 80, cgh);
cgh.parallel_for(
sycl::nd_range<3>(sycl::range<3>(1, 1, 1), sycl::range<3>(1, 1, 1)),
[=](sycl::nd_item<3> item_ct1) {
kernel_sayHello(stream_ct1);
});
});
dpct::get_current_device().queues_wait_and_throw();
}
int main() {
sayHello1();
sayHello2();
} 以上是移植以后的oneAPI代码。这段代码看起来没有问题,实际也可以编译运行,但是性能会很慢。因为每个子函数都重新申请了一个队列,这个操作的开销很大。可以声明一个全局的队列,然后每次执行和函数都调用这个队列。更改后的代码如下:
#include
sycl::queue queue; // 全局队列
void kernel_sayHello(const sycl::stream &stream_ct1) {
stream_ct1 << "hello\n";
}
void sayHello1() {
queue.submit([&](sycl::handler &cgh) {
sycl::stream stream_ct1(64 * 1024, 80, cgh);
cgh.parallel_for(sycl::nd_range<3>(sycl::range<3>(1, 1, 1),
sycl::range<3>(1, 1, 1)), [=](sycl::nd_item<3> item_ct1) {
kernel_sayHello(stream_ct1);
});
});
queue.wait();
}
void sayHello2() {
queue.submit([&](sycl::handler &cgh) {
sycl::stream stream_ct1(64 * 1024, 80, cgh);
cgh.parallel_for(sycl::nd_range<3>(sycl::range<3>(1, 1, 1),
sycl::range<3>(1, 1, 1)), [=](sycl::nd_item<3> item_ct1) {
kernel_sayHello(stream_ct1);
});
});
queue.wait();
}
int main() {
sycl::default_selector selector;
queue = sycl::queue(selector); // 生成队列
sayHello1();
sayHello2();
} 新代码定义了全局的队列变量,然后只初始化一次,之后每个子函数调用都是用这个队列,更改以后性能会提升很多。
2. 在oneAPI中,传入核函数的结构体只能包含基础数据结构。举个例子:
#include
#include
struct Data {
int a;
int b;
int *c;
// std::string name; // 这里会导致结构体无法拷贝到设备,取消注释后编译报错
};
void kernel_add(int a, int b, int *c) {
*c = a+b;
}
int main() {
sycl::default_selector selector;
sycl::queue queue = sycl::queue(selector);
Data data;
data.a = 1;
data.b = 2;
data.c = sycl::malloc_shared(1, queue);
queue.submit([&](sycl::handler &cgh) {
cgh.parallel_for(sycl::nd_range<3>(sycl::range<3>(1, 1, 1),
sycl::range<3>(1, 1, 1)), [=](sycl::nd_item<3> item) {
kernel_add(data.a, data.b, data.c);
});
});
queue.wait();
std::cout << *data.c << std::endl;
} 上面的代码在结构体中生命了一个字符串,这会导致编译报错,如果去掉就可以正常编译运行。但是cuda代码中,类似的结构体是可以给核函数传参数的,并不会报错,因此cuda代码中很可能有类似的结构。这样代码移植到oneAPI以后编译报错很难排查,需要注意。
接下来聊聊为什么我推荐大家不要移植,而是直接用oneAPI进行开发。随着对oneAPI的熟悉,加上我买了搭载intel独显的笔记本电脑,后期的nGIA都是直接用oneAPI进行开发。为了提升代码在Nvidia平台的效率,再把oneAPI代码手工翻译成cuda代码。虽然手工移植代码的工作量不大,但是依然比用cuda开发,再移植到oneAPI更麻烦,我为什么非要用oneAPI开发呢?
促使我直接用oneAPI进行开发的主要原因是oneAPI代码的逻辑更简单。大家可以看到oneAPI的代码其实是不分宿主机代码和设备代码的,都是C++代码,在代码开发的时候看起来更统一。而且如果想偷懒的话,只要能接受大概20%的性能损失,其实可以在不改动代码的前提下,直接把oneAPI的代码跑到Nvidia显卡上的,连移植都不需要。接下来就要介绍oneAPI的跨平台特性。
oneAPI的跨平台特性
oneAPI的代码是可以跨平台执行的。oneAPI的一大特性,就是可以用一套代码运行在不同平台上。相信大家都知道oneAPI的代码有通用性,可以运行在Nvidia平台上,但是对代码到底有多通用,以及代码的性等方面能都有好奇。接下来我会用nGIA做一个实验,在一个Nvidia显卡上分别运行cuda版和oneAPI版的nGIA。证明oneAPI的跨平台特性,以及对比他们的性能。
| CPU | GPU | OS |
| Intel G4560 | Nvidia GTX950M | Ubuntu 18.04 |
首先按照codeplay官网的教程安装编译器,之后下载nGIA的代码。下载的代码中自带两个数据集,分别是基因序列数据与蛋白序列数据,都在data目录下,这里用基因序列作为例子。
1. 解压基因数据集current_NCBI_gg16S_unaligned,之后编译数据库生成工具makeDB,然后生成数据库。
2. 编译聚类工具的代码。由于是单机环境,因此分别编译cuda与OneAPI文件夹下的SignalNode版本的工具。这两个工具的算法是完全一致的,所采用的编程技巧也完全相同,只是实现在不同的平台上,因此可以作为最直接的对比。
3. 分别用两个工具进行聚类,用同样的数据库作为输入,并且设置同样的运行参数。
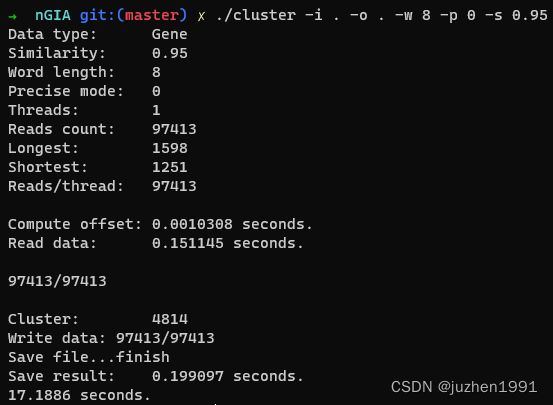
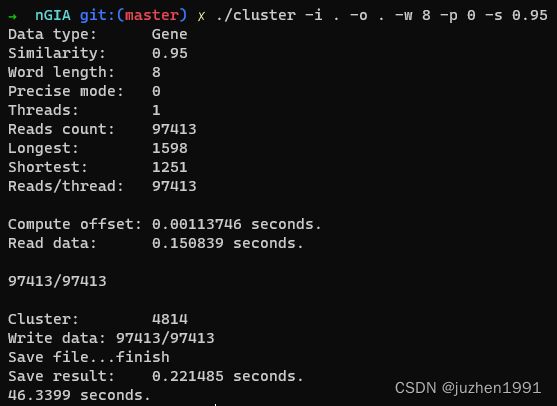
得到的结果如下:
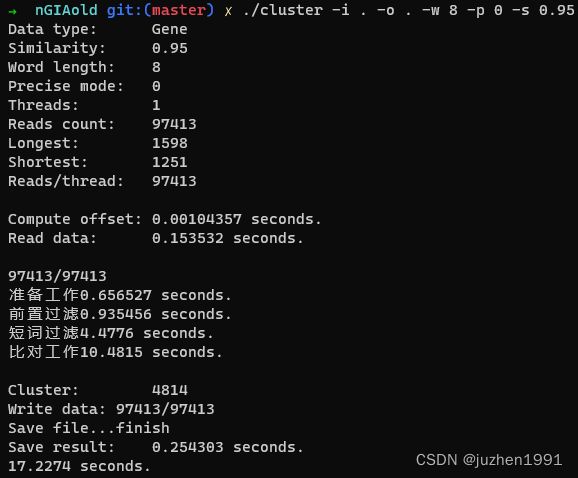
上图中第一张是cuda版工具的运行结果,第二张是oneAPI版工具的运行结果。可以看到cuda版工具耗时17秒左右,oneAPI耗时46秒左右。看起来性能差距很大,关于这一点后面会进行详细的讨论。
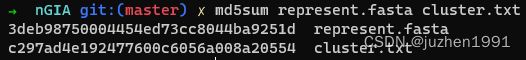
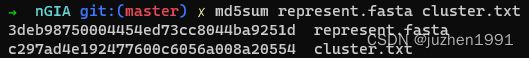
目前先不考虑性能,首先验证一下程序的正确性。对两个工具生成的结果进行一下md5校验,结果如下:
上图中,第一张是cuda版的结果,第二张是oneAPI版的结果,可以看到校验值完全一致,也就是oneAPI代码跨平台的功能是没问题的。做实验的时候,这里是超出我预期的,因为oneAPI的代码一个字母都没改,也就是说,运行在Nvidia显卡上的代码,完全就是运行在Intel显卡上的代码。我以为多少会出现一些兼容问题,但实际证明oneAPI的跨平台特性非常靠谱!
接下来详细分析应用的性能问题。我在代码中添加了对聚类应用中的主要函数的分别计时间,以找到oneAPI版本代码的性能问题根源。结果如下:
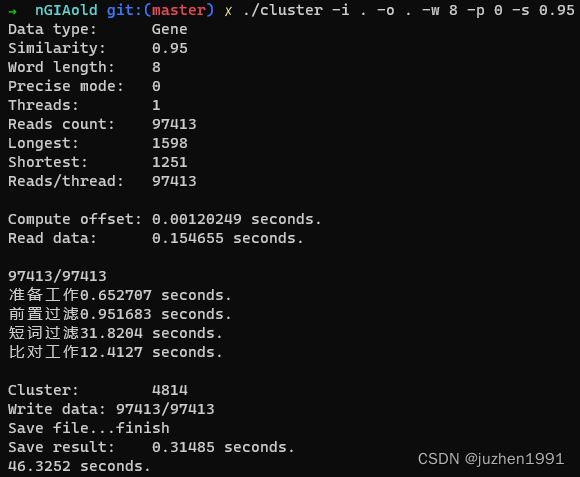
上图中,第一张是cuda版的结果,第二张是oneAPI版的结果。聚类中主要的函数只有四个,分别是准备工作,前置过滤,短词过滤,比对工作。可以看到cuda版的短词过滤部分耗时4秒左右,而oneAPI版耗时超过了31秒,其他函数则差距不大,因此定位到问题出在短词过滤函数。
这里需要对短词过滤函数进行分析,找到瓶颈原因。由于代码是我自己写的,因此可以直接给出结论,原因是短词过滤函数中声明了使用共享内存。我个人推测,由于不同硬件的共享内存大小不同,为了保证代码能正确运行,在编译的时候自动将共享内存替换为了全局显存,因此造成性能大幅下降。
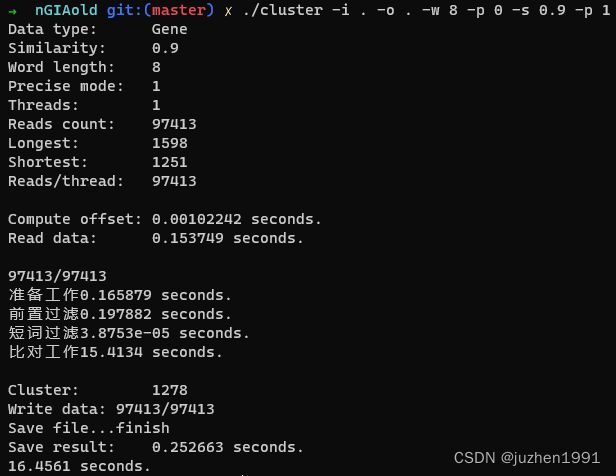
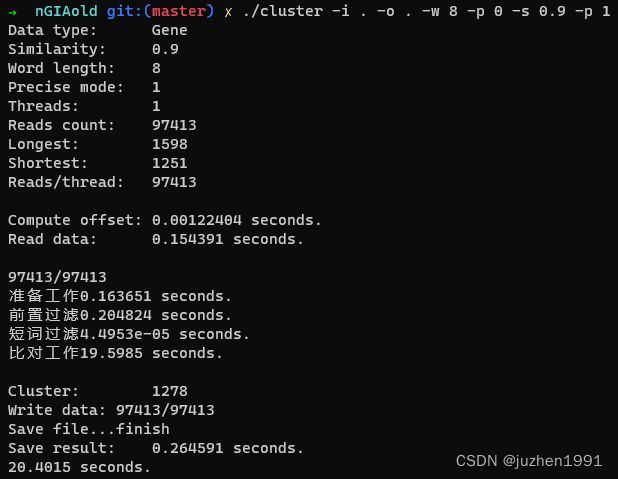
短词过滤的步骤是可以去掉的。聚类工具有准确模式,在准确模式下,会忽略短词比对步骤。去掉短词比对之后就可以得到oneAPI与cuda版代码的真实性能对比,结果如下图所示:
上图中,第一张是cuda的结果,第二张是oneAPI的结果。可以看到cuda版耗时16.46秒,oneAPI版耗时20.40秒。oneAPI版效率可以达到cuda版效率的80%,这是在代码一个字母都不改的前提下达到的。公开课上曾经透露过,优化后oneAPI代码运行在Nvidia显卡上,性能可以达到cuda版代码的95%,现在看应该是真的。另外据我所知,oneAPI代码到AMD的ROCm平台的适配工作也在进行,oneAPI真正实现了跨硬件平台。
总结这一部分内容。
1. oneAPI代码可以在没有任何改动的前提下,运行在包括Nvidia显卡和Intel显卡上,且功能正确,速度可以达到原生代码的80%。
2. 对于跨平台的代码,要慎用共享内存,把共享内存当作一级缓存是更合适的做法。
以上就是我开发nGIA过程中得到的一些关于oneAPI的收获,分享出来,希望对大家有帮助。