Keras实现Unet语义分割医学细胞图像并训练自己的数据集
文章目录
- 一、Unet网络模型
- 二、代码运行
- 三、制作自己的数据集进行训练并测试标注
# 前言 本文实现keras下的Unet语义分割模型并且用自己制作的数据集进行训练并预测。
本文引用了一些博文里面的内容,侵权请联系删改
引用的一些文章链接地址:
1.Unet网络讲解https://blog.csdn.net/weixin_44791964/article/details/108864955
2.U-net源码讲解(Keras)https://blog.csdn.net/mieleizhi0522/article/details/82217677
也有一些相关的博文可以参考
1.https://zhuanlan.zhihu.com/p/57859749
2.unet代码试运行
https://blog.csdn.net/py_yangh/article/details/82726786
3.https://www.bilibili.com/video/BV1v7411Z7b9?from=search&seid=14522545615848347758&spm_id_from=333.337.0.0
4.https://blog.csdn.net/weixin_41036461/article/details/109129810utm_term=unet%E7%9A%84%E6%8D%9F%E5%A4%B1%E5%87%BD%E6%95%B0&utm_medium=distribute.pc_aggpage_search_result.none-task-blog-2allsobaiduweb~default-5-109129810&spm=3001.4430
##Github代码下载地址:
FENGShuanglang/unet https://github.com/FENGShuanglang/unet
##环境
python3.6.8
tensorflow-gpu1.13.1
keras2.1.5
没有硬性要求,tensorflow2.x以下的应该都可以运行
一、Unet网络模型
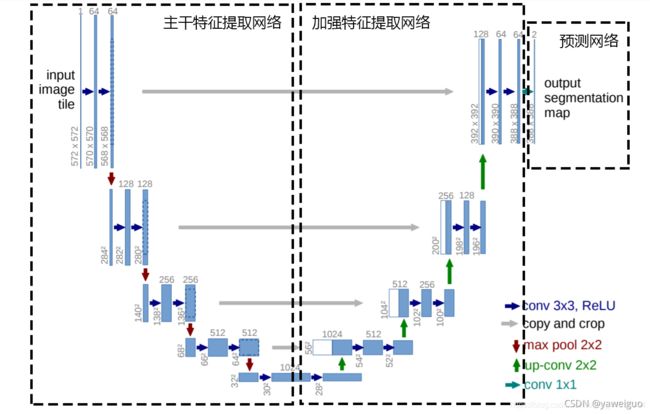
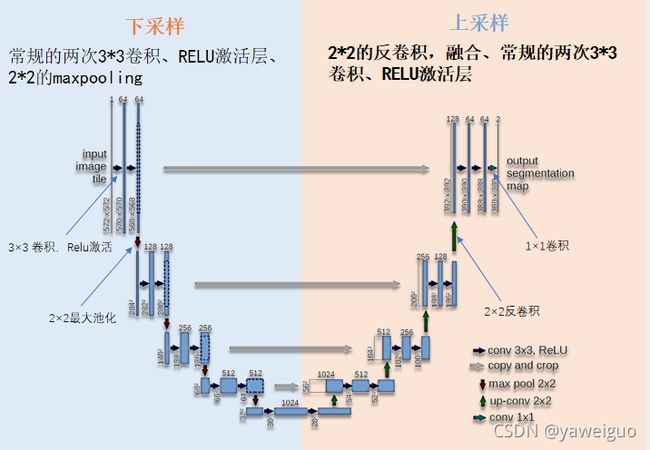
Unet可以分为三个部分,如下图所示:
第一部分是主干特征提取部分,Unet的主干特征提取部分与VGG相似,由卷积+最大池化组成,为卷积和最大池化的堆叠。卷积层用来初步提取有效特征,再通过最大池化进行下采样获得一个有效特征层。利用主干特征提取部分我们可以获得五个初步有效特征层,在第二步中,我们会利用这五个有效特征层可以进行特征融合。
第二部分是加强特征提取部分,我们可以利用主干部分获取到的五个初步有效特征层进行上采样,然后再和主干部分中获得的有效特征层进行特征融合,获得一个最终的,融合了所有特征的有效特征层。
上采样是指将特征图的分辨率还原到原始图片的分辨率大小,此处用反卷积,即通过转置卷积核的方法来实现卷积的逆过程。
第三部分是预测部分,利用最终获得的最后一个有效特征层对每一个特征点进行分类,相当于对每一个像素点进行分类。
 unet就是一个全卷积神经网络,输入和输出都是图像,没有全连接层。较浅的高分辨率层用来解决像素定位的问题,较深的层用来解决像素分类的问题。
unet就是一个全卷积神经网络,输入和输出都是图像,没有全连接层。较浅的高分辨率层用来解决像素定位的问题,较深的层用来解决像素分类的问题。
Over-tile策略
深入浅出了解Unet
Unet使用一种称为overlap-tile的的策略,使得任意大小输入的图片都可以获得一个无缝分割
该策略的思想是:对图像的某一块像素点(黄框内部分)进行预测时,需要该图像块周围的像素点(蓝色框内)提供上下文信息(context),以获得更准确的预测
这样的策略会带来一个问题,图像边界的图像块没有周围像素,因此作者对周围像素采用了镜像扩充。原始图片周围扩充的像素点均由原图对称得到。这样,边界图像块也能得到准确的预测
作者在卷积时只使用有效部分,使得最终传到下一层的只有中间原先图像块(黄色框内)的部分(可理解为不加padding),来解决第一块图像周围的部分会和第二块图像重叠的问题
二、代码运行
unet-master\data\membrane\train\image 中存放的是原数据集
unet-master\data\membrane\train\label 中存放的是数据集对应的标签
unet-master\data\membrane\text 中存放的是预测用的图片,x_predict.png 是之前预测出来的结果,重新预测需要删掉
修改各个文件夹路径,运行main,py进行训练
main.py:
# main_train.py
from model import *
from mydata1 import *
# #os.environ["CUDA_VISIBLE_DEVICES"] = "0"
# data_gen_args = dict() : 为keras自带的图像增强方法
data_gen_args = dict(rotation_range=0.2, # 整数。随机旋转的度数范围。
width_shift_range=0.05, # 浮点数、一维数组或整数
height_shift_range=0.05, # 浮点数。剪切强度(以弧度逆时针方向剪切角度)。
shear_range=0.05,
zoom_range=0.05, # 浮点数 或 [lower, upper]。随机缩放范围
horizontal_flip=True,
fill_mode='nearest') # {"constant", "nearest", "reflect" or "wrap"} 之一。默认为 'nearest'。输入边界以外的点根据给定的模式填充:
# 数据增强时变换方式的字典
# 建立测试集,样本和标签分别放在同一个目录下的两个文件夹中,文件夹名字为:'image','label'
# 得到一个生成器,以batch=2的速率无限生成增强后的数据 save_to_dir为增强后的数据图片的输出路径
myGene = trainGenerator(2, 'F:/unet/pic5', 'image', 'label', data_gen_args, save_to_dir=None) # data
# 调用模型,默认模型输入图像size=(256,256,1),样本位深为8位
model = unet() # model
# 保存训练的模型参数到指定的文件夹,格式为.hdf5; 检测的值是'loss'使其最小。第三个是只保存在验证集上性能最好的模型
#回调函数
model_checkpoint = ModelCheckpoint('F:/unet/unet_membrane.hdf5', monitor='loss', verbose=1,
save_best_only=True) # keras
# 开始训练,steps_per_epoch为迭代次数, epochs:为进行多少次循环
model.fit_generator(myGene, steps_per_epoch=300, epochs=1, callbacks=[model_checkpoint]) # keras
#steps_per_epoch指的是每个epoch有多少个batch_size,也就是训练集总样本数除以batch_size的值
#上面一行是利用生成器进行batch_size数量的训练,样本和标签通过myGene传入
# 输入测试数据集
testGene = testGenerator('data/membrane/text3')
# 预测数据testGene,5, verbose=1 5是要预测text文件夹中的前多少张图片
results = model.predict_generator(testGene,5,verbose=1)
#预测生成的标注图片的存储位置
saveResult("data/membrane/texting",results)
训练完成之后会生成一个unet_membrane.hdf5文件,并且会自动使用模型权重进行一次预测,预测结果存放在设置好的文件夹内
单独的预测代码:
text.py
from model import *
from mydata1 import *
import cv2 as cv
# python main_test.py
"""
注:
A: target_size()为图片尺寸,要求测试集图像尺寸设置和model输入图像尺寸保持一致,
如果不设置图片尺寸,会对输入图片做resize为处理,输入网络和输出图像尺寸默认均为(256,256),
B: 且要求图片位深为8位,24/32的会报错!!
C: 测试集数据名称需要设置为:0.png……
D:model.predict_generator( ,n, ):n为测试集中样本数量,需要手动设置,不然会报错!!
"""
def testGenerator(test_path, target_size=(256,256)):
for pngfile in glob.glob(test_path + "/*.png"):#查找有关目录下的所有文件,pngfile为路径
img = cv.imread(pngfile)#图片读取函数 在OpenCV第一个参数是指定需要读取的图片的路径和图片名,另一个参数,常用的就是"IMREAD_UNCHANGED"、"IMREAD_GRAYSCALE"、"IMREAD_COLOR"三个属性
img = cv.cvtColor(img, cv.COLOR_BGR2GRAY) #将RGB图片转化为灰度图
img = img / 255.0 #正则化
img = np.reshape(img, img.shape + (1,))
img = np.reshape(img, (1,) + img.shape)#将测试图片扩展一个维度,与训练时的输入[2,256,256]保持一致
yield img
# 输入测试数据集
testGene = testGenerator("F:/unet/unet-master/data/membrane/text4", target_size=(256, 256)) # data
# 导入模型
model = unet(input_size=(256, 256, 1)) # model
# 导入训练好的模型
model.load_weights("F:/unet/unet-master/unet_membrane.hdf5")
# 预测数据testGene,20, verbose=1
results = model.predict_generator(testGene, 10, verbose=1) # keras
print(results)
#预测生成的标注图片的存储位置
saveResult("F:/unet/unet-master/data/membrane/texting", results) # data
print("over")
三、制作自己的数据集进行训练并测试标注
##1.labelme的下载及标注
我们使用labelme进行数据集的标注
打开命令行输入
pip install pyqt5
pip install labelme -i https://pypi.tuna.tsinghua.edu.cn/simple
等待安装
提示安装成功
在命令行输入labelme打开软件进行标注
标注效果如下
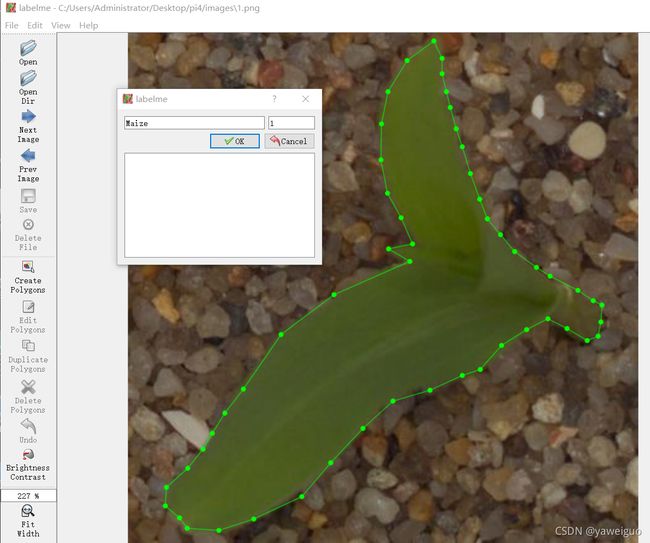
可参考此视频进行标注
https://www.bilibili.com/video/BV1St4y1r7hE?p=13
标注完成后会在你选择的保存路径下生成josn标签文件
需要运行下面代码将josn文件转换成标签图片,路径需要自己修改
将josn文件批量转换成标签图像:
import json
import os
import numpy as np
from PIL import Image
from labelme import utils
def json2mask_multi(json_path, save_path):
if not os.path.exists(save_path):
os.makedirs(save_path)
for json_name in os.listdir(json_path):
data = json.load(open(os.path.join(json_path, json_name)))
json_name = json_name.split('.')[0]
# 根据imageData字段的字符可以得到原图像
img = utils.img_b64_to_arr(data['imageData'])
# lbl为label图像(用类别名对应的数字来标,背景为0)
# lbl_names为label名和数字的对应关系字典
lbl, lbl_names = utils.labelme_shapes_to_label(img.shape, data['shapes'])
mask = Image.fromarray(lbl).convert('L')
# putpalette给对象加上调色板,相当于上色:R,G,B
# 三个数一组,对应于RGB通道,可以自己定义标签颜色
mask.putpalette([0, 0, 0,
255, 255, 255,
255, 255, 255,
128, 128, 128])
mask.save(os.path.join(save_path, json_name + ".png"))
json_path = "C:/Users/Administrator/Desktop/pi4/labels"
save_path = "C:/Users/Administrator/Desktop/pi4/label3"
json2mask_multi(json_path,save_path)
注意:此代码中unet网络输入的标签图片为灰色图像,输入的数据集图像也是灰色的,但是将数据集图片输入网络中后会被强制转换成256x256大小的灰度图像,所以数据集是RGB图像没有影响,但是标签需要自己将其转换成位深为8的灰色图片如下。 预测的图片数据集也要是灰色图像,原文中的代码是没有将预测集的图片转换成灰色图像的,需要自己转换,也可以在预测的代码中testGenerator()导入图片之后将其转换为灰色在进行预测,上面所给的txet代码中有更改
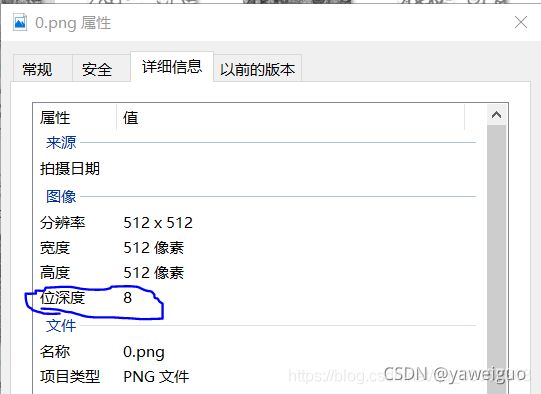
如若不是需要运行如下代码将其改为灰色图像
path :图片存放的路径
newpath :转化完成后,存放的路径
convert :需要更改成那一种为深度的图片的格式.
'''
import os
from PIL import Image
path = r'F:\unet\unet-master\data\membrane\train\image'
newpath = r'F:\unet\unet-master\data\membrane\train\image - 灰'
def picture(path):
files = os.listdir(path)
for i in files:
files = os.path.join(path, i)
img = Image.open(files).convert('L')
dirpath = newpath
file_name, file_extend = os.path.splitext(i)
dst = os.path.join(os.path.abspath(dirpath), file_name + '.png')
img.save(dst)
picture(path)
##2.训练
准备好数据集和标签图像之后将其和原来的细胞数据集和标签进行替换,然后就可以运行main.py函数进行训练自己的数据集了
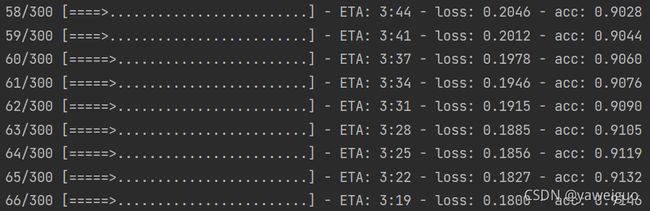
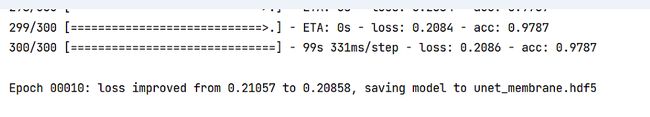 可以通过调节学习率来获得适和自己数据集的效果。一般loss达到0.07以下 acc达到95以上是比较正常的训练结果
可以通过调节学习率来获得适和自己数据集的效果。一般loss达到0.07以下 acc达到95以上是比较正常的训练结果
预测结果如下:

2.
关于预测结果为全黑全白或灰色的解决办法:
1.用来训练的图片数据集大小须是32的倍数,并且尽量宽高相等。
2.检测标签图像是否正确,须是位深为8的灰度图像,注意,不能使用涂抹的方法制作标签,须是标注的josn文件转换生成的标签图片
3.测试集文件也需要转换为灰度图像再输入进行预测
4.看自己训练时的损失和验证的正确率是否正常,可以通过调节学习率参数调整,一般损失和验证的正确率都很大的训练情况是不正常的

