脑机接口实例二:脑电信号CSP处理
文章目录
- 前言
- 一、实例说明
-
- 1.1 期望目标
- 1.2 数据解读
- 二、实例代码及结果分析
-
- 2.1 MATLAB代码
- 2.2 运行结果及分析
- 三、问题与反思
- 四、下一阶段计划
前言
本文在实例一的基础之上,选用BCI大赛08年数据集,基于CSP的特征提取,运用AdaBoost算法进行集成分类,以期实现运动想象中左、右手动作脑电信号的分类。
一、实例说明
1.1 期望目标
- 掌握数据集预处理的方法
- 实际运用CSP算法进行特征提取
- 对提取的特征进行boosting分类(AdaBoost是其中一种)
1.2 数据解读
- 实验范式
数据集由9名受试者的脑电图数据组成。BCI范式由四个不同的运动想象任务组成,即左手(类别1)、右手(类别2)、双脚(类别3)和舌头(类别4)的运动想象。范式记录不同日期的两天数据。(每天6圈,每圈48trails)。(共计576个trails,每个任务144trails)
正式范式之前,记录5分钟估计EOG值,分三块:看屏幕上的十字(2分钟)、闭眼(1分钟)、眼动(1分钟),注:由于技术问题被试A04T的眼动数据较短,仅包含眼动状态。如下图所示:
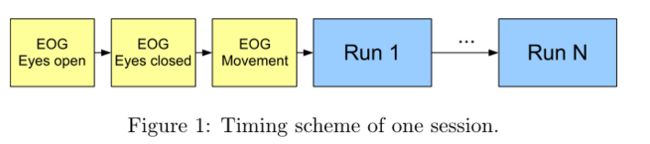
被试者坐在电脑屏幕前面的舒适的扶手椅上,在试验开始时(t=0s),黑屏上出现了一个十字。此外,还提出了一种短声告警音。两秒后(t=2s),以箭头的形式指向左、右、下或上(对应于四个类中左手、右手、脚或舌头的一个)的提示,出现在屏幕上1.25秒。这促使受试者完成所需的运动想象任务。不提供任何反馈。被试在t=6s时被要求执行运动想象任务,直到屏幕上的十字消失为止。随后屏幕再次为黑色的短暂中断,流程如下图所示:(有效数据为2-6s,即运动想象任务记录区间,亦为需提取数据的区间)

- 数据记录
250Hz采样,并在0.5Hz和100Hz之间进行带通滤波。除了22个脑电图通道,3个单极EOG通道,以250Hz的频率记录和采样(见下图)。它们在0.5Hz和100Hz之间进行带通滤波,放大器的灵敏度设置为1mV。EOG通道是为伪像处理方法的后续应用提供的,不得用于分类。

- 数据文件描述
事件类型描述如下 (event type 769/770 对应左右手的动作,以此为基准进行数据提取) :

二、实例代码及结果分析
2.1 MATLAB代码
主函数(main):
clc;clear;
%% 读取数据并对数据预处理
Subjects = 9; %被试数
Fs = 250; %采样率
windowLength = 4; %单次采样时间
chanSelect = [8,10,12]; %通道选择c3,c4,cz
totalFlt = [4 40]; %总的滤波频段选择
load rawdata1.mat
[Data_train,label_train] = preProccess(Fs,windowLength,EEG,chanSelect,totalFlt); %preProccess执行数据提取、分段、滤波处理
load rawdata2.mat
[Data_test,label_test] = preProccess(Fs,windowLength,EEG,chanSelect,totalFlt);
%% CSP特征提取
EEGSignals.x = Data_train;
EEGSignals.y = label_train;
Y = label_train;
classLabels = unique(EEGSignals.y);
CSPMatrix = learnCSP(EEGSignals,classLabels);
nbFilterPairs = 1;
X = extractCSP(EEGSignals, CSPMatrix, nbFilterPairs);
EEGSignals_test.x=Data_test;
EEGSignals_test.y=label_test;
T = extractCSP(EEGSignals_test, CSPMatrix, nbFilterPairs);
save dataCSP.mat X Y T
color_L = [0 102 255] ./ 255;
color_R = [255, 0, 102] ./ 255;
pos = find(Y==1);
plot(X(pos,1),X(pos,2),'x','Color',color_L,'LineWidth',2);
hold on
pos = find(Y==2);
plot(X(pos,1),X(pos,2),'o','Color',color_R,'LineWidth',2);
legend('Left Hand','Right Hand')
xlabel('C3','fontweight','bold')
ylabel('C4','fontweight','bold')
CSP算法:
function CSPMatrix = learnCSP(EEGSignals,classLabels)
%
%Input:
%EEGSignals: the training EEG signals, composed of 2 classes. These signals
%are a structure such that:
% EEGSignals.x: the EEG signals as a [Ns * Nc * Nt] Matrix where
% Ns: number of EEG samples per trial
% Nc: number of channels (EEG electrodes)
% nT: number of trials
% EEGSignals.y: a [1 * Nt] vector containing the class labels for each trial
% EEGSignals.s: the sampling frequency (in Hz)
%
%Output:
%CSPMatrix: the learnt CSP filters (a [Nc*Nc] matrix with the filters as rows)
%
%See also: extractCSPFeatures
%check and initializations
nbChannels = size(EEGSignals.x,2);
nbTrials = size(EEGSignals.x,3);
nbClasses = length(classLabels);
if nbClasses ~= 2
disp('ERROR! CSP can only be used for two classes');
return;
end
covMatrices = cell(nbClasses,1); %the covariance matrices for each class
%% Computing the normalized covariance matrices for each trial
trialCov = zeros(nbChannels,nbChannels,nbTrials);
for t=1:nbTrials
E = EEGSignals.x(:,:,t)'; %note the transpose
EE = E * E';
trialCov(:,:,t) = EE ./ trace(EE);
end
clear E;
clear EE;
%computing the covariance matrix for each class
for c=1:nbClasses
covMatrices{c} = mean(trialCov(:,:,EEGSignals.y == classLabels(c)),3); %EEGSignals.y==classLabels(c) returns the indeces corresponding to the class labels
end
%the total covariance matrix
covTotal = covMatrices{1} + covMatrices{2};
%whitening transform of total covariance matrix
[Ut Dt] = eig(covTotal); %caution: the eigenvalues are initially in increasing order
eigenvalues = diag(Dt);
[eigenvalues egIndex] = sort(eigenvalues, 'descend');
Ut = Ut(:,egIndex);
P = diag(sqrt(1./eigenvalues)) * Ut';
%transforming covariance matrix of first class using P
transformedCov1 = P * covMatrices{1} * P';
%EVD of the transformed covariance matrix
[U1 D1] = eig(transformedCov1);
eigenvalues = diag(D1);
[eigenvalues egIndex] = sort(eigenvalues, 'descend');
U1 = U1(:, egIndex);
CSPMatrix = U1' * P;
2.2 运行结果及分析
图一,由算法代码进行预处理数据后得到的CSP特征提取结果
图二,由导入eeglab进行预处理数据后得到的CSP特征提取结果
分析: 由上图,我们可以看出,当前CSP算法对数据进行了一定程度的特征提取,但特征之间并没有达到理想的分离程度,部分数据集中纠缠在一起。产生此结果的可能原因:(1)数据提取、预处理阶段存在不足;(2)该CSP算法自身存在缺陷(如本实例将导联关系限制在C3、C4、CZ三个通道,与其他通道导联的相关性难以体现)。针对可能原因1,我将数据导入eeglab,用eeglab自身的预处理功能,对数据进行预处理操作,再应用CSP算法进行特征提取,但实验结果同样不理想,如图二所示。于是,猜想出现不理想结果可能更多的是原因2造成的,接下来,我将尝试应用不同的方法,对特征进行提取。
分析: 从CSP提取的特征中,选择144个数据点组成训练集,进行30次迭代集成分类,由图可以看出,随着迭代次数的增加,训练错误率在降低,即分类准确率在提高。尽管如此,分类的错误率仍超过30%,结果并不理想,也在侧面说明,CSP算法存在问题,有进一步改进的空间。
三、问题与反思
- 数据预处理的实际应用能力较弱。例如,在对数据进行分段提取,标签定义,以及应用IIR 滤波,ICA眼电信号去除进行数据预处理的过程中,由于对数组的理解不够准确,时常报错;
- CSP特征提取算法掌握不足,实际应用中,对代码的理解尚浅薄,不能灵活应用和变通;
- 反思:理论知识结合到实际应用中的能力有待加强,接下来,会尝试采用不同的算法实现脑电信号的分类任务,进一步在实践中,加深对算法的理解、掌握。
四、下一阶段计划
- 实例结果的分析表明,当前CSP算法进行特征提取的效果不够理想,有进一步改进的空间,下一阶段计划将围绕这个主题展开。
- 任务一:尝试采用不同的算法,对特征进行提取,以期得到更好的实验结果。
- 任务二:脑机接口理论知识储备不足,尝试系统性学习相关知识。
- 任务三:加强相关算法的学习与应用(分类算法掌握尚浅)。
本文源码:
链接:https://pan.baidu.com/s/1TzEskR1vP4TMx64aTOHRBg (百度网盘)
提取码:muda


