【深度学习】BioBERT文章翻译及个人感悟
文章地址:【https://arxiv.org/abs/1901.08746】
0. 摘要
0.1 动机
随着生物医学文献数量的快速增长,生物医学文本挖掘变得越来越重要。 随着自然语言处理(NLP)的进步,从生物医学文献中提取有价值的信息已在研究人员中普及,深度学习促进了有效的生物医学文本挖掘模型的发展。 但是,由于单词分布从一般领域的语料库转移到生物医学的语料库,直接将NLP的进步应用到生物医学的文本挖掘中通常会产生不令人满意的结果。 在本文中,我们研究了最近引入的预训练语言模型BERT如何适用于生物医学语料库。
0.2 结果
我们介绍了BioBERT(用于生物医学文本挖掘的双向编码器表示Transformers),这是一种在大型生物医学语料库上预先训练的领域特定语言表示模型。 通过在任务上几乎相同的体系结构,在经过生物医学语料库的预训练之后,BioBERT在许多生物医学文本挖掘任务中都大大优于BERT和以前的最新模型。 尽管BERT的性能可与以前的最新模型相媲美,但在以下三个代表性的生物医学文本挖掘任务上,BioBERT的性能明显优于它们:生物医学命名实体识别(F1分数提高0.62%),生物医学关系提取(2.80%) F1分数提高)和生物医学问答(MRR提高12.24%)。 我们的分析结果表明,对生物医学语料库进行BERT的预培训有助于其理解复杂的生物医学文献。
0.3 可用性和应用
我们可以在https://github.com/naver/biobert-pretrained上免费提供BioBERT的预训练权重,并在https://github.com/dmis-lab/biobert上提供用于微调BioBERT的源代码。
1.介绍
生物医学文献的数量继续迅速增加。 平均而言,每天在同行评审的期刊上发表3000多篇新文章,不包括各种档案中的预览版和技术报告,例如临床试验报告。 截至2019年1月,仅PubMed就有2900万篇文章。包含有关新发现和新见解的宝贵信息的报告不断地被添加到本已大量的文献中。 因此,越来越需要用于从文献中提取信息的准确的生物医学文本挖掘工具。
自然语言处理(NLP)中使用的深度学习技术的进步,使生物医学文本挖掘模型的最新进展成为可能。例如,在过去几年中,长短期记忆(LSTM)和条件随机场(CRF)在生物医学命名实体识别(NER)方面的性能有了很大提高(Giorgi和Bader,2018 ; Habibi 等人,2017 ; Wang 等人,2018 ; Yoon 等人,2019)。其他基于深度学习的模型也改善了生物医学文本挖掘任务,例如关系提取(RE)(Bhasuran和Natarajan,2018年 ; Lim和Kang,2018年))和问答(QA)(Wiese 等人,2017年)。
但是,将最新的NLP方法直接应用于生物医学文本挖掘具有局限性。首先,随着最近的单词表示模型(例如Word2Vec(Mikolov 等,2013),ELMo(Peters 等,2018)和BERT(Devlin 等,2019)都在包含通用领域文本的数据集上进行训练和测试(例如Wikipedia),很难估计它们在包含生物医学文本的数据集上的表现。同样,普通语料库和生物医学语料库的单词分布也有很大不同,这对于生物医学文本挖掘模型通常可能是一个问题。结果,生物医学文本挖掘中的最新模型很大程度上依赖于单词表示形式的改编版本(Habibi 等, 2017 ; Pyysalo 等,2013年)。
在这项研究中,我们假设需要在生物医学语料库上训练当前最先进的词表示模型(例如BERT),以有效地进行生物医学文本挖掘任务。以前,Word2Vec是最广为人知的上下文无关单词表示模型之一,它是在生物医学语料库上进行训练的,该语料库包含通常不包含在通用领域语料库中的术语和表达(Pyysalo 等人,2013年)。尽管ELMo和BERT已经证明了上下文化词表示法的有效性,但它们在生物医学语料库上无法获得高性能,因为它们仅在通用领域语料库上进行了预训练。由于BERT在各种NLP任务上都取得了非常出色的结果,同时在整个任务中使用几乎相同的结构,因此将BERT应用于生物医学领域可能会有益于众多生物医学NLP研究。
2. 方法
在本文中,我们介绍了BioBERT,它是针对生物医学领域的预先训练的语言表示模型。预训练和微调BioBERT的总体过程如图1所示。首先,我们使用BERT的权重初始化BioBERT,该BERT已在通用领域语料库(英语Wikipedia和BooksCorpus)上进行了预训练。然后,对BioBERT进行生物医学领域语料库的预训练(PubMed摘要和PMC全文文章)。为了显示我们的方法在生物医学文本挖掘中的有效性,我们对BioBERT进行了微调,并对三种流行的生物医学文本挖掘任务(NER,RE和QA)进行了评估。我们使用通用领域语料库和生物医学语料库的不同组合和大小来测试各种预训练策略,并分析每种语料库对预训练的影响。我们还提供了BERT和BioBERT的深入分析,以显示我们的预训练策略的必要性。
本文的贡献如下:
-
BioBERT是第一个基于领域特定BERT的模型,已在生物医学语料库上经过八个NVIDIA V100 GPU进行了23天的预训练。
-
我们表明,对BERT进行生物医学语料库的预培训可以大大提高其性能。与当前的最新模型相比,BioBERT在生物医学NER(0.62)和生物医学RE(2.80)中获得了更高的F1评分,在生物医学QA中获得了更高的MRR得分(12.24)。
-
与大多数以前主要专注于单个任务(例如NER或QA)的生物医学文本挖掘模型相比,我们的模型BioBERT可在各种生物医学文本挖掘任务上实现最先进的性能,而仅需进行最小的架构修改。
-
我们公开提供了经过预处理的数据集,经过训练的BioBERT权重以及用于微调BioBERT的源代码。
3. 模型及预料
BioBERT基本上具有与BERT相同的结构。我们简要讨论最近提出的BERT,然后详细描述BioBERT的预训练和微调过程。
3.1 BERT
从大量未注释的文本中学习单词表示法是一种悠久的方法。先前的模型(例如Word2Vec(Mikolov 等,2013),GloVe(Pennington 等,2014))专注于学习上下文无关的单词表示,而最近的工作则专注于学习上下文相关的单词表示。例如,ELMo(Peters 等人,2018)使用双向语言模型,而CoVe(McCann 等人,2017)使用机器翻译将上下文信息嵌入单词表示中。
BERT(Devlin 等人,2019)是一个基于上下文的单词表示模型,该模型基于标注语言模型并使用双向Transformer 进行了预训练(Vaswani 等人,2017)。由于无法看到将来的单词的语言建模的本质,以前的语言模型仅限于两个单向语言模型(即从左到右和从右到左)的组合。BERT使用掩蔽语言模型来预测序列中的随机掩蔽词,因此可用于学习双向表示。而且,它在大多数NLP任务上都具有最先进的性能,同时只需要最小的特定于任务的体系结构修改。BERT的作者认为,合并双向表示而不是单向表示的信息对于以自然语言表示单词至关重要(Krallinger 等,2017)。由于篇幅所限,我们请读者参考Devlin 等。(2019)进一步了解BERT。
3.2 预训练BioBERT
作为通用语言表示模型,BERT在英文Wikipedia和BooksCorpus上进行了预培训。但是,生物医学领域文本包含相当多的领域特定专有名词(例如BRCA1,c.248T> C)和术语(例如转录,抗菌剂),大多数生物医学研究人员都理解它们。结果,为通用语言理解而设计的NLP模型通常在生物医学文本挖掘任务中表现不佳。在这项工作中,我们对BioBERT进行了PubMed摘要(PubMed)和PubMed Central全文文章(PMC)的预训练。表1列出了用于BioBERT的预训练的文本语料库,表2列出了经过测试的文本语料库组合。为了提高计算效率,每当使用Wiki + Books语料库进行预训练时,我们就使用Devlin 等提供的预训练的BERT模型初始化BioBERT (2019)。我们将BioBERT定义为一种语言表示模型,其预训练语料库包括生物医学语料库(例如BioBERT(+ PubMed))。
表1:用于BioBERT的文本语料库列表
| 语料库 | 单词数 | 所属领域 |
|---|---|---|
| English Wikipedia | 25亿 | 通用 |
| BooksCorpus | 8亿 | 通用 |
| PubMed摘要 | 45亿 | 生物医学 |
| PMC全文文章 | 135亿 | 生物医学 |
表2:在以下文本语料库的不同组合上对BioBERT进行预训练:English Wikipedia(Wiki),BooksCorpus(Books),PubMed摘要(PubMed)和PMC全文文章(PMC)
| 模型 | 语料库组合 |
|:-----:-----:-----
| BERT(Devlin 等人,2019) | Wiki +Books |
| BioBERT(+ PubMed) | Wiki +Books+ PubMed |
| BioBERT(+ PMC) | Wiki +Books+ PMC |
| BioBERT(+ PubMed + PMC) | Wiki +Books+ PubMed + PMC |
关于词性标注,BioBERT使用WordPiece标注法(Wu 等,2016),这缓解了OOV(未登录词)的问题。使用WordPiece标注法,任何新单词都可以由频繁的子单词表示(例如Immunoglobulin => I ## mm ## uno ## g ## lo ## bul ## in)。我们发现,使用带格词汇(而不是小写)可以在下游任务中获得更好的性能。尽管我们可以基于生物医学语料库构建新的WordPiece词汇表,但是我们使用了BERT BASE的原始词汇表 出于以下原因:
(i)BioBERT与BERT的兼容性,这允许重新使用在通用域语料库上进行过预训练的BERT,并使得更容易互换使用基于BERT和BioBERT的现有模型
(ii)任何新的仍然可以使用BERT的原始WordPiece词汇表来对生物医学领域的单词进行表示和微调。
3.3 微调BioBERT
只需进行最小的架构修改,即可将BioBERT应用于各种下游文本挖掘任务。我们针对以下三个具有代表性的生物医学文本挖掘任务对BioBERT进行了调整:NER,RE和QA。
命名实体识别 是最基本的生物医学文本挖掘任务之一,其中涉及到识别生物医学语料库中的众多领域特定专有名词。虽然以前的大多数解决方案都是基于LSTM和CRF的不同组合构建的(Giorgi和Bader,2018年 ; Habibi 等人,2017年 ; Wang 等人,2018年),但BERT具有基于双向 Transformer 的简单架构。BERT基于来自最后一层的表示使用单个输出层来仅计算令牌级别的BIO2概率。注意:虽然以前在生物医学NER中的工作经常使用在PubMed或PMC语料库上训练的词嵌入(Habibi 等,2017 ;Yoon 等。,2019),BioBERT在预训练和微调期间直接学习WordPiece嵌入。对于NER的评估指标,我们使用了实体级别的准确性,召回率和F1得分。
关系提取 是对生物医学语料库中命名实体的关系进行分类的任务。我们利用了BERT原始版本的句子分类器,该分类器使用[CLS]标记对关系进行分类。基于来自BERT的[CLS]令牌表示,使用单个输出层执行句子分类。我们使用预定义标签(例如@ GENE $或@ DISEASE $)使句子中的目标命名实体匿名。例如,具有两个目标实体(在这种情况下为基因和疾病)的句子表示为“ @ GENE $位置986处的丝氨酸可能是血管造影@ DISEASE $的独立遗传预测因子。”的精确度,召回率和F1得分报告RE任务。
问题解答 是根据相关段落回答以自然语言提出的问题的任务。为了对BioBERT进行质量检查进行微调,我们使用了与SQuAD相同的BERT架构(Rajpurkar 等,2016)。我们使用了BioASQ事实数据集,因为它们的格式与SQuAD相似。使用单个输出层计算答案短语开始/结束位置的令牌级别概率。但是,我们发现,在提取的质量检查设置中,约有30%的BioASQ拟事实问题无法回答,因为确切的答案并未出现在给定的段落中。像Wiese 等人。(2017),我们从训练集中排除了具有无法回答的问题的样本。此外,我们使用了相同的预训练过程Wiese 等。(2017),它使用SQuAD,并在很大程度上改善了BERT和BioBERT的性能。我们使用了BioASQ的以下评估指标:严格的准确性,宽松的准确性和平均倒数排名。
4. 结果
4.1 数据集
如下表3列出了生物医学NER数据集的统计数据。我们使用了Wang 等提供的所有NER数据集的预处理版本。(2018),但2010 i2b2 / VA,JNLPBA和Species-800数据集除外。由于从其训练集中删除了重复的文章,因此预处理的NCBI疾病数据集的注释少于原始数据集。我们使用CoNLL格式(https://github.com/spyysalo/standoff2conll)预处理2010 i2b2 / VA和JNLPBA数据集。Species-800数据集已根据Pyysalo(https://github.com/spyysalo/s800)的数据集进行了预处理和拆分)。我们没有为BC2GM数据集使用备用注释,并且所有NER评估均基于实体级别的精确匹配。请注意:尽管最近还有其他一些引入的高质量生物医学NER数据集(Mohan和Li,2019),但我们使用了许多生物医学NLP研究人员经常使用的数据集,这使得将我们的工作与他们的工作进行比较变得容易得多。
RE数据集包含基因-疾病关系和蛋白质-化学关系(表4)。我们提供的代码提供了预处理的GAD和EU-ADR数据集。对于CHEMPROT数据集,我们使用了Lim和Kang(2018)中描述的相同预处理程序。我们使用了BioASQ事实数据集,可以将其转换为与SQuAD数据集相同的格式(表5)。我们使用了BioASQ组织者提供的完整摘要(PMID)以及相关的问题和答案。我们已经公开提供了经过预处理的BioASQ数据集。对于所有数据集,我们使用先前工作中使用的相同数据集拆分(Lim和Kang,2018;Tsatsaronis 等,2015;Wang 等,2018)进行公平评估;但是,从Giorgi和Bader(2018)中找不到LINAAEUS和Species-800的分裂,并且可能有所不同。像以前的工作(Bhasuran和Natarajan,2018年)一样,我们报告了对没有单独测试集(例如GAD,EU-ADR)的数据集进行10倍交叉验证的性能。
表3:生物医学命名实体识别数据集的统计数据:
| 数据集 | 实体类型 | 标注实体数量 |
|---|---|---|
| NCBI疾病(Dogan 等,2014) | 疾病 | 6881 |
| 2010 i2b2 / VA(Uzuner等,2011) | 疾病 | 19665 |
| BC5CDR(Li 等,2016) | 疾病 | 12694 |
| BC5CDR(Li 等,2016) | 药物/化学 | 15411 |
| BC4CHEMD(Krallinger 等,2015) | 药物/化学 | 79842 |
| BC2GM(Smith 等,2008) | 基因/蛋白质 | 20703 |
| JNLPBA(Kim 等,2004年) | 基因/蛋白质 | 35460 |
| LINNAEUS(Gerner 等,2010) | 种类 | 4077 |
| 物种-800(Pafilis 等,2013) | 种类 | 3708 |
注意:实体标注数量统计由:Habibi (2017)和Zhu (2018)等人提供。
表4:生物医学关系提取数据集的统计:
| 数据集 | 实体类型 | 关系数 |
|---|---|---|
| GAD(Bravo et al。,2015) | 基因疾病 | 5330 |
| 欧盟ADR(Van Mulligen et al。,2012) | 基因疾病 | 355 |
| CHEMPROT(Krallinger 等人,2017) | 蛋白质化学 | 10031 |
注意:对于CHEMPROT数据集,对训练,验证和测试集中的关系数求和。
表5:生物医学问答数据集的统计:
| 数据集 | 训练集大小 | 测试集大小 |
|---|---|---|
| BioASQ 4b-类固醇(Tsatsaronis 等,2015) | 327 | 161 |
| BioASQ 5b-类固醇(Tsatsaronis 等,2015) | 486 | 150 |
| BioASQ 6b-类固醇(Tsatsaronis 等,2015) | 618 | 161 |
我们将BERT和BioBERT与当前的最新模型进行比较,并报告其得分。请注意,每个最新模型都具有不同的体系结构和训练过程。例如,Yoon 等人的最新模型。在JNLPBA数据集上训练的(2019)基于具有字符级CNN的多个Bi-LSTM CRF模型,而在LINNAEUS数据集上训练的Giorgi和Bader(2018)的最新模型使用Bi-LSTM CRF具有字符级LSTM的模型,并且还接受了银标准数据集的培训。另一方面,BERT和BioBERT具有完全相同的结构,并且仅使用金标准数据集,而不使用任何其他数据集。
4.2 硬件环境及训练时长
我们使用了在英语维基百科和BooksCorpus上预训练的BERT BASE模型进行1M步。BioBERT v1.0(+ PubMed + PMC)是经过训练的470K步骤的BioBERT(+ PubMed + PMC)版本。当同时使用PubMed和PMC语料库时,我们发现200K和270K的预训练步骤分别对于PubMed和PMC是最佳的。我们还使用了BioBERT v1.0的消融版本,仅在PubMed上进行了200K步的预训练(BioBERT v1.0(+ PubMed)),在PMC上进行了270K步的训练(BioBERT v1.0(+ PMC))。在最初发布BioBERT v1.0之后,我们在PubMed上对BioBERT进行了100万步的预培训,我们将此版本称为BioBERT v1.1(+ PubMed)。除非另有说明,否则其他超参数(例如用于预训练BioBERT的批处理大小和学习速率计划)与用于预训练BERT的参数相同。
我们使用Naver智能机器学习(NSML)(Sung 等人,2017)对BioBERT进行了预培训,该技术用于需要在多个GPU上运行的大规模实验。我们使用了八个NVIDIA V100(32GB)GPU进行预训练。最大序列长度固定为512,最小批量大小设置为192,每次迭代产生98304个单词。在这种情况下,要对BioBERT v1.1(+ PubMed)进行BioBERT v1.0(+ PubMed + PMC)的培训要花费将近23天的时间,超过10天。尽管我们竭尽全力使用BERTLARGE,但由于BERTLARGE的计算复杂性,我们仅使用BERTBASE。
我们使用单个NVIDIA Titan Xp(12GB)GPU在每个任务上微调BioBERT。请注意,微调过程比预训练BioBERT的计算效率更高。为了进行微调,选择了10、16、32或64的批量大小,并选择了5e-5、3e-5或1e-5的学习率。由于训练数据的大小比Devlin 等人使用的训练数据小得多,因此在QA和RE任务上对BioBERT进行微调不到一个小时(2019)。另一方面,BioBERT在NER数据集上达到最高性能需要花费20多个epochs。
4.3 实验结果
NER的结果示于表6。首先,我们观察到仅对通用域语料库进行了预训练的BERT是非常有效的,但是BERT的微观平均F1得分要比最新模型低(2.01)。另一方面,在所有数据集上,BioBERT的得分均高于BERT。在9个数据集中,有6个数据集的BioBERT均优于最新模型,而按微观平均F1得分计算,BioBERT v1.1(+ PubMed)则较最新模型高0.62。LINNAEUS数据集的相对较低的得分可归因于以下方面:(i)缺乏用于训练先前的最新模型的银标准数据集,以及(ii)先前使用的不同训练/测试集划分作品(Giorgi and Bader,2018),这是不可用的。
表6:生物医学命名实体识别中的测试结果:

注意:报告每个数据集的精度(P),召回率(R)和F1(F)分数。最佳成绩以粗体显示,第二最佳成绩用下划线标出。我们在不同的数据集上列出了最新技术(SOTA)模型的得分,如下所示:Xu 等人的得分。(2019)关于NCBI疾病,Sachan 等人的分数。(2018)关于BC2GM,Zhu 等人的分数。(2018)(单一模型)于2010 i2b2 / VA,Lou 等人的得分。(2017)关于BC5CDR疾病,Luo 等人的评分。(2018) on BC4CHEMD,Yoon 等人的得分。(2019)BC5CDR-chemical和JNLPBA上的得分,以及Giorgi和Bader(2018)在LINNAEUS和Species-800上的得分。
每个模型的RE结果如表7所示。BERT的性能优于CHEMPROT数据集上最新模型的性能,这证明了它在RE中的有效性。从平均水平(微型)来看,BioBERT v1.0(+ PubMed)的F1得分高于最新模型。此外,BioBERT在3个生物医学数据集中有2个获得了最高的F1分数。
表7:生物医学关系提取测试结果

注意:报告每个数据集的精度(P),召回率(R)和F1(F)分数。最佳成绩以粗体显示,第二最佳成绩用下划线标出。GAD和EU-ADR的得分来自Bhasuran和Natarajan(2018),CHEMPROT的得分来自Lim和Kang(2018)。
QA结果显示在表8中。我们对每批最新模型的最佳分数进行微平均。BERT获得了比最新模型更高的微观平均MRR分数(高7.0)。所有版本的BioBERT均明显优于BERT和最新模型,尤其是BioBERT v1.1(+ PubMed)的严格准确度为38.77,宽大的准确度为53.81,平均倒数排名得分为44.77 ,所有数据均进行了微平均。在所有生物医学QA数据集上,BioBERT的MRR均达到了最新水平。
表8:生物医学问答测试结果:

注意:报告了每个数据集上的严格精度(S),宽容精度(L)和平均倒数排名(M)分数。最佳成绩以粗体显示,第二最佳成绩用下划线标出。最佳的BioASQ 4b / 5b / 6b分数来自BioASQ排行榜(http://participants-area.bioasq.org)。
5. 讨论
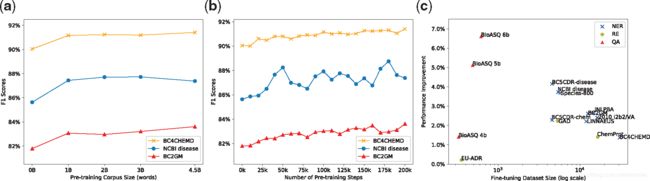
我们使用了其他大小不同的语料库进行预训练,并研究了它们对性能的影响。对于BioBERT v1.0(+ PubMed),我们将预训练步骤数设置为200K,并更改PubMed语料库的大小。图2(a)显示,BioBERT v1.0(+ PubMed)在三个NER数据集(NCBI Disease,BC2GM,BC4CHEMD)上的性能随PubMed语料库的大小而变化。对10亿个单词进行预训练是非常有效的,每个数据集的性能最多可以提高到45亿个单词。我们还在不同的预训练步骤中保存了BioBERT v1.0(+ PubMed)中的预训练权重,以衡量预训练步骤的数量如何影响其在微调任务上的性能。图2(b)显示了相对于预训练步骤数,相同的三个NER数据集上的BioBERT v1.0(+ PubMed)的性能变化。结果清楚地表明,随着预训练步骤数量的增加,每个数据集的性能都会提高。最后,图2(c)显示了在所有15个数据集上,相对于BERT,BioBERT v1.0(+ PubMed + PMC)的绝对性能提高。F1分数用于NER / RE,MRR分数用于QA。BioBERT大大提高了大多数数据集的性能。
(a)更改PubMed语料库大小以进行预训练的影响。 (b)BioBERT在不同检查点的NER性能。 (c)BioBERT v1.0(+ PubMed + PMC)与BERT相比的性能提高
在新标签页中打开下载幻灯片
(a)改变PubMed语料库大小以进行预训练的影响。(b)BioBERT在不同检查点的NER性能。(c)BioBERT v1.0(+ PubMed + PMC)与BERT相比的性能提高
如表9所示,我们对BERT和BioBERT v1.1(+ PubMed)的预测进行了采样,以了解预训练对下游任务的影响。BioBERT可以识别BERT无法识别的生物医学命名实体,并且可以找到命名实体的确切边界。虽然BERT通常会为简单的生物医学问题提供错误的答案,但是BioBERT会为此类问题提供正确的答案。此外,BioBERT可以提供名称较长的实体作为答案。
表9:来自NER和QA数据集的BERT和BioBERT的预测样本:

注意: NER的预测命名实体和QA的预测答案以粗体显示。
6. 结论
在本文中,我们介绍了BioBERT,它是用于生物医学文本挖掘的预训练语言表示模型。我们证明了在生物医学语料库上对BERT进行预培训对于将其应用于生物医学领域至关重要。只需对特定任务进行最小的架构修改,BioBERT就可以在生物医学文本挖掘任务(例如NER,RE和QA)上胜过先前的模型。
BioBERT的预发行版本(2019年1月)已被证明在许多生物医学文本挖掘任务中非常有效,例如临床笔记的NER(Alsentzer 等人,2019),人类表型基因RE(Sousa 等人。,2019)和临床颞叶RE(Lin 等人,2019)。以下BioBERT的更新版本将提供给bioNLP社区:(i)仅基于PubMed摘要进行训练的BioBERT BASE和BioBERT LARGE,无需从现有BERT模型进行初始化;以及(ii)根据特定领域的词汇进行训练的BioBERT BASE和BioBERT LARGE在WordPiece上。
7. 个人心得
根据阅读本论文,个人心得如下:
-
该论文旨在原本的BERT模型下,做了专业的数据收集与训练,从而能够对于专业领域内的命名实体识别、关系抽取以及问答内容得到不错的提升,本质上是数据集上的聚焦,模型层面的变化文章中并未有说明之处。
-
训练一套BERT模型确实耗时耗力,如果有相应已经与训练完成的权重的话,最好是能借鉴就借鉴,然后根据自己的需求做微调,这也是BERT出现之后很普遍的行业做法了,迁移学习将会是以后算法工程师应该必备的技能了。
-
另外就是关于数据在国内的应用,论文借鉴的依旧是英文相关数据,虽说作者们大多都是来自韩国,可见数据本身也是页内极其匮乏(并未有韩语相关数据),因此个人觉得工业级应用的话,首先依旧是积攒数据,认真做好基础工作。
-
最后,想要吐槽的一点是Bio应该包括的不仅仅是训练中提及的几种类型,而且也没有同同类型的模型结果做对比,而是对标的BERT,例如NER就没有与之前主流的BiLSTM+CRF做横向对比。有点看不见房间里的大象的意思。
感谢:感谢谷歌翻译
另外,以上观点纯属个人感悟,欢迎批评指正。