机器学习实战3-随机森林算法
文章目录
-
- 集成算法概述
- sklearn中的集成算法模块
- RandomForestClassifier
-
- 重要参数&&随机森林的分类器
-
- 控制基评估器的参数
- n_estimators
- sklearn建模流程复习
-
- 交叉验证
- 我们进行10次交叉验证,观察随机森林和决策树的效果
- n_estimators学习曲线
- bootstrap & oob_score
- 随机森林回归器
- 随机森林回归填补缺失值案例
- 机器学习中调参的基本思想
- 实例随机森林在乳腺癌数据上的调参
-
- 随机森林调参第一步:无论如何先调n_estimators
- 调max_depth
- 调min_samples_leaf
- 调min_samples_split
- 调criterion
集成算法概述


![]()
对于随机森林这个集成算法来说,它的基评估器就是决策树,决策树长成的森林就是随机森林也就是集成评估器。
sklearn中的集成算法模块
sklearn中的集成算法模块是ensemble,写法:sklearn.ensemble

RandomForestClassifier
随机森林是非常具有代表性的Bagging集成算法,它的所有基评估器都是决策树,分类树组成的随机森林就叫做随机森林的分类器,回归树所集成的森林就叫随机森林回归器
重要参数&&随机森林的分类器
控制基评估器的参数
n_estimators
这时森林中树木的数量,就是基评估器的数量,这个参数对随机森林模型的精确性影响是单调的n_estimators越大,模型的结果往往越好,但是相应的任何模型都有决策边界,当n_estimators达到一定的数值之后,随机森林的精确性就不会再提升了
sklearn建模流程复习
#实例化
#训练集带入实例化后的模型去训练,使用的接口是fit
#使用其他接口将测试集导入我们训练好的模型去获取我希望获取的结果(score, Ytest是我们预测出来的标签)
sklearn中所有的属性和标签是分开导入的
# 实例化
clf = DecisionTreeClassifier(random_state=0)
rfc = RandomForestClassifier(random_state=0)
# 训练
clf = clf.fit(Xtrain, Ytrain)
rfc = rfc.fit(Xtrain, Ytrain)
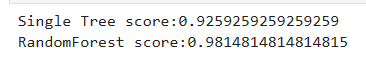
score_c = clf.score(Xtest, Ytest)
score_r = rfc.score(Xtest, Ytest)
print(f"Single Tree score:{score_c}")
print(f"RandomForest score:{score_r}")
交叉验证
# 交叉验证
from sklearn.model_selection import cross_val_score
import matplotlib.pyplot as plt
rfc = RandomForestClassifier(n_estimators=25)
rfc_s = cross_val_score(rfc, wine.data, wine.target, cv = 10)
clf = DecisionTreeClassifier()
clf_s = cross_val_score(clf, wine.data, wine.target, cv = 10)
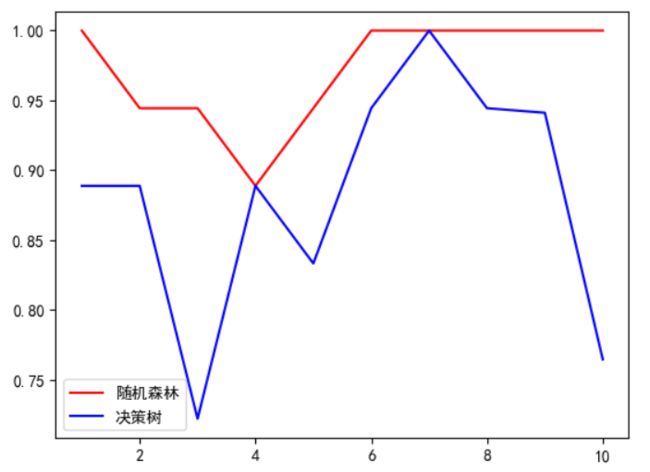
plt.plot(range(1, 11), rfc_s, label="随机森林", color = 'red')
plt.plot(range(1, 11), clf_s, label="决策树", color = 'blue')
plt.legend()
plt.show()
这里在画图的时候也是出现了一点小插曲,我们matplotlib不能直接在图中出现中文,这里可以下载中文字体百度解决
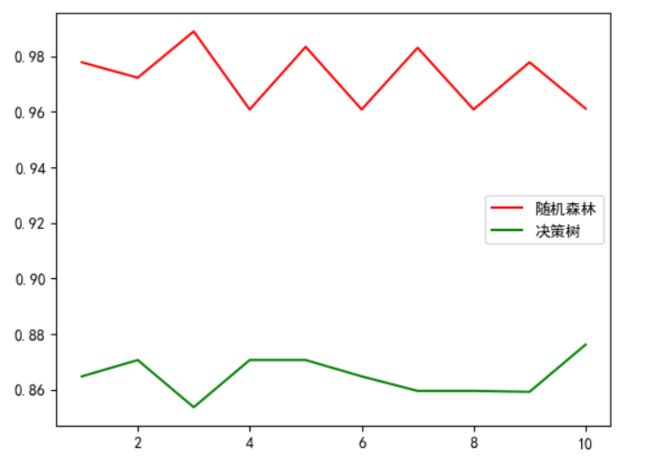
我们进行10次交叉验证,观察随机森林和决策树的效果
rfc_l = []
clf_l = []
for i in range(10):
rfc = RandomForestClassifier(n_estimators=25)
rfc_s = cross_val_score(rfc, wine.data, wine.target, cv=10).mean()
rfc_l.append(rfc_s)
clf = DecisionTreeClassifier()
clf_s = cross_val_score(clf, wine.data, wine.target, cv=10).mean()
clf_l.append(clf_s)
plt.plot(range(1, 11), rfc_l, color = 'red', label = "随机森林")
plt.plot(range(1, 11), clf_l, color = 'green', label = "决策树")
plt.legend()
plt.show()
n_estimators学习曲线
superpa = []
for i in range(200):
rfc = RandomForestClassifier(n_estimators=i+1, n_jobs=-1)
rfc_s = cross_val_score(rfc, wine.data, wine.target, cv=10).mean()
superpa.append(rfc_s)
print(max(superpa), superpa.index(max(superpa)))
plt.figure(figsize=[20, 5])
plt.plot(range(1,201), superpa)
plt.show()

可以从学习曲线中看到,在前期随机森林的精确度随n_estimators的增大而增大,后面随n_estimators的增大上下波动
bootstrap & oob_score
有放回抽样,自助集平均包含63%的原始数据
bootstrap参数默认为True,代表默认采用这种有放回的抽样技术
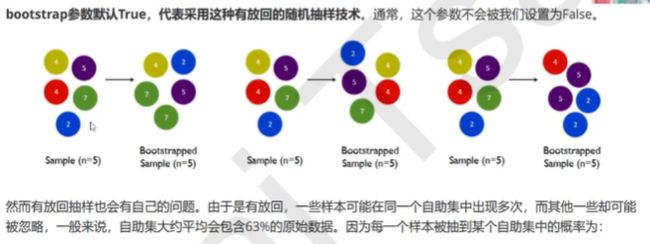


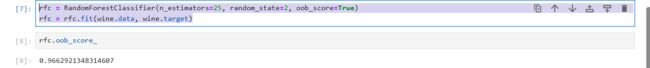
也就是说我们在使用随机森林时,可以不自己设置测试集和训练集,因为有一些数据本身就没有被装进袋中,我们可以用这些数据作为测试集,oob_score可以帮组我们查看袋外数据测试的结果
rfc = RandomForestClassifier(n_estimators=25, random_state=2, oob_score=True)
rfc = rfc.fit(wine.data, wine.target)
也就是说我们在使用随机森林的时候可以自己划分测试集和训练集进行交叉验证,当然我们也可以用袋外数据进行验证
除了有以上比较重要的参数外…feature_importances_这个属性也是有的,接口是用来得到我们输入测试集后得出来的结果的也是用来训练的
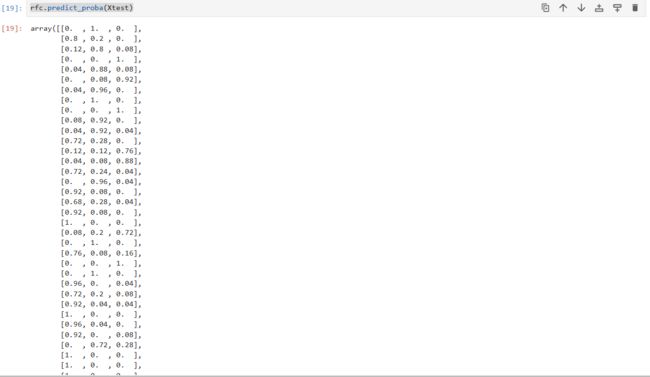
sklearn中随机森林返回的结果是每个标签返回的概率
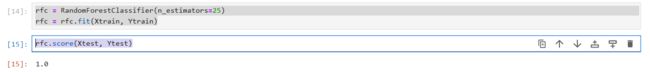
rfc = RandomForestClassifier(n_estimators=25)
rfc = rfc.fit(Xtrain, Ytrain)
rfc.score(Xtest, Ytest)
rfc.apply(Xtest)
rfc.predict(Xtest)
rfc.predict_proba(Xtest)
rfc.predict_proba(Xtest).shape
随机森林回归器
相较于随机森林分类器,随机森林回归器是没有predict_proba这个接口的,因为回归问题并没有某一样本被划分到某个标签的概率
导库
from sklearn.datasets import load_diabetes
from sklearn.model_selection import cross_val_score
from sklearn.ensemble import RandomForestRegressor
建模+交叉验证
regressor = RandomForestRegressor(n_estimators=100
,random_state=0
)
diabets = load_diabetes()
cross_val_score(regressor, diabets.data, diabets.target, cv=10, scoring="neg_mean_squared_error")
随机森林回归填补缺失值案例
sklearn中有一个用于填补缺失值的类
sklearn.impute.SimpleImputer
导库
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import load_diabetes
from sklearn.impute import SimpleImputer
from sklearn.ensemble import RandomForestRegressor
导入数据
dataset = load_diabetes()
dataset.data.shape
备份数据
x_full, y_full = dataset.data, dataset.target
n_samples = x_full.shape[0]
n_features = x_full.shape[1]
添加缺省值
#为完整的数据放入缺失值
#首先确定我们希望放入的缺失数据的比例
rng = np.random.RandomState(0)
missing_rate = 0.5
n_mising_samples = int(np.floor(n_samples * n_features * missing_rate))
# 所有数据要随机遍布在数据集的各行各列中,而一个缺失值会需要一个行索引和一个列索引,所以我们就需要创造这样的数组
missing_features = rng.randint(0, n_features, n_mising_samples)
missing_samples = rng.randint(0, n_samples, n_mising_samples)
# rng.randint(下限,上限, n)的作用是在上限和下限之间取出n个整数
x_missing = x_full.copy()
y_missing = y_full.copy()
x_missing[missing_samples, missing_features] = np.nan
X_missing = pd.DataFrame(x_missing)
from sklearn.impute import SimpleImputer
imp_mean = SimpleImputer(missing_values=np.nan, strategy='mean')
X_missing_mean = imp_mean.fit_transform(X_missing)
X_missing_mean_ = pd.DataFrame(X_missing_mean)
X_missing_mean_
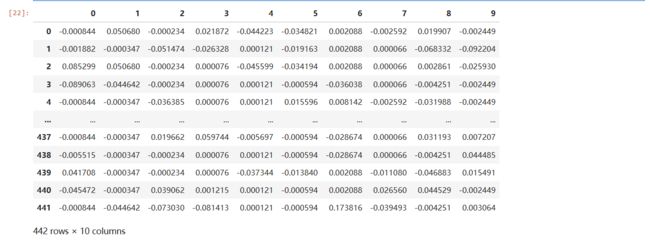
可以看到我们的数据集中的空缺值已经被填补完整了
我们除了通过观察判断是否还有缺失值,还可以已通过info(),或则isnull()
用0来填补缺失值
imp_0 = SimpleImputer(missing_values=np.nan, strategy="constant", fill_value=0)
x_missing_0 = imp_0.fit_transform(x_missing)
x_missing_0_ = pd.DataFrame(x_missing_0)
x_missing_0_
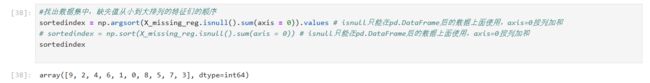
for i in sortedindex:
df = X_missing_reg
#构建新标签
fillc = df.iloc[:, i]
# 构建新特征矩阵,剔除我们要预测的属性列+原始标签
df = pd.concat([df.iloc[:, df.columns != i], pd.DataFrame(y_full)], axis = 1) # 前面是我们要连接的所有的内容,后面的参数是我们希望如何连接,按行还是按列
# 在新特征矩阵里面缺失值用0来填补
df_0 = SimpleImputer(missing_values=np.nan, strategy='constant', fill_value=0).fit_transform(df)
# 找我们的训练集和测试集
Ytrain = fillc[fillc.notnull()]
Ytest = fillc[fillc.isnull()]
Xtrain = df_0[Ytrain.index, :]
Xtest = df_0[Ytest.index, :] # 我们这里不需要Ytest的值因为这些值都是空的我们需要的是Ytest的索引
# 实例化
rfc = RandomForestRegressor(n_estimators=100)
rfc = rfc.fit(Xtrain, Ytrain) # 导入训练集训练
Ypredict = rfc.predict(Xtest) # 用predict接口将Xtest导入得到预测结果
X_missing_reg.loc[X_missing_reg.iloc[:, i].isnull(), i] = Ypredict
对填充好的数据进行建模
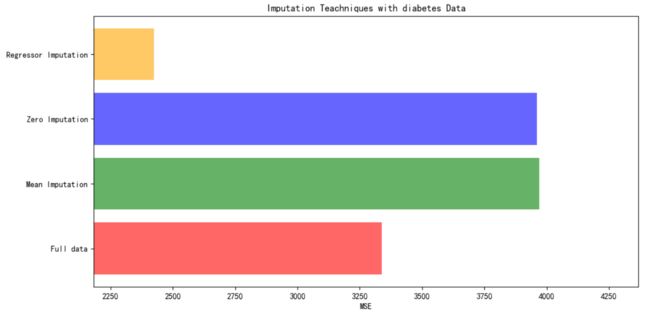
X = [x_full, X_missing_mean, x_missing_0, X_missing_reg]
mse = []
for x in X:
estimator = RandomForestRegressor(n_estimators=100, random_state=0) # 实例化
score_s = cross_val_score(estimator, x, y_full, scoring='neg_mean_squared_error', cv=5).mean()
mse.append(score_s*(-1))
作图
X_labels = ['Full data'
,'Mean Imputation'
,'Zero Imputation'
,'Regressor Imputation'
]
colors = ['r', 'g', 'b', 'orange']
plt.figure(figsize=(12, 6)) # 画出画布
ax = plt.subplot(111) #plt.subplot画出子图
for i in range(len(mse)):
ax.barh(i, mse[i], color = colors[i], alpha=0.6, align='center')
ax.set_title("Imputation Teachniques with diabetes Data")
ax.set_xlim(left=np.min(mse) * 0.9, right=np.max(mse)*1.1)
ax.set_yticks(np.arange(len(mse)))
ax.set_xlabel('MSE')
ax.set_yticklabels(X_labels)
plt.show()
机器学习中调参的基本思想

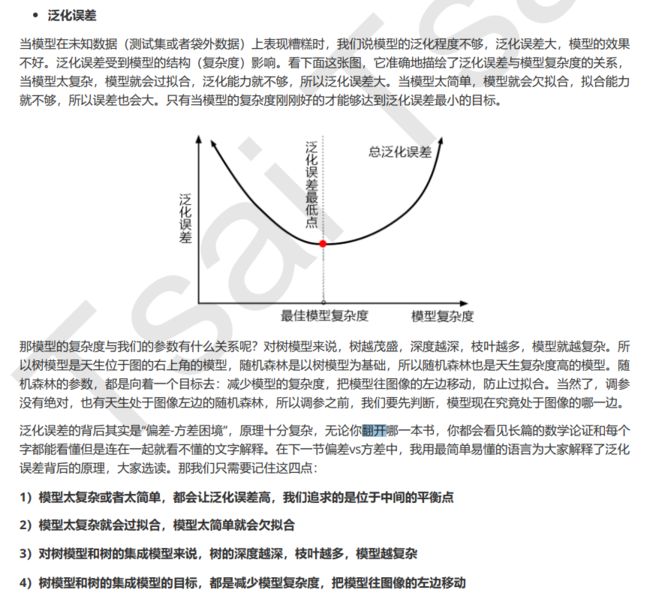
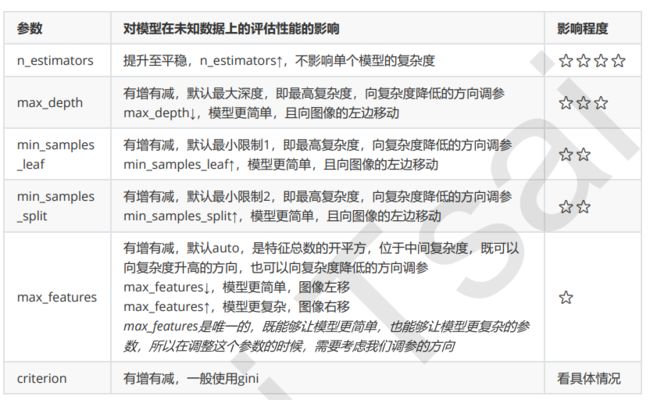
树模型天生比较复杂学习能力比较好很容易过拟合,我们一般都是向模型复杂度低的方向调节
实例随机森林在乳腺癌数据上的调参
导库
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import cross_val_score
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
导入数据集
data = load_breast_cancer()
data.data.shape
rfc = RandomForestClassifier(n_estimators=100, random_state=90)
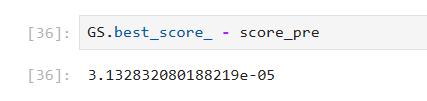
score_pre = cross_val_score(rfc, data.data, data.target, cv=10).mean()
score_pre
随机森林调参第一步:无论如何先调n_estimators
scorel = []
for i in range(0, 200, 10):
rfc = RandomForestClassifier(n_estimators=i+1
,n_jobs=-1
,random_state=90
)
score = cross_val_score(rfc, data.data, data.target, cv=10).mean()
scorel.append(score)
画图
print(max(scorel), scorel.index(max(scorel)) * 10 + 1)
plt.figure(figsize=[20, 5])
plt.plot(range(1, 201, 10), scorel, label='n_estimators学习曲线')
plt.legend()
plt.show()
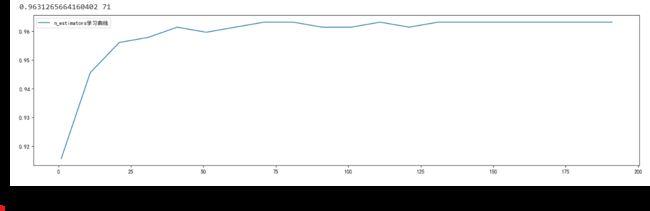
这一步我们只能大致确定一个范围,接下来我们要进一步细化我们的学习曲线
scorel = []
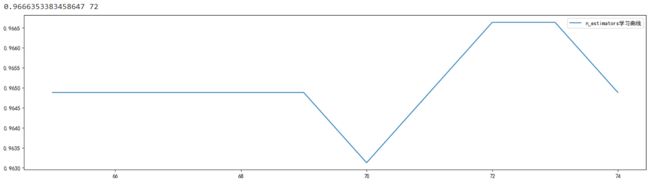
for i in range(65, 75, 1):
rfc = RandomForestClassifier(n_estimators=i+1
,n_jobs=-1
,random_state=90
)
score = cross_val_score(rfc, data.data, data.target, cv=10).mean()
scorel.append(score)
print(max(scorel), [*range(65,75)][scorel.index(max(scorel))])
plt.figure(figsize=[20, 5])
plt.plot(range(65, 75, 1), scorel, label='n_estimators学习曲线')
plt.legend()
plt.show()
调max_depth
# 调整max_features
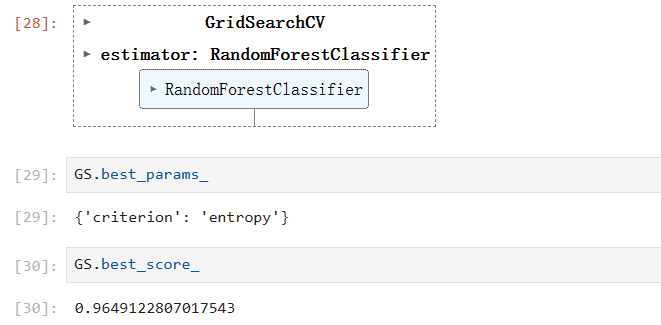
param_grid = {'max_features' : np.arange(5, 30, 1)}
rfc = RandomForestClassifier(n_estimators=72
,random_state=90
)
GS = GridSearchCV(rfc, param_grid, cv=10)
GS.fit(data.data, data.target)
调min_samples_leaf
#调整min_samples_leaf
param_grid={'min_samples_leaf':np.arange(1, 1+10, 1)}
#对于min_samples_split和min_samples_leaf,一般是从他们的最小值开始向上增加10或20
#面对高维度高样本量数据,如果不放心,也可以直接+50,对于大型数据,可能需要200~300的范围
#如果调整的时候发现准确率无论如何都上不来,那可以放心大胆调一个很大的数据,大力限制模型的复杂度
rfc = RandomForestClassifier(n_estimators=39
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)

可以看到我们的得分下降了,说明我们的模型复杂度已经在泛化误差最低点的左边了
调min_samples_split
#调整min_samples_split
param_grid={'min_samples_split':np.arange(2, 2+20, 1)}
rfc = RandomForestClassifier(n_estimators=39
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)

可以看到我们的得分下降了,说明我们的模型复杂度已经在泛化误差最低点的左边了
上面两个参数都是降低随机森林模型复杂度的两个参数,当我们调节他们无法使模型的精确度提高时说明模型复杂度已经在泛化误差最低点的左边了
调criterion
#调整Criterion
param_grid = {'criterion':['gini', 'entropy']}
rfc = RandomForestClassifier(n_estimators=39
,random_state=90
)
GS = GridSearchCV(rfc,param_grid,cv=10)
GS.fit(data.data,data.target)