R语言 人工神经网络(nnet包)
数据
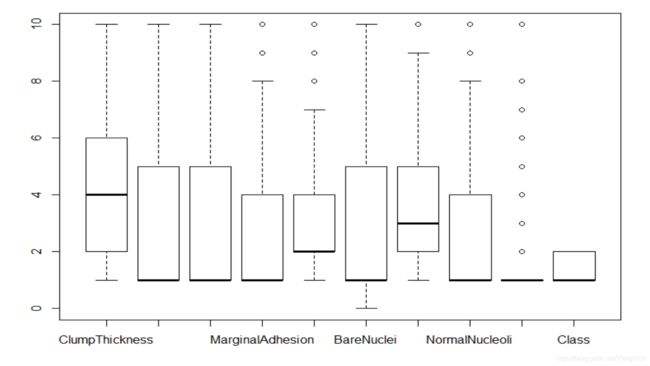
威斯康乳腺癌数据集由699个样本和11个特征组成,第一列为Sample code number (id number),最后一列为Class: (2是良性, 4是恶性),是需要预测的变量。其余几个特征的大小均介于1-10之间。数据可以在UCI的网站上得到。
data <- read.csv("breast-cancer-wisconsin.data")
str(data)
levels(data[,11]) <- c(1,2)
boxplot(data[,-1])
聚类
用kmeans()或hclust()做聚类分析,主要目的是看看数据能不能被分为两类:
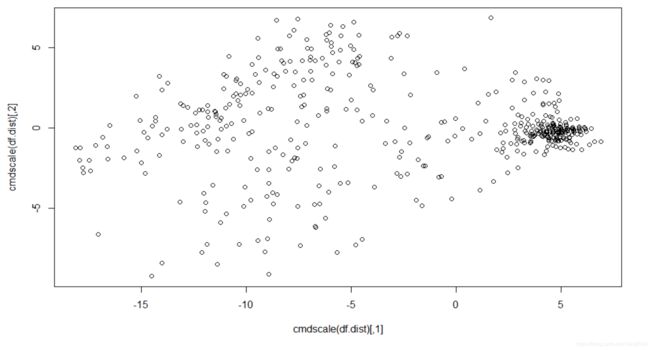
# 计算距离矩阵
df <- data[-c(1,11)] #去掉第一列的id number和最后一列
df.dist<-dist(df)
# 绘制散点图
plot(cmdscale(df.dist))
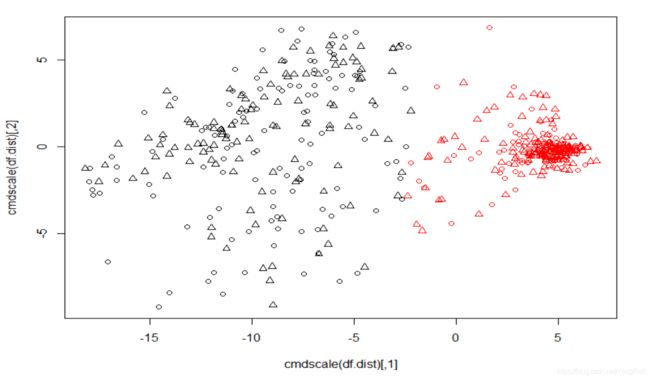
k-means
kcl <- kmeans(df, centers = 2)
table(kcl$cluster)
plot(cmdscale(df.dist),
col = kcl$cluster,
# 聚类预测的类别用不同的形状表示
pch = as.numeric(levels(data[,11])))
# 真实的类别用不同颜色表示
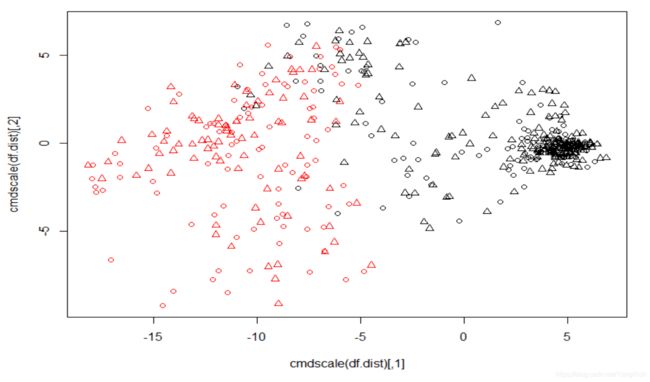
hclust
hcl <- hclust(df.dist)
plot(hcl)
hcutree <- cutree(hcl, k = 2)
table(hcutree)
plot(cmdscale(df.dist),
col = hcutree,
pch = as.numeric(levels(data[,11])))
用nnet()函数建立模型
训练集和测试集
随机将数据集分为训练集和测试集,比例为7:3
train <- sample(nrow(data), 0.7*nrow(data))
data.train <- data[train,-1]
data.test<-data[-train,-1]
建立神经网络模型:
nnet()函数只能建立单隐藏层神经网络,size参数可以确定隐藏层的节点数量,maxit控制迭代次数。在这里我们暂时分别设为4和200,在后面的模型评估中我们会进一步确定这两个超参数的大小。
library(nnet)
nn.output <- matrix(0,nrow(data.train),2)
nn.output[which(data.train[,10]==1),1] <- 1
nn.output[which(data.train[,10]==1),2] <- 0
nn.output[which(data.train[,10]==2),1] <- 0
nn.output[which(data.train[,10]==2),2] <- 2
# 或者 nn.output = class.ind(data.train[,10])
model <- nnet(data.train[,-10],
nn.output,
size = 4, softmax=TRUE, maxit=200, )
summary(model)
# weights: 50
initial value 466.144512
iter 10 value 125.847663
iter 20 value 52.835144
iter 30 value 37.154291
iter 40 value 32.582990
iter 50 value 24.168803
iter 60 value 22.890567
iter 70 value 22.802388
iter 80 value 22.802011
iter 90 value 22.801953
iter 100 value 22.801869
final value 22.801868
converged
a 9-4-2 network with 50 weights
options were - softmax modelling
b->h1 i1->h1 i2->h1 i3->h1 i4->h1 i5->h1
293.24 92.76 -67.92 31.32 69.96 -13.00
i6->h1 i7->h1 i8->h1 i9->h1
-141.57 -80.53 -53.78 -120.86
b->h2 i1->h2 i2->h2 i3->h2 i4->h2 i5->h2
1046.31 -93.73 -59.34 -60.35 -40.37 31.52
i6->h2 i7->h2 i8->h2 i9->h2
-117.63 32.67 -7.86 -54.05
b->h3 i1->h3 i2->h3 i3->h3 i4->h3 i5->h3
-607.51 40.55 -93.22 25.86 29.17 45.55
i6->h3 i7->h3 i8->h3 i9->h3
-20.59 135.62 28.40 216.72
b->h4 i1->h4 i2->h4 i3->h4 i4->h4 i5->h4
404.67 544.41 230.22 161.05 93.63 500.41
i6->h4 i7->h4 i8->h4 i9->h4
77.77 522.08 -19.89 398.93
b->o1 h1->o1 h2->o1 h3->o1 h4->o1
-244.03 0.95 232.22 -228.89 470.29
b->o2 h1->o2 h2->o2 h3->o2 h4->o2
243.56 -1.94 -231.70 228.08 -469.58
模型评估
根据选择的不同超参数评估测试误差:使用不同的超参数建立多个不同的模型,评估并选择其中最优的模型。真实值与学习器预测的结果的差值称为“误差”,在训练集上的误差叫“训练误差”或“经验误差”,在新样本上的叫“泛化误差”,在测试集上的叫“测试误差”,我们使用测试误差作为泛化误差的近似。
可以使用循环,通过改变某参数值,建立多个模型;再针对每一个模型,计算测试误差,然后找到测试误差最小的模型。例如:迭代次数从1到500,每次增加10,建立50个模型,计算每个模型的测试误差,绘制“迭代次数-测试误差”图。
这里我们用k折交叉验证来评估模型。
k-fold Cross-validation
将数据分层采样(即按照需要预测的类别的比例来抽样),分成k个子集,选取其中k-1个子集合并成train set,第k个为test set,进行k次训练和测试,最后取这k次测试的结果的均值。
将数据集划分为k个子集也有多种划分方法,为减小因样本划分不同而导致差别,通常进行p次划分,例如10次10折交叉验证(共100次)。
# 将数据集划分为大小相近的k个子集
cvsamp <- function(k, datasize){
cvlist <- list()
subsetSize <- datasize/k
datarow <- 1:datasize
for (i in 1:k-2) {
group <- sample(datarow, subsetSize)
cvlist <- append(cvlist, list(group))
datarow <- setdiff(datarow, group)
}
cvlist <- append(cvlist, list(datarow))
cvlist <- cvlist[-length(cvlist)]
}
这k个子集的序号分别作为列表储存在cvlist中,结果如下:
k <- 10
cvlist <- cvsamp(k = k,datasize = nrow(data.train))
cvlist
[[1]]
[1] 99 63 422 408 134 35 409 145 20 464 313 489
[13] 105 369 336 230 358 49 112 272 84 427 269 344
[25] 21 406 213 115 423 323 443 360 411 364 479 400
[37] 240 452 198 425 137 15 4 201 450 138 340 88
[[2]]
[1] 236 393 109 118 172 57 383 315 260 428 16 355
[13] 53 273 245 270 206 224 275 174 68 317 67 305
[25] 371 385 353 159 365 18 434 215 160 46 153 7
[37] 51 8 86 14 304 354 319 120 121 488 462 69
[[3]]
[1] 222 368 226 254 314 181 311 97 414 82 401 394
[13] 343 271 307 43 250 281 283 324 308 144 458 439
[25] 436 302 186 72 430 257 293 56 130 435 375 114
[37] 438 234 381 296 165 22 471 142 190 338 298 28
[[4]]
[1] 238 413 194 167 47 188 33 34 40 362 10 472
[13] 207 23 278 276 162 126 280 239 178 419 62 299
[25] 171 325 455 264 465 279 456 111 316 391 108 104
[37] 204 70 485 60 248 149 367 64 321 141 377 85
[[5]]
[1] 96 457 370 45 13 256 11 98 348 255 468 331
[13] 214 146 119 217 129 197 442 378 9 5 297 132
[25] 482 487 58 290 330 163 420 208 263 95 225 447
[37] 417 396 320 259 12 453 231 345 484 136 220 335
[[6]]
[1] 481 93 249 125 52 122 346 61 54 24 152 38
[13] 32 128 161 76 454 26 389 48 110 185 486 66
[25] 351 350 262 285 37 342 150 282 328 90 143 327
[37] 374 173 258 407 19 356 310 55 192 267 177 372
[[7]]
[1] 449 251 241 36 247 133 3 157 116 196 384 349
[13] 27 77 373 39 469 292 405 295 433 135 200 476
[25] 440 155 78 451 480 100 287 470 463 156 113 92
[37] 6 403 347 398 80 286 223 170 75 306 326 189
[[8]]
[1] 366 94 252 380 421 246 474 219 337 341 228 397
[13] 300 426 83 333 148 89 106 402 253 81 101 312
[25] 117 131 233 473 266 205 291 218 277 195 475 329
[37] 103 191 477 59 301 154 102 180 2 182 166 227
[[9]]
[1] 44 139 444 210 216 229 437 65 445 124 30 418
[13] 183 382 140 461 415 322 221 265 334 446 151 29
[25] 459 460 332 274 376 242 268 91 483 71 73 212
[37] 199 432 74 399 209 359 184 357 193 303 169 318
[[10]]
[1] 158 175 164 395 79 203 448 235 467 211 361 441
[13] 412 17 466 261 388 202 294 1 288 424 50 187
[25] 232 147 309 176 390 478 352 431 237 42 363 386
[37] 387 404 429 123 179 41 289 107 339 127 244 243
# 计算k次训练得到的模型的测试误差大小
error <- function(k,hp){
err <- 0
for (i in 1:k){
cvlist <- cvsamp(k = k,datasize = nrow(data.train))
trainset <- data.train[-unlist(cvlist[i]),]
testset <- data.train[unlist(cvlist[i]),]
Y=class.ind(trainset[,10])
ytrue <- testset$Class
model <- nnet(trainset[,-10],Y,softmax=TRUE,
size=hp[1],maxit=hp[2])
Ypred <- predict(model,testset[,-10])
ypred <- max.col(Ypred)
err <- err + sum(ypred != ytrue)/nrow(testset)
}
return(err/k)
}
控制隐藏层节点数:
size <- 1:10
errlist <- vector()
for (i in size){
errlist <- c(errlist, error(k,c(i,200)))
}
plot(size, errlist)
控制迭代次数:
maxit <- seq(1,500,10)
errlist <- vector()
for (i in maxit){
errlist <- c(errlist, error(k,c(4,i)))
}
plot(maxit[2:length(maxit)], errlist[2:length(errlist)],
type = "b")
Leave-one-out
留一法(LOO)是k-fold的特例。当数据大小为N,且k=N时,即为留一法。
好处:不受样本划分方法的影响;比训练集只缺了一个样本,所以得到的模型与训练集训练的模型很相似。
坏处:当模型过大时,花费时长过长,很难实现。
本题中的数据有699个个体,LOO费时稍多,故用k折交叉验证。