绿原酸酯与CALB Docking(2) - Covalent Docking(3upo案例)
目录
- 案例学习 - penicillin G 与 3upo 共价docking
-
- 目标
- 配置要求
- 尝试
-
- 找脚本
- 脚本文件比较
- 分子文件
- 试运行脚本
- 操作
-
- 1. 打开cmd
- 2. 生成ligcovalent.pdb文件
- 3. 生成PDBQT文件
- 4. 生成 flexible 和 rigid PDBQT 文件
- 5. 生成 GPF 和 DPF 文件
- 6. 进行grid and dock
- 7. 查看结果
- 8. 分析
- 命令
- 文件路径
案例学习 - penicillin G 与 3upo 共价docking
参考资料:AutoDock Covalent docking
目标
- 命令行操作进行docking
- 共价docking结构准备
- 柔性docking流程
配置要求
Python >= 2.6
OpenBabel Python bindings
尝试
找脚本
- 首先在虚拟机上安装AutoDock
在官网下载安装包,下载 3-Linux:Inter (64-bit)
uname -r
3.10.0-1127.el7.x86_64
tar -xf autodocksuite-4.2.6-x86_64Linux3.tar
解压之后直接就是了,也不用安装。。。。。。
- 配置python环境用于脚本运行
(这时发现跟虚拟机关系也不大…) 【废废】
D:\Program Files (x86)\MGLTools-1.5.7
该文件夹中有许多看上去有用的东西,包括 python, OpenBabel-2.3.2 等等。
参考本问题:Where can I find the Python scripts for preparing and analysing AutoDock dockings?
在Windows下:
\MGLToolsPckgs\AutoDockTools\Utilities24
通过搜索找到了Utilities24文件夹的位置:
"D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24"
参考问题:Setting up AutoDockTools utility scripts
If you installed MGLTools using one of our latest standalone installers, then you are all set.
D:\Program Files (x86)\MGLTools-1.5.7>python.exe
Python 2.7.11 (v2.7.11:6d1b6a68f775, Dec 5 2015, 20:32:19) [MSC v.1500 32 bit (Intel)] on win32
Type “help”, “copyright”, “credits” or “license” for more information.
python.exe "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_ligand4.py"
prepare_ligand4: ligand filename must be specified.
Usage: prepare_ligand4.py -l filename
(more)
有一个 prepare_covalent_flexres.py 的脚本。
D:\Program Files (x86)\MGLTools-1.5.7>python.exe "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_covalent_flexres.py"
prepare_covalent_flexres: receptor filename must be specified!
Usage: prepare_covalent_flexres.py -r receptor_filename -l ligand_to_superimpose_filename -b atom1_atom2_atom3 -R residue_name
Description of command...
-r receptor_filename
-l ligand_filename
-b ligand atom1_atom2_atom3 to superimpose (eg SD_CG_CB)
-R name of receptor residue onto which the bond atoms
in the ligand will be superimposed
Optional parameters:
[-B] alternate atom1_atom2_atom3 in receptor residue to superimpose
(default uses atom1_atom2_atom3 names specified with -b flag)
[-v] verbose output
[-g pdbqt_filename] (rigid output filename)
[-x pdbqt_filename] (flexible output filename)
[-n pdbqt_filename] (flexible output filename)
NOTE the order of the atoms specified for the bond is crucial:
'-b atom1_atom2_atom3'
specifies this superposition:
LIGAND-atom1-atom2-atom3
atom1-atom2-atom3-RECEPTOR
Process removes atom1-atom2-atom3-RECEPTOR bond and adds bond LIGAND-atom1-atom2-atom3-RECEPTOR
^ ^
# To execute this command type:
# prepare_covalent_flexres.py -l filename -r receptor_filename -l ligand_to_superimpose_filename -b atom1_atom2 -R residue
#specific example:
#python -i prepare_covalent_flexres.py -r 1pwc-protein.pdbqt -l 1pwc-ligand.pdbqt -b OG_CB -R SER62 -g 1pwc_rigid.pdbqt -x 1pwc_flex.pdbqt
下午的任务:
- 比较两个文件是否相同? 看起来有一定的差异?接口上?文件风格上?
- 用prepare_covalent_flexres.py 完成案例的docking.
脚本文件比较
![]()
![]()
"D:\Program Files (x86)\MGLTools-1.5.7/python.exe"
分子文件
先看一下手上有的几个pdb文件:
3upo_protein.pdb 是一个二聚体;与B:SER222进行Docking。
ligand.mol2 是一个小分子,带有H原子,Docking的是一个羧基的C-O(2)-C(1)。
试运行脚本
运行脚本 prepareCovalent.py 时会报错。
报错:
swig_import_helper _mod = imp.load_module(’_openbabel’, fp, pathname, description) ImportError: DLL load failed: 找不到指定的模块
imp 模块为用于实现该import语句的机制提供了一个接口。 imp
参考:
[Open Babel] problems with python libraries
解决:
将OpenBabel-2.3.2的文件夹加入PATH中…
D:\Program Files (x86)\MGLTools-1.5.7\OpenBabel-2.3.2
操作
1. 打开cmd
2. 生成ligcovalent.pdb文件
生成ligcovalent.pdb文件和residues.log文件。
疯狂地输出之后:
==============================
*** Open Babel Warning in OpenBabel::MOL2Format::ReadMolecule
This Mol2 file is non-standard. Problem with molecule: Penicillin Cannot interpret atom types correctly, instead attempting to interpret atom type: H as elements instead.
==============================
*** Open Babel Warning in OpenBabel::OBTypeTable::Translate
Cannot perform atom type translation: table cannot find requested types.
Processing residue B:SER222
[start] output filename is: ligcovalent.pdb
./adcovalent/prepareCovalent.py:76: FutureWarning: comparison toNonewill result in an elementwise object comparison in the future.
if not p2 == None:
==============================
*** Open Babel Warning in OpenBabel::OBGlobalDataBase::Init
Cannot open phmodel.txt defaulting to compiled data.
CHAIN B
Writing output filename: ligcovalent.pdb
生成的ligcovalent.pdb是一个小分子文件,包含了与之共价连接的B:SER222,位置匹配到了B:SER222的位置。
但是原子类型都是XX:
HETATM 1 XX SER B 222 16.639 -66.404 9.375 1.00 0.00 Xx
HETATM 2 XX SER B 222 16.593 -65.987 8.225 1.00 0.00 Xx
HETATM 3 XX SER B 222 16.170 -65.510 10.505 1.00 0.00 Xx
HETATM 4 XX SER B 222 16.380 -66.153 11.914 1.00 0.00 Xx
HETATM 5 XX SER B 222 15.781 -65.371 13.011 1.00 0.00 Xx
HETATM 6 XX SER B 222 16.692 -64.263 13.298 1.00 0.00 Xx
HETATM 7 XX SER B 222 18.107 -64.880 13.455 1.00 0.00 Xx
HETATM 8 XX SER B 222 18.183 -66.251 12.242 1.00 0.00 Xx
HETATM 9 XX SER B 222 18.350 -65.486 14.853 1.00 0.00 Xx
HETATM 10 XX SER B 222 19.192 -63.837 13.139 1.00 0.00 Xx
HETATM 11 XX SER B 222 16.227 -63.426 14.474 1.00 0.00 Xx
HETATM 12 XX SER B 222 16.846 -62.425 14.797 1.00 0.00 Xx
HETATM 13 XX SER B 222 15.120 -63.769 15.162 1.00 0.00 Xx
HETATM 14 XX SER B 222 14.786 -65.113 10.278 1.00 0.00 Xx
HETATM 15 XX SER B 222 13.741 -65.985 10.209 1.00 0.00 Xx
HETATM 16 XX SER B 222 13.921 -67.187 10.343 1.00 0.00 Xx
HETATM 17 XX SER B 222 12.357 -65.438 9.939 1.00 0.00 Xx
HETATM 18 XX SER B 222 11.278 -66.492 9.870 1.00 0.00 Xx
HETATM 19 XX SER B 222 10.966 -67.265 11.001 1.00 0.00 Xx
HETATM 20 XX SER B 222 9.954 -68.228 10.936 1.00 0.00 Xx
HETATM 21 XX SER B 222 9.249 -68.426 9.745 1.00 0.00 Xx
HETATM 22 XX SER B 222 9.554 -67.660 8.616 1.00 0.00 Xx
HETATM 23 XX SER B 222 10.563 -66.694 8.677 1.00 0.00 Xx
HETATM 24 N SER B 222 19.175 -69.711 10.030 1.00 0.00 N1+
HETATM 25 CA SER B 222 17.945 -69.880 9.243 1.00 0.00 C
HETATM 26 C SER B 222 17.931 -70.976 8.176 1.00 0.00 C
HETATM 27 O SER B 222 16.841 -71.389 7.759 1.00 0.00 O
HETATM 28 CB SER B 222 17.496 -68.565 8.618 1.00 0.00 C
HETATM 29 OG SER B 222 17.087 -67.662 9.631 1.00 0.00 O
HETATM 30 H SER B 222 19.447 -70.608 10.433 1.00 0.00 H
HETATM 31 H SER B 222 19.010 -69.038 10.778 1.00 0.00 H
HETATM 32 H SER B 222 19.922 -69.370 9.425 1.00 0.00 H
HETATM 33 H SER B 222 17.254 -70.219 9.986 1.00 0.00 H
HETATM 34 H SER B 222 18.468 -70.599 7.331 1.00 0.00 H
HETATM 35 H SER B 222 18.357 -71.824 8.671 1.00 0.00 H
HETATM 36 H SER B 222 16.382 -70.674 7.291 1.00 0.00 H
HETATM 37 H SER B 222 16.676 -68.749 7.955 1.00 0.00 H
HETATM 38 H SER B 222 18.313 -68.137 8.075 1.00 0.00 H
HETATM 39 H SER B 222 16.347 -68.046 10.126 1.00 0.00 H
与output文件夹里对比发现确实不对…
原文件中有不标准的原子类型名,因此经过GaussView将其转化。
将 ligand.mol2 文件载入GaussView中又保存得到ligand_v.mol2文件,进行尝试,也是在疯狂地跳atom type error,但是得到的文件是正确的。
3. 生成PDBQT文件
生成 protein.pdbqt 文件和 ligcovalent.pdbqt文件。
会报错:
swig/python detected a memory leak of type ‘BHtree *’, no destructor found.
但是对结果似乎没有影响。
4. 生成 flexible 和 rigid PDBQT 文件
理论上应该生成 3upo_protein_rigid.pdbqt 和 3upo_protein_flex.pdbqt
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_flexreceptor4.py" -r 3upo_protein.pdbqt -s B:SER222
指定一个错误的-s 可以生成文件,但是没有内容。 【先跳过这一步】
生成了 “ligcovalent_rigid.pdbqt” and “ligcovalent_flex.pdbqt”
5. 生成 GPF 和 DPF 文件
生成了 3upo_protein.gpf
输出命令:
!unable to use ligand.TORSDOF! torsdof always set to ligand.ndihe= 9
swig/python detected a memory leak of type ‘BHtree *’, no destructor found.
生成了 ligcovalent_3upo_protein.dpf,该文件为文本文件,可以修改。
copy nul empty # 生成 empty 空文件
6. 进行grid and dock
D:\Desktop\adt\autodock4.exe: I’m sorry; I can’t find or open “‘empty’”
windows 创建、生成0字节空文件
copy nul empty # 生成 empty 空文件
dock生成 ligcovalent_3upo_protein.dlg 文件。
7. 查看结果
载入 3upo_protien.pdbqt
载入ligcovalent.pdbqt 【不必要】
Analyze -> Dockings -> Open .dlg 文件
报错:
ERROR *********************************************
Traceback (most recent call last):
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\ViewerFramework\VF.py", line 941, in tryto
result = command( *args, **kw )
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\autoanalyzeCommands.py", line 3860, in doit
d.readDlg(dlgFile)
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\Docking.py", line 96, in readDlg
dlo = DockingLogObject(self, dlgFile)
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\Docking.py", line 728, in __init__
parser.parse(filename)
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\DlgParser.py", line 121, in parse
self.match(allLines)
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\DlgParser.py", line 236, in match
apply(d['func'], (lines,), {})
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\DlgParser.py", line 468, in getClusterInfo
self.getClusterRecord(cl)
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\DlgParser.py", line 490, in getClusterRecord
newList2 = map(lambda x:float(x),ll[3:-1])
File "D:\Program Files (x86)\MGLTools-1.5.7\lib\site-packages\AutoDockTools\DlgParser.py", line 490, in <lambda>
newList2 = map(lambda x:float(x),ll[3:-1])
ValueError: invalid literal for float(): -1.#J
在文件中搜索 “-1.#J” 在计算RMSD的时候出现了这个,全部替换成 “-nan”.
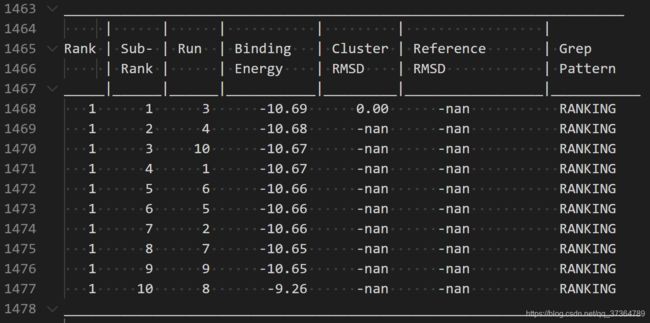
成功读入了结构。
将所有的conformation Build All.
将前三个结构保存为PDB文件,载入VMD中进行查看。
将蛋白的结构保存为pdb文件:protein.pdb,相比3upo_protein.pdb,该结构在于其带有极性氢。便于我们分析氢键的形成。
8. 分析
载入文件:
dir *.pdb # 查看后缀为.pdb的文件
2015/09/10 07:47 523,584 3upo_protein.pdb
2021/05/12 15:59 261,872 complex.pdb
2021/05/12 15:24 2,423 empty_conf_0.pdb
2021/05/12 15:25 2,423 empty_conf_1.pdb
2021/05/12 15:25 2,423 empty_conf_2.pdb
2021/05/11 15:24 9,795 ligcovalent.pdb
2021/05/12 16:10 650,609 protein.pdb
其中,complex.pdb 是由 protein.pdb 的chain B 和empty_conf_0.pdb结构组合而成的。将小分子配体加在后面。
修改原子序数和resid?
(似乎怎么修改都不太行,因为原子位置有冲突,导致 New Cartoon 识别不正确。) >> 只能开一个protein.pdb提供New Cartoon的显示。
将residue name (resname)修改为 LIG.
ligcovalent.pdb的结构与3upo_protein.pdb的结构有collapse。(ligcovalent.pdb中带有非极性氢)
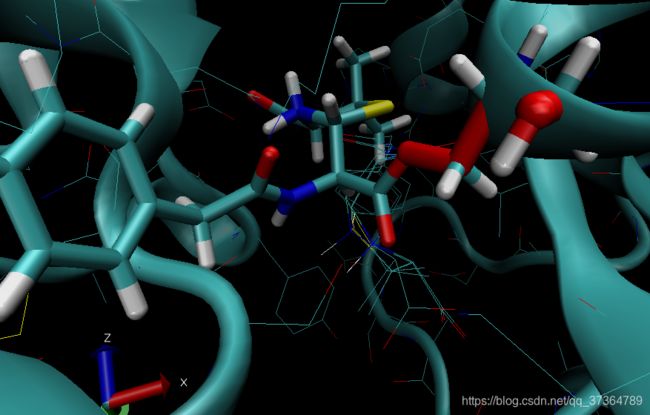
排名靠前的几个conformation具有相似性:
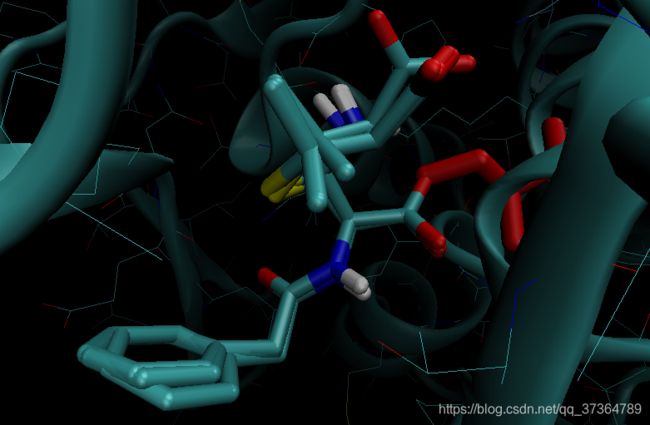
分析conformation = 0 的结构的氢键。
载入complex.pdb.
氢键显示出来了:
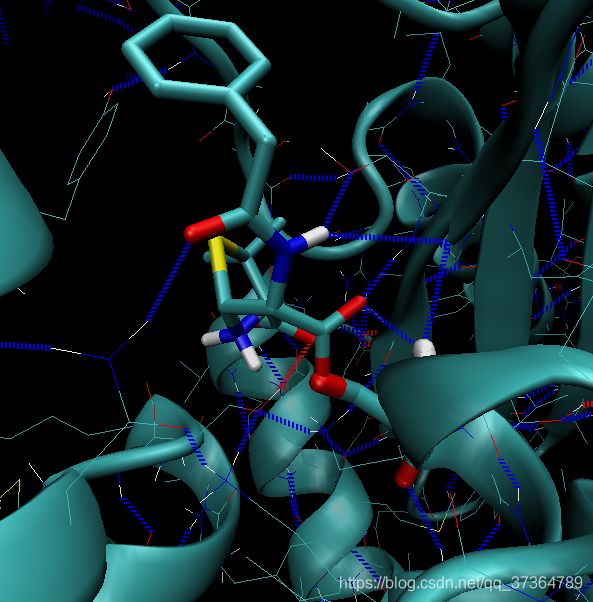
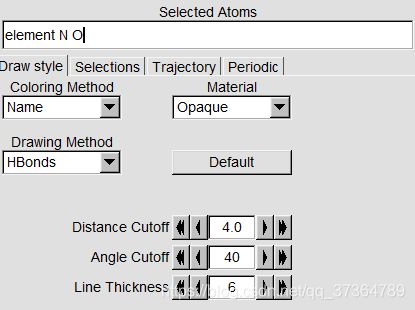
其设置为:(参考:VMD tutorial)
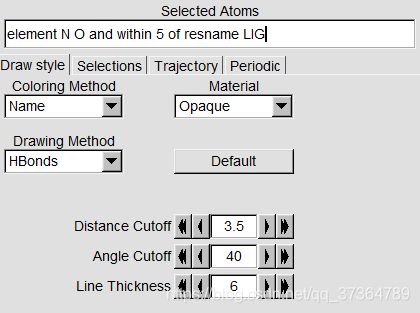
显示lig附近的氢键:

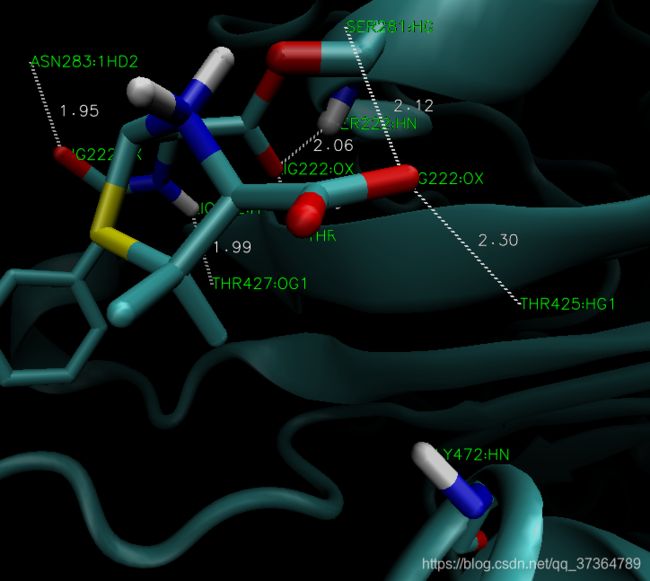
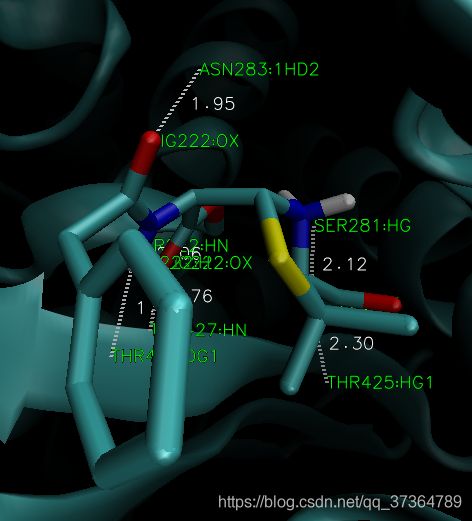
得到6个氢键,做 label。
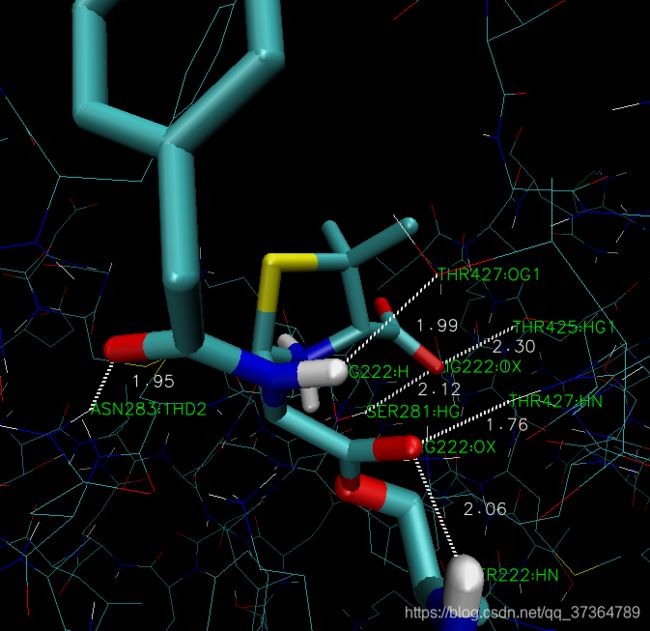
噻唑烷(Thiazolidine | C3H7NS )
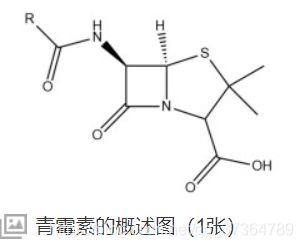
THR427:HN LIG222:OX
SER222:HN LIG222:OX
与噻唑烷末端的羧基形成氢键的:
SER281:HG LIG222:OX
THR425:HG – LIG222:OX
与苯环前的酰胺形成氢键的
THR427:OG LIG222:H
ASN283:1HD LIG222:OX
3UPO
Structure of penicillin-binding protein A from M. tuberculosis: penicillin G acyl-enzyme complex
对接的结果与实测的Complex结构基本一致。
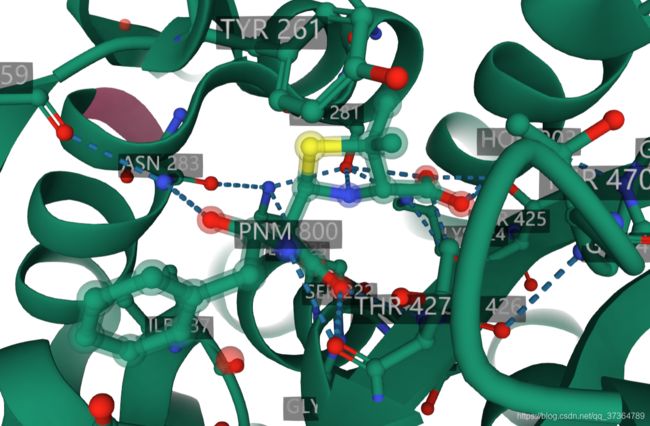

令人注意的是羧基左上侧的水分子。
参考文献:https://doi.org/10.1016/j.jmb.2012.02.021
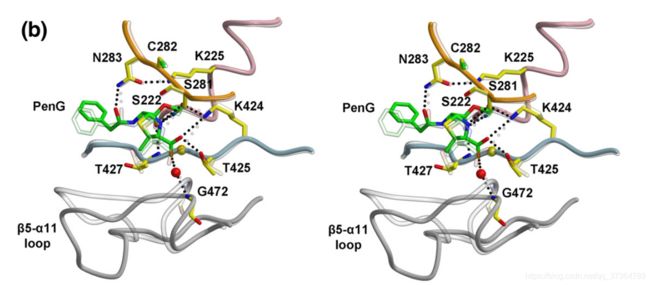
Fig6.(B) Docking结果与之基本吻合:与苯基前的酰胺形成氢键的残基:N283;与噻唑烷末端羧基形成氢键:K424/T425/通过水分子介导的G472。
THR427:HN LIG222:OX ----- T427
SER222:HN LIG222:OX -----
SER281:HG LIG222:OX -----
THR427:OG LIG222:H -----
ASN283:1HD LIG222:OX -----
THR425:HG – LIG222:OX -----
命令
命令行操作如下:
D:
cd D:\Desktop\adt\adCovalentDockResidue
# 利用Gauss重新生成一下 ligand.mol2,生成ligcovalent.pdb
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" ./adcovalent/prepareCovalent.py --ligand ./3upo_test/ligand.mol2 --ligindices 1,2 --receptor ./3upo_test/3upo_protein.pdb --residue B:SER222 --outputfile ligcovalent.pdb
# 生成 PDBQT 文件
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_receptor4.py" -r ./3upo_test/3upo_protein.pdb -A hydrogens
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_receptor4.py" -r ligcovalent.pdb
# 生成 flexible/rigid PDBQT 文件
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_flexreceptor4.py" -r 3upo_protein.pdbqt -s 3upo_protein:B:SER222
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_flexreceptor4.py" -r ligcovalent.pdbqt -s ligcovalent:B:SER222
# 生成 gpf 和 dpf 文件
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_gpf4.py" -r 3upo_protein_rigid.pdbqt -x ligcovalent_flex.pdbqt -l ligcovalent_flex.pdbqt -y -I 20 -o 3upo_protein.gpf
"D:\Program Files (x86)\MGLTools-1.5.7\python.exe" "D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24\prepare_dpf4.py" -r 3upo_protein_rigid.pdbqt -x ligcovalent_flex.pdbqt -l ligcovalent_flex.pdbqt -o ligcovalent_3upo_protein.dpf -p move=empty
# 编辑 ligcovalent_3upo_protein.dpf
# unbound_energy 0.0 # 修改原参数
copy nul empty # 生成 empty 空文件
# 运行 AutoGrid and AutoDock
D:\Desktop\adt\autogrid4.exe -p 3upo_protein.gpf -l 3upo_protein.glg
D:\Desktop\adt\autodock4.exe -p ligcovalent_3upo_protein.dpf -l ligcovalent_3upo_protein.dlg
# 打开 AutoDockTools 查看结果
文件路径
将几个脚本的路径粘贴在下面:
D:\Desktop\adt\adCovalentDockResidue\adcovalent
D:\Desktop\adt\adCovalentDockResidue\3upo_test
“D:\Program Files (x86)\MGLTools-1.5.7\Lib\site-packages\AutoDockTools\Utilities24”
“D:\Program Files (x86)\MGLTools-1.5.7\python.exe”
D:\Desktop\adt