图文详解 VCF 生信格式 (变异信息)
文章目录
- 一、vcf 格式介绍
- 二、vcf 资源文件
- 三、vcf 文件详解
-
- 3.1 主要字段
- 3.2 INFO 中的常见信息
- 3.3 FORMAT 和 SAMPLEs 中的信息
- 四、vcf 的记录模式
-
- 4.1 只记录变异本身的信息
- 4.2 记录个体或个体组织的变异信息
- 4.3 记录群体或家系的变异信息
- 五、记录标准
-
- 5.1 记录多核苷酸多样性 (multi nucleotide polymorphism,MNP)
- 5.2 记录缺失或插入 (INDEL)
- 5.3 记录结构变异 (SV)
- 六、举例说明
一、vcf 格式介绍
vcf (Variant Call Format)是一种用于存储基因组序列中的变异信息
-
一般用在 单核苷酸变异(SNV),小片段插入缺失(INDEL)等
-
也用于 拷贝数变异(CNV),SV(结构变异)等
-
SNV:参考基因组在1号染色体7845190为 C,但检测样本在同样位置为 A
-
INDEL:包含插入和缺失两种
-
- Insertion:参考基因组某片段为 ACTTG,但是检测样本同样位置为 ACCCTTG,插入了CC
- Deletion:参考基因组某片段为 TTCGG,但是检测样本同样位置为 TTGG,缺失 C
二、vcf 资源文件
这里的示例文件来自 1000 Genomes 计划,数据主要为不同地区人类变异数据。
数据来源:https://www.internationalgenome.org/
数据下载:https://s3.amazonaws.com/1000genomes/release/20130502/ALL.wgs.phase3_shapeit2_mvncall_integrated_v5b.20130502.sites.vcf.gz
或者https://ftp.1000genomes.ebi.ac.uk//vol1/ftp/technical/reference/phase2_reference_assembly_sequence/hs37d5.fa.gz
数据示例
#CHROM POS ID REF ALT QUAL FILTER INFO
1 10177 rs367896724 A AC 100 PASS AC=2130;AF=0.425319;AN=5008;NS=2504;DP=103152;EAS_AF=0.3363;AMR_AF=0.3602;AFR_AF=0.4909;EUR_AF=0.4056;SAS_AF=0.4949;AA=|||unknown(NO_COVERAGE);VT=INDEL
1 10235 rs540431307 T TA 100 PASS AC=6;AF=0.00119808;AN=5008;NS=2504;DP=78015;EAS_AF=0;AMR_AF=0.0014;AFR_AF=0;EUR_AF=0;SAS_AF=0.0051;AA=|||unknown(NO_COVERAGE);VT=INDEL
1 10352 rs555500075 T TA 100 PASS AC=2191;AF=0.4375;AN=5008;NS=2504;DP=88915;EAS_AF=0.4306;AMR_AF=0.4107;AFR_AF=0.4788;EUR_AF=0.4264;SAS_AF=0.4192;AA=|||unknown(NO_COVERAGE);VT=INDEL
1 10505 rs548419688 A T 100 PASS AC=1;AF=0.000199681;AN=5008;NS=2504;DP=9632;EAS_AF=0;AMR_AF=0;AFR_AF=0.0008;EUR_AF=0;SAS_AF=0;AA=.|||;VT=SNP
1 10506 rs568405545 C G 100 PASS AC=1;AF=0.000199681;AN=5008;NS=2504;DP=9676;EAS_AF=0;AMR_AF=0;AFR_AF=0.0008;EUR_AF=0;SAS_AF=0;AA=.|||;VT=SNP
1 10511 rs534229142 G A 100 PASS AC=1;AF=0.000199681;AN=5008;NS=2504;DP=9869;EAS_AF=0;AMR_AF=0.0014;AFR_AF=0;EUR_AF=0;SAS_AF=0;AA=.|||;VT=SNP
1 10539 rs537182016 C A 100 PASS AC=3;AF=0.000599042;AN=5008;NS=2504;DP=9203;EAS_AF=0;AMR_AF=0.0014;AFR_AF=0;EUR_AF=0.001;SAS_AF=0.001;AA=.|||;VT=SNP
1 10542 rs572818783 C T 100 PASS AC=1;AF=0.000199681;AN=5008;NS=2504;DP=9007;EAS_AF=0.001;AMR_AF=0;AFR_AF=0;EUR_AF=0;SAS_AF=0;AA=.|||;VT=SNP
1 10579 rs538322974 C A 100 PASS AC=1;AF=0.000199681;AN=5008;NS=2504;DP=5502;EAS_AF=0;AMR_AF=0;AFR_AF=0.0008;EUR_AF=0;SAS_AF=0;AA=.|||;VT=SNP
1 10616 rs376342519 CCGCCGTTGCAAAGGCGCGCCG C 100 PASS AC=4973;AF=0.993011;AN=5008;NS=2504;DP=2365;EAS_AF=0.9911;AMR_AF=0.9957;AFR_AF=0.9894;EUR_AF=0.994;SAS_AF=0.9969;VT=INDEL
1 10642 rs558604819 G A 100 PASS AC=21;AF=0.00419329;AN=5008;NS=2504;DP=1360;EAS_AF=0.003;AMR_AF=0.0014;AFR_AF=0.0129;EUR_AF=0;SAS_AF=0;AA=.|||;VT=SNP
1 11008 rs575272151 C G 100 PASS AC=441;AF=0.0880591;AN=5008;NS=2504;DP=2232;EAS_AF=0.0367;AMR_AF=0.0965;AFR_AF=0.1346;EUR_AF=0.0885;SAS_AF=0.0716;AA=.|||;VT=SNP
1 11012 rs544419019 C G 100 PASS AC=441;AF=0.0880591;AN=5008;NS=2504;DP=2090;EAS_AF=0.0367;AMR_AF=0.0965;AFR_AF=0.1346;EUR_AF=0.0885;SAS_AF=0.0716;AA=.|||;VT=SNP
三、vcf 文件详解
文件一般包含两部分:
- 注释信息(header):位于文件开始,每行以
#开始 - 变异信息(body):没有
#即为记录的变异信息
3.1 主要字段
黑体字为必选字段
| 字段 | 描述 | 举例 |
|---|---|---|
| CHROM | 染色体号,注意不需要前缀 chr |
1 |
| POS | 变异位点,对于 INDEL,为 INDEL 的第一个碱基位点 | 10616 |
| ID | dbSNP 的编号,.为置空 |
rs376342519 |
| REF | 参考基因组的碱基,也就是等位基因 | CCGCCGTTGCAAAGGCGCGCCG |
| ALT | 检测样本的碱基,同一位置有多个则以,分隔 |
C |
| QUAL | Phred 的质量值,表示改位点存在变异的可能性。 分数越高则认为越可靠,但同时需要考虑测序深度,覆盖度等因素。.代表置空,不代表质量值为 0。 |
100 |
| FILTER | 过滤标志,如果为 PASS则认为是一个变异 |
PASS |
| INFO | 详细信息,用 key=value的格式来表示。key 一般是简写,具体描述在文件开头的 header lines 中显示。 |
AC=4973;AF=0.993011;AN=5008;VT=INDEL |
| FORMAT | 可选,变异位点格式,包括 GT,AD,DP,GQ,PL/ GT,AD,DP,GQ,PGT,PID,PL,PS | GT:DP:GQ:PL |
| SAMPLEs | 可选,各个样本的值,来自 BAM 文件 @RG 的 SM 标签。一般每个样本对应一列,因此该文件会超过十列。每个样本会与 FORMAT 列的格式一一对应,不同格式用 :分隔 |
0/1:50:99:0,20,200 |
3.2 INFO 中的常见信息
| 字段 | 全称 | 描述 | 举例 |
|---|---|---|---|
| AA | Ancestral Allele | 一个群体或物种的共同祖先中存在的该等位基因 | AA=A |
| AC | Allele Count | 该变异的等位基因(ALT列)在样本集合中出现的次数。如果有多个 ALT,使用 ,分隔 |
AC=4973 |
| AF | Alternate Allele Frequency | 该变异在样本集合中的频率。对于 1000 Genomes 来说,EAS_AF,AMR_AF,AFR_AF,EUR_AF, SAS_AF 分别表示东亚,美洲,非洲,欧洲,南亚人群的等位基因频率 | AF=0.993011 |
| AN | Allele Number | 该变异的等位基因总数。以二倍体生物为例,如果样本为杂合子(基因型 0/1),AN 值为 1,表示改位点只有一个等位基因发生突变。如果样本为纯合子(基因型 1/1),AN 值为 2 | AN=5008 |
| DP | Read Depth | 该变异位点测序深度,也就是改位点 reads 覆盖度 | DP=2365 |
| MQ | Mapping Quality | 该变异比对时,reads 的平均质量 | MQ=100 |
| QD | Quality by Depth | 该变异质量分数(QUAL)与测序深度(DP)的比值。用于评估改位点的质量。 | QD=0.12 |
| VT | Variant Type | 变异类型,一般包括 SNP,MNP,INDEL,SV 等 | VT=INDEL |
MAF(minor allele frequency)次要等位基因频率
这个测量可以用来粗略地了解给定人群中给定SNP的基因型变异,换句话说,它告诉你这个SNP有多普遍
EAF(effect allele frequency)效应等位基因频率
它本质上是等位基因,其与疾病的关系正在被研究。因此,效应等位基因总是次要等位基因。
3.3 FORMAT 和 SAMPLEs 中的信息
| 字段 | 全称 | 描述 |
|---|---|---|
| GT | Genotype | 表示基因型。对于二倍体样本,用两个数字中间以 /或 ` |
| AD | Allele Depth | 样本中等位基因的 reads 覆盖度。对于二倍体,1000,1100用逗号分隔,前者是 REF,后者是 ALT |
| DP | Read Depth | 该位点 reads 覆盖度 |
| GQ | Genotype Quality | 基因型的质量值,表示该基因型的可能性,值越高,可能性越大。计算:Phred 值=-10log10§,p为基因型错误的概率 |
| PL | Provieds the Likelihoods of the given genotypes | 三种基因型的质量值,即0/0,0/1,1/1,三种基因型的概率总和为1。值越小表示是该基因型的概率越大。同样是计算 Phred 值,但是 p 为基因型存在的概率。 |
| PGT | Phased Genotype | 只出现在进行过相分离的样本中。表示相分离后的基因型,两个数字间使用 ` |
| PID | Phase ID | 描述基因型相位的标识符。 |
| PS | Phase Set | 描述同一样本中基因型相位的信息。 |
相位化(phasing)是确定某个个体在某个基因位点所携带的等位基因来自哪个亲本的过程。
GT 字段中的 /表示基因型未相位化,表示我们不确定哪个等位基因来自父亲或母亲。
GT 字段中的 |表示基因型相位化,也就是说可以确定等位基因的来源亲本。
四、vcf 的记录模式
VCF 文件可以记录不同级别的变异信息,从单一变异到个体、组织、群体或家系的变异。
4.1 只记录变异本身的信息
通常用于描述特定变异的特征,不涉及特定个体或群体的信息。
#CHROM POS ID REF ALT QUAL FILTER INFO
1 69511 rs75062661 G A 99 PASS AC=1;AF=0.0002;AN=5008;NS=2504;DP=2184;EAS_AF=0;AMR_AF=0.0008;AFR_AF=0;EUR_AF=0.001;SAS_AF=0.0007;VT=SNP
4.2 记录个体或个体组织的变异信息
在VCF文件的末尾通常会有一个或多个样本列,其中每一列都代表一个个体或个体的某个组织。
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT sample1
1 899282 rs123456 A G,T 50 PASS AC=2;AF=0.5;AN=4;NS=1;DP=100 GT:DP:GQ:PL 0/1:50:99:0,20,200
4.3 记录群体或家系的变异信息
包括多个样本的数据,可以用于群体遗传学分析。
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT Person1 Person2 Person3
1 945874 rs7891011 A G 99 PASS AC=3;AF=0.75;AN=4;NS=3;DP=300 GT:DP:GQ:PL 0/1:100:99:0,20,200 1/1:100:99:0,0,100 0/0:100:99:0,0,0
1000 genomes 比较特殊,不同人群的等位基因频率在 INFO 中以不同的字段表示
1 10177 rs367896724 A AC 100 PASS AC=2130;AF=0.425319;AN=5008;NS=2504;DP=103152;EAS_AF=0.3363;AMR_AF=0.3602;AFR_AF=0.4909;EUR_AF=0.4056;SAS_AF=0.4949;AA=|||unknown(NO_COVERAGE);VT=INDEL
五、记录标准
注意,这个标准并不是 vcf 规范中要求的,但是为了后续样本合并和解读,最好遵循以下标准。
GATK 和 bcftools 都提供了相应的标准化工具。
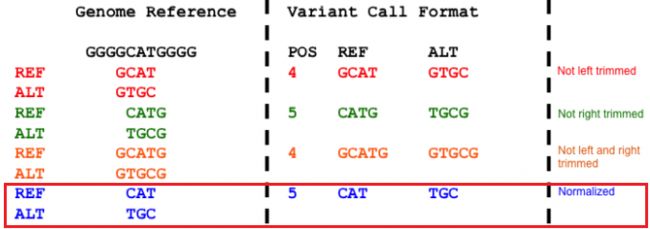
5.1 记录多核苷酸多样性 (multi nucleotide polymorphism,MNP)
REF: GGGCATGGG
ALT: GGGTGCGGG
有下面四种表示方式:
左边是参考基因组(REF)和检测样本(ALT)间比较,一种颜色是一种记录方式。
右边是在 vcf 文件中的表示方式。
那么怎样用尽可能少的核苷酸表示变异,减少冗余的记录。
前三种无论在左右都包含了同样多余的信息,显然,最后一种只包含变异位点的最合适
一个 vcf 中的例子:
12 6608369 ss1388023103 CTTTCTTTCT ATTTCTTTCT 100 PASS AC=2;AF=0.000399361;AN=5008;NS=2504;DP=18116;EAS_AF=0;AMR_AF=0;AFR_AF=0;EUR_AF=0.002;SAS_AF=0;VT=MNP
5.2 记录缺失或插入 (INDEL)
REF: GGGCACACACAGGG
ALT: GGGCACACAGGG
保持所有等位位点的长度不变,而且变异位置不能再向左移动,同样是为了减少冗余的记录。
最后一种是既可以锚定缺失位点,又可以展示 deletion
在 vcf 文件中的 deletion
1 43098430 rs534335349 TC T 100 PASS AC=3;AF=0.000599042;AN=5008;NS=2504;DP=18120;EAS_AF=0.001;AMR_AF=0;AFR_AF=0.0015;EUR_AF=0;SAS_AF=0;AA=?|C|-|unsure;VT=INDEL
在 vcf 文件中的 Insertion
12 6607940 rs150221708 A AT 100 PASS AC=695;AF=0.138778;AN=5008;NS=2504;DP=16205;EAS_AF=0.0089;AMR_AF=0.1412;AFR_AF=0.1392;EUR_AF=0.2097;SAS_AF=0.1973;AA=TTTTTT|TTTTTT|TTTTTTT|insertion;VT=INDEL
5.3 记录结构变异 (SV)
复杂到离谱,当然还有 bedpe 文件格式可以作为替代方案。
| REF | ALT | 描述 |
|---|---|---|
| s | t[p[ | 片段p在以从右侧开始的顺序,从t所在位置取代了s |
| s | t]p] | 片段 p 反转后(从左侧),从t所在位置取代s |
| s | ]p]t | 片段p在以从右侧开始的顺序,在t之前的位置取代s |
| s | [p[t | 片段 p 反转后(从左侧),从t之前位置取代s |
六、举例说明
这一行来自文章开始的数据
1 10616 rs376342519 CCGCCGTTGCAAAGGCGCGCCG C 100 PASS AC=4973;AF=0.993011;AN=5008;NS=2504;DP=2365;EAS_AF=0.9911;AMR_AF=0.9957;AFR_AF=0.9894;EUR_AF=0.994;SAS_AF=0.9969;VT=INDEL
逐个说明:
-
CHROM: 1 - 变异发生在第1号染色体。
-
POS: 10616 - 变异发生的位置是染色体的第10616个碱基。
-
ID: rs376342519 - 变异的参考数据库ID,这里是dbSNP数据库中的ID。
-
REF: CCGCCGTTGCAAAGGCGCGCCG - 参考基因组序列在这个位置的碱基序列。
-
ALT: C - 变异后的碱基序列,这里显示为单个碱基C,表示相对于参考序列,其余的部分(CGCCGTTGCAAAGGCGCGCCG)被删除了。
-
QUAL: 100 - 变异检测的质量分数,100 代表质量是高的。
-
FILTER: PASS - 变异通过了质量控制。
-
INFO字段:
-
- AC: 4973 - 等位基因数(Allele Count),表示在所有样本中有4973个这样的变异等位基因。
- AF: 0.993011 - 等位基因频率(Allele Frequency),表示在所有检测的等位基因中,几乎所有的(大约99.3%)都是这个变异等位基因。
- AN: 5008 - 等位基因总数(Allele Number),表示所有样本中等位基因的总数。
- NS: 2504 - 样本数量(Number of Samples),表示有2504个样本被用来检测这个变异。
- DP: 2365 - 测序深度(Depth of Coverage),表示所有样本中变异位置上的测序深度。
- 各人群等位基因频率(Allele Frequency by Population):
-
-
- EAS_AF: 0.9911 - 东亚人群中的等位基因频率。
- AMR_AF: 0.9957 - 美洲人群中的等位基因频率。
- AFR_AF: 0.9894 - 非洲人群中的等位基因频率。
- EUR_AF: 0.994 - 欧洲人群中的等位基因频率。
- SAS_AF: 0.9969 - 南亚人群中的等位基因频率。
-
-
VT: INDEL - 变异类型(Variant Type),这里表示是一个插入/删除事件。
综合来看,这行记录表明在第1号染色体上有一个非常常见的INDEL变异,在不同人群中频率都非常高,几乎接近于固定。也就是说,这个变异在样本集合中广泛存在。
这里有个连连看的小游戏帮助大家理解
https://www.ebi.ac.uk/training/online/courses/human-genetic-variation-introduction/variant-identification-and-analysis/understanding-vcf-format/
https://samtools.github.io/hts-specs/VCFv4.2.pdf
https://gatk.broadinstitute.org/hc/en-us/articles/360035531692-VCF-Variant-Call-Format