IF:16+ 基于 scRNA-seq 研究免疫抑制肿瘤微环境与胰腺癌肝转移的相关性
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.这期分享一篇2023年8月份发表在Nature Communications(IF:16.6),作者基于 scRNA-seq 研究免疫抑制肿瘤微环境与胰腺癌肝转移的相关性。该文章使用桓峰基因公众号里面生信分享教程即可实现,有需要类似思路的老师可以联系我们!
摘 要
胰腺导管腺癌(PDAC)是一种高度转移性疾病,对所有靶向和免疫治疗都难治。然而,我们对PDAC微环境特别是转移性微环境的了解非常有限,部分原因是转移性肿瘤组织难以接近。在这里,我们展示了同步切除PDAC原发肿瘤和匹配的肝转移瘤的单细胞转录组学景观。我们对原发性和转移性肿瘤的细胞组成和功能表型进行了比较分析。肿瘤细胞在肝转移中表现出不同的转录组谱,具有明确的从原发性肿瘤细胞进化的途径。我们还鉴定了转移性病变中对促肿瘤微环境形成至关重要的基质细胞和免疫细胞的特定亚型,包括RGS5+癌症相关成纤维细胞、CCL18+脂质相关巨噬细胞、S100A8+中性粒细胞和FOXP3+调节性T细胞。细胞相互作用组分析进一步揭示,转移组织中缺乏肿瘤-免疫细胞相互作用有助于形成免疫抑制微环境。我们的研究提供了PDAC肝转移的转录景观的全面表征。
生信分析流程
相关数据集选择:
4例病理诊断为PDAC伴肝转移的患者,TCGA-PDAC bulk RNA-seq
基因集选择
炎症相关基因:IFNG, IFNGR1, IFNGR2, IL10, IL12A, IL12B, IL12RB1, IL12RB2, IL13, IL17A, IL17F, IL18, IL18R1, IL18RAP, IL1A, IL1B, IL2, IL21, IL21R, IL22, IL23A, IL23R, IL2RG, IL4, IL4R, IL5, IL6, JUN, NFKB1, RELA, RORA, RORC, S100A8, S100A9, STAT1, STAT3, STAT4, STAT6, TGFB1, TGFB2, TGFB3, and TNF
细胞毒性相关基因:GZMA, GZMB, GZMH, GZMK, GZMM, GNLY, PRF1, and FASLG, IFNG, TNF, IL2R, and IL2
生信分析方法:
我们从文章的分析流程中提取所有的分析内容,整理出来就 17个分析条目,每个条目都包括分析的内容,这些分析构成了整个文章,本文属于单细胞测序生信分析类文章,下面我们就看看哪些分析可以利用桓峰基因公众号的教程来实现,点击分析条码就会跳转到对应公众号的教程,跟着教程做,您也能发轻松发高分,如下:
1. 单细胞数据比对(CellRanger V2.1.1)
2. 单细胞去除双细胞(DoubletFinder)
3. 单细胞数据指控及分析(Seurat V4.0.6)
4. 导管单细胞细胞CNV估计(InferCNV)
5. GO注释分析(clusterProfiler)
6. 基因变异富集分析(GSVA)
7. 细胞发育轨迹时间分析(Monocle2)
8. 计算RNA转录速度(velocity)
9. 细胞-细胞相互作用分析(CellPhoneDB)
10. TCGA-PDAC表达数据的免疫浸润分析(CIBERSORTx)
11. TCGA数据分析(TCGAbiolinks)
12. 构建Cox风险比率模型(survival)
13. 标记基因的生存分析(Kaplan–Meier)
14. 绘制条形图(Barplot)
15. 绘制小提琴图(ViolinPlot)
16. 绘制箱线图(BoxPlot)
17. 多组比较分析统计(ANOVA)
研究结果
1. 原发性和匹配转移性PDAC组织的单细胞转录组学分析
a. 本研究的样本采集和数据分析流程。
b. 箱形图显示了不同样本组细胞中炎症特征(n = 42)的标度平均表达。
c. 统一流形逼近和投影(UMAP)图显示了集成的细胞图,该图由来自12种注释细胞类型的29个细胞簇组成。细胞是按簇着色的。
d. 点图显示跨细胞群的代表性标记基因。斑点大小与表达特定基因的细胞比例成正比。颜色强度与特定基因的相对表达量相对应。
e. 条形图显示了不同组样本的细胞类型丰度,通过本研究中的scRNA-seq数据或Yang等人的反卷积大量RNA-seq数据测量。
f. 基于Jensen-Shannon散度(JSD)评分显示不同患者细胞类型异质性的条形图。
g. UMAP显示了主要细胞类型的分布(上图)和每种细胞类型中差异表达基因(DEG)的数量。
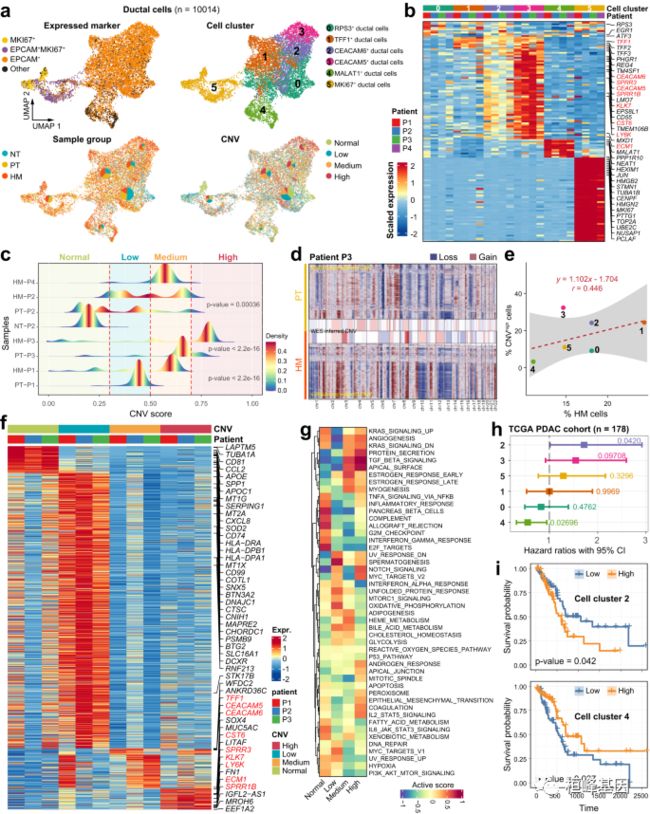
2. 导管细胞的转录特征和CNV异质性
a. UMAPs显示表达指示的maker基因的导管细胞的分布(左上),导管细胞亚型(n = 6;右上),来自不同样本组的细胞百分比(左下)或不同CNV水平的细胞百分比(右下)。
b. 热图显示6种导管细胞亚型中标记基因的表达。
c. 根据CNV评分将导管细胞分为不同的类别。游戏图显示了CNV得分在不同样本中的分布。
d. 通过scRNA-seq和全外显子组测序(WES)数据推断P3患者的CNV。
e. HM样品中细胞百分比与高水平CNVs细胞比例呈正相关。
f. 热图显示不同CNV水平的细胞间差异表达基因的比例表达水平。
g. 热图显示了使用GSVA分析在不同CNV水平的细胞中激活的功能途径。
h. 使用TCGA PDAC队列(n = 178)进行COX回归分析,相对细胞丰度(通过CIBERSORTx估计)与患者生存率的关系。
i. TCGA PDAC患者的Kaplan-Meier曲线(n = 178)显示了导管细胞簇2和4中细胞丰度分组的生存率。
3. 伪时间轨迹分析揭示了导管细胞分化状态的多样性
a. 小提琴图显示了导管细胞亚型中与PDAC增殖或恶性肿瘤相关的代表性基因的表达。
b. 通过RNA速度观察导管细胞亚型的无监督伪时间轨迹。箭头方向表示细胞假颞分化趋势。
c. 通过Monocle2推测导管细胞亚型的半监督伪时间轨迹。
d. 通过RNA速度显示患者P1-P3导管细胞亚型潜伏时间的箱线图。箱线图的左侧显示了每个类别中的单元格数量。
e. 热图显示了(c)中差异表达基因在假时间轨迹中的比例表达。
f. 患者P1-P3的导管细胞沿monocle2估计轨迹分布。
g. 每位患者导管细胞RNA速度分析(P1-P3)。
h. 维恩图显示高水平CNV、细胞状态S5和HM导管细胞中deg的重叠。
i. TCGA PDAC队列患者Kaplan-Meier曲线(n = 178)。p值采用双侧logrank检验计算。
j. 免疫组化分析(左)PT组和HM组LITAF表达。
4. 原发性和转移性PDAC组织肿瘤微环境中成纤维细胞的转录谱分析
a. UMAP显示癌症相关成纤维细胞(CAFs)的亚型,包括iCAF、apCAF和myCAF,按亚簇着色。
b. 小提琴图(左)显示了不同亚型成纤维细胞的代表性表达模式。
c. 不同样本组在UMAP上的CAFs分布。饼状图显示了每个CAF子簇中三个样本组的比例。
d. 特征图显示了选定的簇特异性基因的表达。表达水平最高的细胞用红色表示。
e. 维恩图(下图)显示了caf的子簇和样本组之间的deg重叠。
f. 免疫荧光染色显示PT和HM样品中RGS5(绿色)、a-SMA(红色)、PanCK(紫色)和DAPI(蓝色)共定位。
g. 通过RNA速度显示CAF亚型潜伏时间的箱线图。
h. 利用Monocle2观察CAF亚型的半监督伪时间轨迹。
i. 热图显示了从h开始的假时间内差异表达基因的比例表达。
j. 热图显示了通过GSVA分析在5种细胞状态(S1-S5)富集的功能通路。
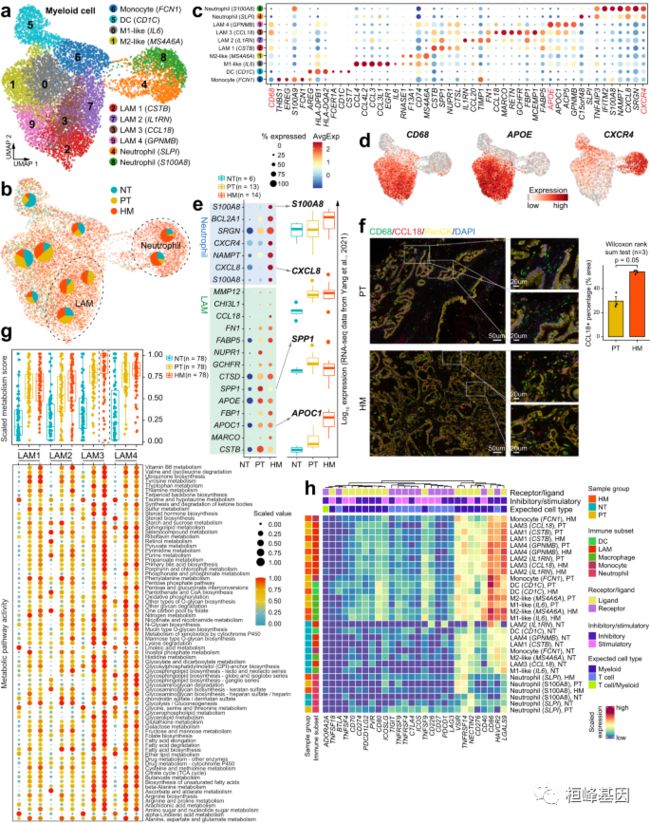
5. 原发肿瘤和肝转移组织肿瘤微环境中髓系细胞的转录全景
a. UMAP显示骨髓细胞的亚型,以亚型标记。
b. 不同样品组骨髓细胞在UMAP上的分布。饼状图显示每个细胞子簇中三个样本组的比例。
c. 点图表示每个髓细胞亚群中代表性标记基因的平均表达和频率。
d. 特征图显示了选定的簇特异性基因的表达。表达水平最高的细胞用红色表示。
e. 点图显示了三个样本组中性粒细胞和脂质相关巨噬细胞(lam)中的deg(左)。
f. 免疫荧光染色显示PT和HM样品中CD68(绿色)、CCL18(红色)、PanCK(黄色)和DAPI(蓝色)共定位。
g. 箱线图(上)显示了四个LAM亚簇(LAM1-LAM4)中代谢途径的代谢评分。
h. 热图显示一系列免疫检查点基因在骨髓细胞亚型中的按比例表达水平。亚型按样本来源和髓细胞类型注释(DC、LAM、巨噬细胞、单核细胞和中性粒细胞)分组。
6. 淋巴样细胞在原发肿瘤和肝转移组织肿瘤微环境中的转录全景
a. 亚簇状淋巴样细胞的UMAP投影,用不同的颜色标记。
b. 特征图显示了选定的簇特异性基因的表达。表达水平最高的细胞用红色表示。
c. 点图显示淋巴样细胞亚群中代表性标记基因的平均表达和频率。
d. 不同样本组淋巴样细胞在UMAP上的分布。
e. 3个样本组CD4+ T、CD8+ T和NK细胞比例箱线图。
f. 热图显示每个淋巴样细胞亚群中选定基因集的表达,包括初始、常驻、细胞毒性、耗尽、共刺激、转录因子(TF)和细胞类型。
g. 免疫荧光染色显示PT和HM样品(n = 3)中CD4(绿色)、CD8(黄色)、GNLY(红色)和DAPI(蓝色)共定位。
h. 柱状图显示了g, n = 3例配对PT和HM样本的定量结果。
i. 热图显示一系列免疫检查点基因在淋巴样细胞亚型中的按比例表达水平。亚型根据样本来源和淋巴细胞类型注释(CD4+ T, CD8+ T和NK细胞)分组。基因按受体或配体、抑制或刺激状态以及已知表达该基因的预期主要谱系细胞类型(淋巴细胞和髓细胞)分组。
j. 点图显示了三个样本组调节性T细胞中5个检查点基因(CTLA4、TIGIT、ICOS、TNFRSF4和TNFRSF9)的表达水平。
k. 热图显示来自三个样本组的调节性T细胞中选定基因集的表达,包括细胞毒性、幼稚、转录因子(TF)、常驻、细胞类型、耗尽和共刺激。
7. 原发性和转移性PDAC组织肿瘤微环境中细胞-细胞相互作用网络的动态
a. 热图说明NT、PT和HM样品中细胞-细胞相互作用模式。
b. 热图分别显示了NT、PT和HM组464对配体-受体的相互作用得分。
c. 柱状图显示了三个样品组中基团特异性配体-受体对。
d. 热图显示了细胞亚型中样品特异性配体-受体对的富集。
e. 箱线图显示TFH (CXCL13)与导管细胞之间以及Treg (FOXP3)与导管细胞之间的细胞-细胞相互作用计数。
f. 免疫荧光染色显示PT和HM样品中CD4(绿色)、FOXP3(红色)、PanCK(黄色)和DAPI(蓝色)共定位。
g. 点图显示NT、PT和HM样本中代表性促炎和细胞毒性中介基因的表达和频率。
Reference:
1. Zhang, S., Fang, W., Zhou, S. et al. Single cell transcriptomic analyses implicate an immunosuppressive tumor microenvironment in pancreatic cancer liver metastasis. Nat Commun 14, 5123 (2023). https://doi.org/10.1038/s41467-023-40727-7
号外号外,桓峰基因单细胞生信分析免费培训课程即将开始快来报名吧!
单细胞生信分析教程
桓峰基因公众号推出单细胞生信分析教程并配有视频在线教程,目前整理出来的相关教程目录如下:
Topic 6. 克隆进化之 Canopy
Topic 7. 克隆进化之 Cardelino
Topic 8. 克隆进化之 RobustClone
SCS【1】今天开启单细胞之旅,述说单细胞测序的前世今生
SCS【2】单细胞转录组 之 cellranger
SCS【3】单细胞转录组数据 GEO 下载及读取
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【5】单细胞转录组数据可视化分析 (scater)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【14】单细胞调节网络推理和聚类 (SCENIC)
SCS【15】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (CellPhoneDB)
SCS【16】从肿瘤单细胞RNA-Seq数据中推断拷贝数变化 (inferCNV)
SCS【17】从单细胞转录组推断肿瘤的CNV和亚克隆 (copyKAT)
SCS【18】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (iTALK)
SCS【19】单细胞自动注释细胞类型 (Symphony)
SCS【20】单细胞数据估计组织中细胞类型(Music)
SCS【21】单细胞空间转录组可视化 (Seurat V5)
SCS【22】单细胞转录组之 RNA 速度估计 (Velocyto.R)
SCS【23】单细胞转录组之数据整合 (Harmony)
SCS【24】单细胞数据量化代谢的计算方法 (scMetabolism)
SCS【25】单细胞细胞间通信第一部分细胞通讯可视化(CellChat)
SCS【26】单细胞细胞间通信第二部分通信网络的系统分析(CellChat)
SCS【27】单细胞转录组之识别标记基因 (scran)
SCS【28】单细胞转录组加权基因共表达网络分析(hdWGCNA)
SCS【29】单细胞基因富集分析 (singleseqgset)
SCS【30】单细胞空间转录组学数据库(STOmics DB)
SCS【31】减少障碍,加速单细胞研究数据库(Single Cell PORTAL)
SCS【32】基于scRNA-seq数据中推断单细胞的eQTLs (eQTLsingle)
SCS【33】单细胞转录之全自动超快速的细胞类型鉴定 (ScType)
SCS【34】单细胞/T细胞/抗体免疫库数据分析(immunarch)
SCS【35】单细胞转录组之去除双细胞 (DoubletFinder)
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/