本次目标
K-Means (K均值)
Vector Quantization(矢量量化)
传统模型
传统图像分类
如图,下面三个样本的维度不同:
第一个是20x144维
第二个是10x144维
第三个是15x144维
因为维度不同,所以无法喂给Logistic回归(LR)。
但是换一个思路,因为都是x144,所以可以将20,10,15看成各自独立的样本。每一张图片,看成是一个词袋,第一个袋子放了20个样本,第二个袋子放了10个样本,第三个袋子放了15个样本。
然后对于这45个样本,做一个聚类。
如中间部分,做了5种聚类,即5个簇。(K=5)
然后观察三张图片各自样本,落在这5个簇的聚类情况。
即特征变成了5维的向量。
即各个图片,在分类器看来,就是长度为5的列向量。
然后将列向量横过来,就形成3行(3张图片)五列的数据,如此即可将其喂给SVM或者随机森林都是可以的。
当然也可以映射为1000个数的向量,即变成3x1000。
无论是映射成5个还是1000个,其实都比原始维度要低的多。即通过聚类,达到降维的目的。
而中间这一步(即聚类),是一个无监督的学习。可以看做对144维向量,映射到某一个数字上去。对Vector做了量化,即矢量量化,即VQ。
然后回过头来看Vector Quantization这张ppt,
任何一张图片可以看成RGB三通道的彩色图像,任何一个像素点是三维的一个数字,换句话说,任何一个像素点其实是一个向量。
假定是800x600的图片,有48万个向量,将这项矢量做聚类,用簇的中心代替原始的像素。即可以用调色板,将图像压缩了。
课堂问答
问:图像有噪声会有影响么?
答: 应该有影响,但这个影响,有些时候可以自适应。比如图像偏红,其中有一片是绿色的,作为噪声。如果将噪声作为一个簇,则问题不大。但是椒盐噪声,而且颜色随机,会影响很大。不过椒盐噪声,如果颜色一致,则会聚类,影响就不会很大。
问:聚类跟PCA类似么?
答:不类似。
问:聚类主要用在哪?
答:一般不会把聚类作为主算法。比如用卷积网络,识别物体,有可能把聚类当成中间环节,对数据做变换。此时聚类会产生价值。比如预处理,中间预处理,或者后处理的一个步骤。完善整个环节中的其中一环。聚类可以帮助选择特征。
问:聚类可以达到降维的目的?
答:是的
问:如果是RGB,是RGB一起聚类,还是RGB各自聚类?
答:是一起聚类。
问:如果是多张图片,是一起聚类,还是各自聚类?
答:多张图片,也是一起聚类,一起做特征。
问:选特征与VQ是什么关系呢?
答:我们是利用VQ来最终选特征,选出来这1000个数(特征)。
问:在应用中,具体如何选择使用Kmeans最大密度聚类,谱聚类,DBScan哪个算法?需要逐个尝试么?
答:如果数据量特别大,只能选Kmeans;
如果数据量不是特别大,可以选密度最大聚类,或DBSCAN。
如果数据量中等或偏小,可使用谱聚类试一试。因为谱聚类不会过分探讨特征距离是怎么来的。
问:图像压缩是不是把图像所有的颜色压缩到100种颜色中去?
答:是的
问:可否理解为处理后的结果就是把占据图像主要色彩的部分给留下来?
答:不是。我们是把所有的像素给替换了。
问:聚类之后的操作是干什么?
答:之后的操作,可能是做SVM, Logisitc回归,或者将特征喂给卷积网络。具体问题具体处理。
问:能用聚类做二值化么?
答:当然能。比如可以选择黑白图像做二值化。
问:把图像变成字符的图像,是不是也可以用VQ?
答:先用VQ,倒是有可能。
问:聚类可以用于一致性问题么?就是把所有点演化成最终汇聚到一个点上?
答:这个取决于应用场景。
AP聚类
时间复杂度太高,计算速度慢一些,只能适用于中小型数据。
调参还好。
MeanShift
把均值做移动,然后生成下面的结果
密度聚类
参数有ε, m以及聚类数目。
谱聚类
参数为给出相似度的值,实际上就是标准差。
用处,在于做分隔:
代码解析
视频对应的代码:链接:https://pan.baidu.com/s/1LOeY5sWr7dyX-LBhSFI9WQ 密码:cipq
参见:第十八课_代码.zip
1. K-means
代码
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.colors
import matplotlib.pyplot as plt
import sklearn.datasets as ds
from sklearn.metrics import homogeneity_score, completeness_score, v_measure_score, adjusted_mutual_info_score,\
adjusted_rand_score, silhouette_score
from sklearn.cluster import KMeans
def expand(a, b):
d = (b - a) * 0.1
return a-d, b+d
if __name__ == "__main__":
N = 400
centers = 4
data, y = ds.make_blobs(N, n_features=2, centers=centers, random_state=2)
data2, y2 = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=(1,2.5,0.5,2), random_state=2)
data3 = np.vstack((data[y == 0][:], data[y == 1][:50], data[y == 2][:20], data[y == 3][:5]))
y3 = np.array([0] * 100 + [1] * 50 + [2] * 20 + [3] * 5)
m = np.array(((1, 1), (1, 3)))
data_r = data.dot(m)
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
cm = matplotlib.colors.ListedColormap(list('rgbm'))
data_list = data, data, data_r, data_r, data2, data2, data3, data3
y_list = y, y, y, y, y2, y2, y3, y3
titles = '原始数据', 'KMeans++聚类', '旋转后数据', '旋转后KMeans++聚类',\
'方差不相等数据', '方差不相等KMeans++聚类', '数量不相等数据', '数量不相等KMeans++聚类'
model = KMeans(n_clusters=4, init='k-means++', n_init=5)
plt.figure(figsize=(8, 9), facecolor='w')
for i, (x, y, title) in enumerate(zip(data_list, y_list, titles), start=1):
plt.subplot(4, 2, i)
plt.title(title)
if i % 2 == 1:
y_pred = y
else:
y_pred = model.fit_predict(x)
print(i)
print('Homogeneity:', homogeneity_score(y, y_pred))
print('completeness:', completeness_score(y, y_pred))
print('V measure:', v_measure_score(y, y_pred))
print('AMI:', adjusted_mutual_info_score(y, y_pred))
print('ARI:', adjusted_rand_score(y, y_pred))
print('Silhouette:', silhouette_score(x, y_pred), '\n')
plt.scatter(x[:, 0], x[:, 1], c=y_pred, s=30, cmap=cm, edgecolors='none')
x1_min, x2_min = np.min(x, axis=0)
x1_max, x2_max = np.max(x, axis=0)
x1_min, x1_max = expand(x1_min, x1_max)
x2_min, x2_max = expand(x2_min, x2_max)
plt.xlim((x1_min, x1_max))
plt.ylim((x2_min, x2_max))
plt.grid(b=True, ls=':')
plt.tight_layout(2, rect=(0, 0, 1, 0.97))
plt.suptitle('数据分布对KMeans聚类的影响', fontsize=18)
plt.show()
结果如下:
1
Homogeneity: 1.0
completeness: 1.0
V measure: 1.0
AMI: 1.0
ARI: 1.0
Silhouette: 0.616436816839852
2
Homogeneity: 0.9898828240244267
completeness: 0.9899006758819153
V measure: 0.9898917498726852
AMI: 0.9897991568445268
ARI: 0.9933165272203728
Silhouette: 0.6189656317733315
3
Homogeneity: 1.0
completeness: 1.0
V measure: 1.0
AMI: 1.0
ARI: 1.0
Silhouette: 0.5275987244664399
4
Homogeneity: 0.7248868671759175
completeness: 0.7260584887742589
V measure: 0.7254722049396414
AMI: 0.7226116135013659
ARI: 0.6703250071796025
Silhouette: 0.5349498852778517
5
Homogeneity: 1.0
completeness: 1.0
V measure: 1.0
AMI: 1.0
ARI: 1.0
Silhouette: 0.4790725752982868
6
Homogeneity: 0.7449364376693913
completeness: 0.7755445167472194
V measure: 0.7599323988656884
AMI: 0.7428234768685047
ARI: 0.7113213508090338
Silhouette: 0.5737260449304202
7
Homogeneity: 1.0
completeness: 1.0
V measure: 1.0
AMI: 1.0
ARI: 1.0
Silhouette: 0.5975066093204152
8
Homogeneity: 0.9776347312784609
completeness: 0.9728632742060752
V measure: 0.975243166591057
AMI: 0.9721283376882836
ARI: 0.9906840043816505
Silhouette: 0.6013877858619149
silhouette系数
轮廓系数(Silhouette Coefficient),是聚类效果好坏的一种评价方式。最早由 Peter J. Rousseeuw 在 1986 提出。它结合内聚度和分离度两种因素。可以用来在相同原始数据的基础上用来评价不同算法、或者算法不同运行方式对聚类结果所产生的影响。
- si接近1,则说明样本i聚类合理;
- si接近-1,则说明样本i更应该分类到另外的簇;
- 若si 近似为0,则说明样本i在两个簇的边界上。
聚类评估算法-轮廓系数(Silhouette Coefficient )
如第二张图(KMeans++聚类),其silhouette系数是0.6189656317733315,为某簇的点到该簇其他样本点的平均距离为A,以及该点到其他簇的样本平均距离为B。然后1- A/B,即得到轮廓系数。
最优情况,是等于1,但是所有数据都不会等于1的。因为其到自身样本的平均距离不可能为0,其到其他簇的平均距离不可能为无穷大。一定是小于1的数字。
KMean算法本质
KMean算法本质上认为样本的每一个簇都是服从方差相等的高斯分布,如果方差不相等,则需要EM算法期望最大化算法来进行建模,计算。
代码解析
# make_blobs: 这个函数本质是用于生成若干个高斯分布的样本。
# N = 400, 表明生成400个样本
# centers = 4,表明生成4个中心点(簇)
# n_features = 2: 设置为2维数据,因为好画图
# random_state = 2,用于保证每次生成数据一样,生成随机数种子,保证样本是特定的
N = 400
centers = 4
data, y = ds.make_blobs(N, n_features=2, centers=centers, random_state=2)
# cluster_std = (1,2.5,0.5,2),表明对高斯分布的方差做个变换,默认情况下,方差都是1.
data2, y2 = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=(1,2.5,0.5,2), random_state=2)
# 以下代码,使得四个类别的样本不均衡
# y == 0: 取全部;y == 1: 取50个; y == 2: 取20个; y ==3: 取5个
data3 = np.vstack((data[y == 0][:], data[y == 1][:50], data[y == 2][:20], data[y == 3][:5]))
# 使得y3与data3数量一致
y3 = np.array([0] * 100 + [1] * 50 + [2] * 20 + [3] * 5)
# 对矩阵做乘法,本质上就是对矩阵做旋转
m = np.array(((1, 1), (1, 3)))
data_r = data.dot(m)
# 生成KMeans的模型,init为初始样本的方法, n_init = 5: 做5次计算,从5次结果选择最好的结果输出,默认是10
model = KMeans(n_clusters=4, init='k-means++', n_init=5)
# 预测聚类
y_pred = model.fit_predict(x)
from sklearn.metrics import homogeneity_score, \
completeness_score, \
v_measure_score, \
adjusted_mutual_info_score, \
adjusted_rand_score, \
silhouette_score
...
...
# 衡量指标的方法在sklearn里面都已经内置了,只需要调用即可
# 这些指标:取1是最优的,取0是最差的;当然轮廓系数理论上是达不到1的。
print('Homogeneity:', homogeneity_score(y, y_pred))
print('completeness:', completeness_score(y, y_pred))
print('V measure:', v_measure_score(y, y_pred))
print('AMI:', adjusted_mutual_info_score(y, y_pred))
print('ARI:', adjusted_rand_score(y, y_pred))
print('Silhouette:', silhouette_score(x, y_pred), '\n')
# 样本x是二维数据,所以通过下面方法做散状图
# 计算出来的y_pred为类别(c: classification)
# cmap: 颜色的映射表: r:红,g:绿,b:蓝,m:品红
cm = matplotlib.colors.ListedColormap(list('rgbm'))
plt.scatter(x[:, 0], x[:, 1], c=y_pred, s=30, cmap=cm, edgecolors='none')
输出为三维聚类图像的代码
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.colors
import matplotlib.pyplot as plt
import sklearn.datasets as ds
from sklearn.metrics import homogeneity_score, completeness_score, v_measure_score, adjusted_mutual_info_score,\
adjusted_rand_score, silhouette_score
from sklearn.cluster import KMeans
from mpl_toolkits.mplot3d import Axes3D
def expand(a, b):
d = (b - a) * 0.1
return a-d, b+d
if __name__ == "__main__":
N = 400
centers = 4
# data, y = ds.make_blobs(N, n_features=2, centers=centers, random_state=2)
# data2, y2 = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=(1,2.5,0.5,2), random_state=2)
data, y = ds.make_blobs(N, n_features=3, centers=centers, random_state=2)
data2, y2 = ds.make_blobs(N, n_features=3, centers=centers, cluster_std=(1,2.5,0.5,2), random_state=2)
data3 = np.vstack((data[y == 0][:], data[y == 1][:50], data[y == 2][:20], data[y == 3][:5]))
y3 = np.array([0] * 100 + [1] * 50 + [2] * 20 + [3] * 5)
# m = np.array(((1, 1), (1, 3)))
# 对3x3的矩阵,乘上一个数据,就是对数据做一个旋转,平移,对称,错切的变换
m = np.array(((1, 1, 1), (1, 3, 2), (3, 6, 1)))
data_r = data.dot(m)
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
cm = matplotlib.colors.ListedColormap(list('rgbm'))
data_list = data, data, data_r, data_r, data2, data2, data3, data3
y_list = y, y, y, y, y2, y2, y3, y3
titles = '原始数据', 'KMeans++聚类', '旋转后数据', '旋转后KMeans++聚类',\
'方差不相等数据', '方差不相等KMeans++聚类', '数量不相等数据', '数量不相等KMeans++聚类'
model = KMeans(n_clusters=4, init='k-means++', n_init=5)
# plt.figure(figsize=(8, 9), facecolor='w')
fig = plt.figure(figsize=(8, 9), facecolor='w')
for i, (x, y, title) in enumerate(zip(data_list, y_list, titles), start=1):
# plt.subplot(4, 2, i)
ax = fig.add_subplot(4, 2, i, projection='3d')
plt.title(title)
if i % 2 == 1:
y_pred = y
else:
y_pred = model.fit_predict(x)
print(i)
print('Homogeneity:', homogeneity_score(y, y_pred))
print('completeness:', completeness_score(y, y_pred))
print('V measure:', v_measure_score(y, y_pred))
print('AMI:', adjusted_mutual_info_score(y, y_pred))
print('ARI:', adjusted_rand_score(y, y_pred))
print('Silhouette:', silhouette_score(x, y_pred), '\n')
# plt.scatter(x[:, 0], x[:, 1], c=y_pred, s=30, cmap=cm, edgecolors='none')
ax.scatter(x[:, 0], x[:, 1], x[:, 2], s=30, c=y_pred, cmap=cm, edgecolors='none', depthshade=True)
# x1_min, x2_min = np.min(x, axis=0)
# x1_max, x2_max = np.max(x, axis=0)
# x1_min, x1_max = expand(x1_min, x1_max)
# x2_min, x2_max = expand(x2_min, x2_max)
# plt.xlim((x1_min, x1_max))
# plt.ylim((x2_min, x2_max))
ax.grid(b=True, ls=':')
plt.tight_layout(2, rect=(0, 0, 1, 0.97))
plt.suptitle('数据分布对KMeans聚类的影响', fontsize=18)
plt.show()
效果如下:
问答
- 问:四个特征怎么画?
答:四个特征很麻烦,画不了。。。
一般是先画三个特征,或者是先做PCA(主成分分析),然后再去画它
2. Criteria
# !/usr/bin/python
# -*- coding:utf-8 -*-
from sklearn import metrics
if __name__ == "__main__":
y = [0, 0, 0, 1, 1, 1]
y_hat = [0, 0, 1, 1, 2, 2]
h = metrics.homogeneity_score(y, y_hat)
c = metrics.completeness_score(y, y_hat)
print('y: {0}, y_hat: {1}'.format(y, y_hat))
print('同一性(Homogeneity):', h)
print('完整性(Completeness):', c)
v2 = 2 * c * h / (c + h)
v = metrics.v_measure_score(y, y_hat)
print('V-Measure:', v2, v)
print()
y = [0, 0, 0, 1, 1, 1]
y_hat = [0, 0, 1, 3, 3, 3]
print('y: {0}, y_hat: {1}'.format(y, y_hat))
h = metrics.homogeneity_score(y, y_hat)
c = metrics.completeness_score(y, y_hat)
v = metrics.v_measure_score(y, y_hat)
print('同一性(Homogeneity):', h)
print('完整性(Completeness):', c)
print('V-Measure:', v)
# 允许不同值
print()
y = [0, 0, 0, 1, 1, 1]
y_hat = [1, 1, 1, 0, 0, 0]
print('y: {0}, y_hat: {1}'.format(y, y_hat))
h = metrics.homogeneity_score(y, y_hat)
c = metrics.completeness_score(y, y_hat)
v = metrics.v_measure_score(y, y_hat)
print('同一性(Homogeneity):', h)
print('完整性(Completeness):', c)
print('V-Measure:', v)
print()
y = [0, 0, 1, 1]
y_hat = [0, 1, 0, 1]
print('y: {0}, y_hat: {1}'.format(y, y_hat))
ari = metrics.adjusted_rand_score(y, y_hat)
print('adjusted_rand_score: {0}'.format(ari))
print()
y = [0, 0, 0, 1, 1, 1]
y_hat = [0, 0, 1, 1, 2, 2]
print('y: {0}, y_hat: {1}'.format(y, y_hat))
ari = metrics.adjusted_rand_score(y, y_hat)
print('adjusted_rand_score: {0}'.format(ari))
输出值:
y: [0, 0, 0, 1, 1, 1], y_hat: [0, 0, 1, 1, 2, 2]
同一性(Homogeneity): 0.6666666666666669
完整性(Completeness): 0.420619835714305
V-Measure: 0.5158037429793889 0.5158037429793889
y: [0, 0, 0, 1, 1, 1], y_hat: [0, 0, 1, 3, 3, 3]
同一性(Homogeneity): 1.0
完整性(Completeness): 0.6853314789615865
V-Measure: 0.8132898335036762
y: [0, 0, 0, 1, 1, 1], y_hat: [1, 1, 1, 0, 0, 0]
同一性(Homogeneity): 1.0
完整性(Completeness): 1.0
V-Measure: 1.0
y: [0, 0, 1, 1], y_hat: [0, 1, 0, 1]
adjusted_rand_score: -0.49999999999999994
y: [0, 0, 0, 1, 1, 1], y_hat: [0, 0, 1, 1, 2, 2]
adjusted_rand_score: 0.24242424242424246
名词解释
同一性 Hommogeneity
这种衡量标准建立在一个簇只包含一个类别样本的信息熵,h越大说明分类越好
完整性 Completeness
这种衡量标准建立在同种样本属于同一个簇的信息熵,h越大说明分类越好
V-Measure
归一性和完整性的加权平均
2 * 均一性 * 完整性 / (均一性 + 完整性)
ARI
3. 矢量量化
代码
以下代码的本质,是将图片做一百个簇的聚类,每个簇的中心点代表具体的RGB颜色,即得到100种颜色。
然后将图片512x512个像素,每一个像素归类到这一百个簇的某一个(即获取对应簇的中心点的值),然后重新绘制。
本质就是将原始图片,使用聚类之后的100个颜色重新绘制~~~
# !/usr/bin/python
# -*- coding: utf-8 -*-
from PIL import Image
import numpy as np
from sklearn.cluster import KMeans
import matplotlib
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
def restore_image(cb, cluster, shape):
row, col, dummy = shape
image = np.empty((row, col, 3))
index = 0
for r in range(row):
for c in range(col):
image[r, c] = cb[cluster[index]]
index += 1
return image
def show_scatter(a):
N = 10
print('原始数据:\n', a)
density, edges = np.histogramdd(a, bins=[N,N,N], range=[(0,1), (0,1), (0,1)])
# 将数据显示为传统小数形式
np.set_printoptions(suppress=True, linewidth=500, edgeitems=N)
print('原始density: \n', density)
print('原始density.shape: \n', density.shape)
density /= density.sum()
print('转换后density: \n', density)
x = y = z = np.arange(N)
d = np.meshgrid(x, y, z)
print('d = \n', d)
fig = plt.figure(1, facecolor='w')
ax = fig.add_subplot(111, projection='3d')
ax.scatter(d[1], d[0], d[2], c='r', s=100*density/density.max(), marker='o', edgecolors='k', depthshade=True)
ax.set_xlabel('红色分量')
ax.set_ylabel('绿色分量')
ax.set_zlabel('蓝色分量')
plt.title('图像颜色三维频数分布', fontsize=13)
plt.figure(2, facecolor='w')
den = density[density > 0]
print(den.shape)
den = np.sort(den)[::-1]
t = np.arange(len(den))
plt.plot(t, den, 'r-', t, den, 'go', lw=2)
plt.title('图像颜色频数分布', fontsize=13)
plt.grid(True)
plt.show()
if __name__ == '__main__':
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
num_vq = 100
im = Image.open('.\\lena.png') # son.bmp(100)/flower2.png(200)/son.png(60)/lena.png(50)
# 图像是512x512像素
image = np.array(im).astype(np.float) / 255
#这里构建512x512像素,每个像素各有RGB三原色的复合矩阵
image = image[:, :, :3]
# 降维,使其成为262144 x 3的矩阵
image_v = image.reshape((-1, 3))
show_scatter(image_v)
N = image_v.shape[0] # 图像像素总数
print('图像像素总数: ', N)
# 选择足够多的样本(如1000个),计算聚类中心
idx = np.random.randint(0, N, size=1000)
print('idx is: \n', idx)
image_sample = image_v[idx]
print('image_sample: \n', image_sample)
model = KMeans(num_vq)
model.fit(image_sample)
c = model.predict(image_v) # 聚类结果
# 聚类结果的值事实上对应聚类中心的索引
print('聚类结果:\n', c, '聚类结果个数: \n', len(c))
print('聚类中心:\n', model.cluster_centers_, '聚类中心个数: \n', len(model.cluster_centers_))
# 得到聚类结果后的使用方式:
# 如果想知道第1个聚类结果的聚类中心的值,用如下代码实现:
print('聚类结果第1个聚类中心的值是: \n', model.cluster_centers_[c[0]])
plt.figure(figsize=(12, 6), facecolor='w')
plt.subplot(121)
plt.axis('off')
plt.title('原始图片', fontsize=14)
plt.imshow(image)
# plt.savefig('1.png')
plt.subplot(122)
print('image shape: ', image.shape)
vq_image = restore_image(model.cluster_centers_, c, image.shape)
plt.axis('off')
plt.title('矢量量化后图片:%d色' % num_vq, fontsize=14)
plt.imshow(vq_image)
plt.savefig('lena100.png')
plt.tight_layout(2)
plt.show()
运行图例
- 可以发现红色占比最多,绿色有一些,蓝色几乎没有,所以图像基本偏黄
- 占比最多的图像颜色,只有少数数量。占比最高的,超过7%
- 代码中有通过np.histogramdd对数组进行直方图统计,并求占比的例子
- 可见前100几乎占据所有颜色,那么num_vq如果设为100,效果将会好很多
问答
问:Kmeans聚类(二、三百类别)结果如果某一类很多,差不多占一半样本,其他类很少,怎么判断聚类结果是否合理?
答:不合理。特例是:中国五十六个民族,汉族人口占据大多数。
但是实际工作中,可能有噪声。
问:可以不可以加一些特征让它更清晰?
答:可以
问:几百维特征,选择特征还是挺麻烦的事
答:是的。
问:如果用Tensorflow,定义输出维度为K,Loss为输入和输出的误差平方和,采取梯度下降算法实现聚类可行么?
答:这么做的话,会有一个问题。聚类没有Y,于是就没有所谓标记的问题。但是我们可以按照如下方式去尝试:
比如有M个向量,每个向量100维,然后喂给Tensorflow,使用CNN或U-NET进行训练,输出依然是原数据。
此时,我们拿出倒数第二层的神经元数据,可以是20个进行降维,也可以是1000个进行升维。目的是拿到经过训练后的训练数据的特征。
问:如果类别数目K事先未知,且数据有噪声,直观上觉得哪种算法更适合一些?
答:选择基于密度的DBScan或Density Peak,因为它们都不需要事先知道K,并且密度聚类天然可以发现噪声。只要没有核心对象指向它们,我们都判定为噪声。
4. AP聚类
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
import sklearn.datasets as ds
import matplotlib.colors
from sklearn.cluster import AffinityPropagation
from sklearn.metrics import euclidean_distances
if __name__ == "__main__":
N = 400
centers = [[1, 2], [-1, -1], [1, -1], [-1, 1]]
data, y = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=[0.5, 0.25, 0.7, 0.5], random_state=0)
# 使用欧式距离(欧几里得距离)
m = euclidean_distances(data, squared=True)
preference = -np.median(m)
print('Preference:', preference)
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
plt.figure(figsize=(12, 9), facecolor='w')
for i, mul in enumerate(np.linspace(1, 4, 9)):
print(mul)
p = mul * preference
model = AffinityPropagation(affinity='euclidean', preference=p)
af = model.fit(data)
center_indices = af.cluster_centers_indices_
n_clusters = len(center_indices)
print(('p = %.1f' % mul), p, '聚类簇的个数为:', n_clusters)
y_hat = af.labels_
plt.subplot(3, 3, i+1)
plt.title('Preference:%.2f,簇个数:%d' % (p, n_clusters))
clrs = []
for c in np.linspace(16711680, 255, n_clusters, dtype=int):
clrs.append('#%06x' % c)
# clrs = plt.cm.Spectral(np.linspace(0, 1, n_clusters))
for k, clr in enumerate(clrs):
cur = (y_hat == k)
# 绘制中心点周边的圆点
plt.scatter(data[cur, 0], data[cur, 1], s=15, c=clr, edgecolors='none')
center = data[center_indices[k]]
for x in data[cur]:
# 将中心点与周边的圆点连线
plt.plot([x[0], center[0]], [x[1], center[1]], color=clr, lw=0.5, zorder=1)
# 绘制中心点,用星表示
plt.scatter(data[center_indices, 0], data[center_indices, 1], s=80, c=clrs, marker='*', edgecolors='k', zorder=2)
plt.grid(b=True, ls=':')
plt.tight_layout()
plt.suptitle('AP聚类', fontsize=20)
plt.subplots_adjust(top=0.92)
plt.show()
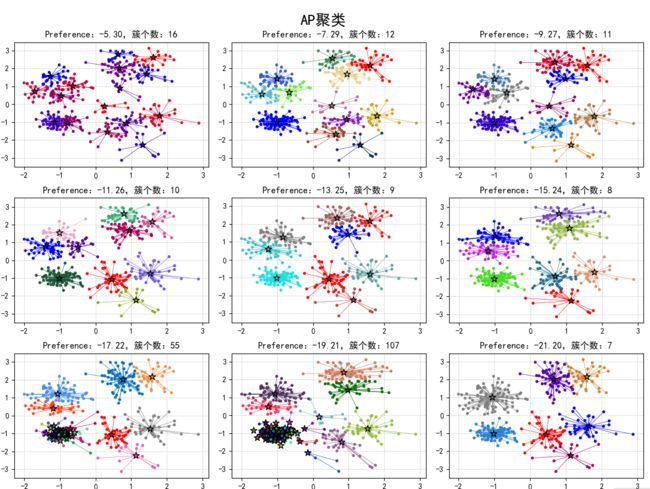
展示图形示例:
理论上, 簇个数应该越来越少,但是如图示居然有55余107个簇的情况,估计是sklearn的小bug
代码中,data的shape是(400, 2),求欧氏距离,是两两之间求距离,即得到一个400x400的距离矩阵。加负号的话,可以认为是相似度。np.median(m)目的是从16万个数中获得中位数,-np.median(m)的目的是获得preference的初值。
可以从上面的代码,了解绘图的一些技巧
5. MeanShift聚类
Mean: 均值,Shift: 变换
Mean shift 算法是基于核密度估计的爬山算法,可用于聚类、图像分割、跟踪等
代码如下:
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
import sklearn.datasets as ds
import matplotlib.colors
from sklearn.cluster import MeanShift
from sklearn.metrics import euclidean_distances
if __name__ == "__main__":
N = 1000
centers = [[1, 2], [-1, -1], [1, -1], [-1, 1]]
data, y = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=[0.5, 0.25, 0.7, 0.5], random_state=0)
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
plt.figure(figsize=(8, 7), facecolor='w')
m = euclidean_distances(data, squared=True)
bw = np.median(m)
print(bw)
for i, mul in enumerate(np.linspace(0.1, 0.4, 4)):
band_width = mul * bw
model = MeanShift(bin_seeding=True, bandwidth=band_width)
ms = model.fit(data)
centers = ms.cluster_centers_
y_hat = ms.labels_
n_clusters = np.unique(y_hat).size
print('带宽:', mul, band_width, '聚类簇的个数为:', n_clusters)
plt.subplot(2, 2, i+1)
plt.title('带宽:%.2f,聚类簇的个数为:%d' % (band_width, n_clusters))
clrs = []
# 这个颜色不好看,通过下面的方式固定颜色
# for c in np.linspace(16711680, 255, n_clusters, dtype=int):
# clrs.append('#%06x' % c)
# 用rgbm不合适,因为只能为4个聚类绘制数据点
# clrs = list('rgbm')
clrs = plt.cm.Spectral(np.linspace(0, 1, n_clusters))
for k, clr in enumerate(clrs):
cur = (y_hat == k)
# 绘制中心附近的点
plt.scatter(data[cur, 0], data[cur, 1], s=10, c=clr, edgecolors='none')
# 绘制中心点
plt.scatter(centers[:, 0], centers[:, 1], s=200, c=clrs, marker='*', edgecolors='k')
plt.grid(b=True, ls=':')
plt.tight_layout(2)
plt.suptitle('MeanShift聚类', fontsize=15)
plt.subplots_adjust(top=0.9)
plt.show()
效果如图:
MeanShift的参数:
- bandwidth
bandwidth : float, optional
Bandwidth used in the RBF kernel.
RBF: 即高斯核函数。
所谓径向基函数 (Radial Basis Function 简称 RBF), 就是某种沿径向对称的标量函数。 通常定义为空间中任一点x到某一中心xc之间欧氏距离的单调函数 , 可记作 k(||x-xc||), 其作用往往是局部的 , 即当x远离xc时函数取值很小。最常用的径向基函数是高斯核函数 ,形式为 k(||x-xc||)=exp{- ||x-xc||^2/(2*σ^2) } 其中xc为核函数中心,σ为函数的宽度参数 , 控制了函数的径向作用范围。
If not given, the bandwidth is estimated using
sklearn.cluster.estimate_bandwidth; see the documentation for that
function for hints on scalability (see also the Notes, below).
- bin_seeding
bin_seeding : boolean, optional
If true, initial kernel locations are not locations of all
points, but rather the location of the discretized version of
points, where points are binned onto a grid whose coarseness
corresponds to the bandwidth. Setting this option to True will speed
up the algorithm because fewer seeds will be initialized.
default value: False
Ignored if seeds argument is not None.
下图可以很直观表示中心点(均值Mean)不断漂移(Shift),渐趋合理的过程(即不再收敛,中心点漂移就结束了)
图中的圆的半径,即band width
带宽太小,则簇过于琐碎
带宽太大,则簇不再明显与合理。
所以合理的带宽是关键。
问答
- Preference是什么?
答:是指AP算法里面的那个值,是不是愿意作为聚类中心的初始值。因为所谓相似是指我想做聚类中心,对其他样本的吸引程度;以及某一个样本不想做聚类中心,它依靠于某一个聚类中心的依赖程度。 - AP为啥要给定初始聚类中心?
答:我们不需要给定初始聚类中心。我们需要给定的是任何一个值,它的初始a和r值是几,那个是用中位数做的。 - sklearn中的k-means算法如何选择度量距离的类别?比如余弦相似度距离?
答:k-means类的构造函数有precompute_distances的参数,默认值是auto。
官方解释如下:
precompute_distances : {'auto', True, False}
Precompute distances (faster but takes more memory).
'auto' : do not precompute distances if n_samples * n_clusters > 12 million. This corresponds to about 100MB overhead per job using double precision.
True : always precompute distances
False : never precompute distances
- sklearn.datasets的make_blob,返回值得y值是中心的标记么,返回的值结果是什么?
答:返回的y是0,1,2,3,..., k-1类别的值,表示data的每一个成员属于哪个类别。
6. DBSCAN 密度聚类
代码:
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
import sklearn.datasets as ds
import matplotlib.colors
from sklearn.cluster import DBSCAN
from sklearn.preprocessing import StandardScaler
def expand(a, b):
d = (b - a) * 0.1
return a-d, b+d
if __name__ == "__main__":
N = 1000
centers = [[1, 2], [-1, -1], [1, -1], [-1, 1]]
data, y = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=[0.5, 0.25, 0.7, 0.5], random_state=0)
data = StandardScaler().fit_transform(data)
# 数据1的参数:(epsilon, min_sample)
params = ((0.2, 5), (0.2, 10), (0.2, 15), (0.3, 5), (0.3, 10), (0.3, 15))
# 数据2
# t = np.arange(0, 2*np.pi, 0.1)
# data1 = np.vstack((np.cos(t), np.sin(t))).T
# data2 = np.vstack((2*np.cos(t), 2*np.sin(t))).T
# data3 = np.vstack((3*np.cos(t), 3*np.sin(t))).T
# data = np.vstack((data1, data2, data3))
# # # 数据2的参数:(epsilon, min_sample)
# params = ((0.5, 3), (0.5, 5), (0.5, 10), (1., 3), (1., 10), (1., 20))
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
plt.figure(figsize=(9, 7), facecolor='w')
plt.suptitle('DBSCAN聚类', fontsize=15)
for i in range(6):
eps, min_samples = params[i]
model = DBSCAN(eps=eps, min_samples=min_samples)
model.fit(data)
y_hat = model.labels_
core_indices = np.zeros_like(y_hat, dtype=bool)
core_indices[model.core_sample_indices_] = True
# y_hat中的-1值,代表数据噪声。
y_unique = np.unique(y_hat)
n_clusters = y_unique.size - (1 if -1 in y_hat else 0)
print(y_unique, '聚类簇的个数为:', n_clusters)
# clrs = []
# for c in np.linspace(16711680, 255, y_unique.size):
# clrs.append('#%06x' % c)
plt.subplot(2, 3, i+1)
clrs = plt.cm.Spectral(np.linspace(0, 0.8, y_unique.size))
print(clrs)
for k, clr in zip(y_unique, clrs):
cur = (y_hat == k)
if k == -1:
# 绘制噪声数据,显示为黑点
plt.scatter(data[cur, 0], data[cur, 1], s=10, c='k')
continue
plt.scatter(data[cur, 0], data[cur, 1], s=15, c=clr, edgecolors='k')
plt.scatter(data[cur & core_indices][:, 0], data[cur & core_indices][:, 1], s=30, c=clr, marker='o', edgecolors='k')
x1_min, x2_min = np.min(data, axis=0)
x1_max, x2_max = np.max(data, axis=0)
x1_min, x1_max = expand(x1_min, x1_max)
x2_min, x2_max = expand(x2_min, x2_max)
plt.xlim((x1_min, x1_max))
plt.ylim((x2_min, x2_max))
plt.plot()
plt.grid(b=True, ls=':', color='#606060')
plt.title(r'$\epsilon$ = %.1f m = %d,聚类数目:%d' % (eps, min_samples, n_clusters), fontsize=12)
plt.tight_layout()
plt.subplots_adjust(top=0.9)
plt.show()
执行效果:
如果使用数据2,(在代码中,将数据2的注释去掉即可使用),会得到环形数据的聚类结果:
- 一般来说,参数epsilon大的时候,m值也要跟着变大。因为epsilon变大,意味着半径变大
7. HDBSCAN聚类
HDBSCAN的介绍:
可以这样理解,HDBSCAN是密度聚类与层次聚类统一的一个聚类算法。
HDBSCAN - Hierarchical Density-Based Spatial Clustering of Applications with Noise. Performs DBSCAN over varying epsilon values and integrates the result to find a clustering that gives the best stability over epsilon. This allows HDBSCAN to find clusters of varying densities (unlike DBSCAN), and be more robust to parameter selection.
In practice this means that HDBSCAN returns a good clustering straight away with little or no parameter tuning -- and the primary parameter, minimum cluster size, is intuitive and easy to select.
HDBSCAN is ideal for exploratory data analysis; it's a fast and robust algorithm that you can trust to return meaningful clusters (if there are any).
更详细的介绍:How HDBSCAN Works
hdbscan: 并不存在于scikit-learn里面,需要自行安装相关的组件包:
pip install hdbscan
从代码中,能看出没有epsilon这个参数了,这样聚类结果就非常合理
代码:
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
import sklearn.datasets as ds
import matplotlib.colors
from sklearn.cluster import DBSCAN
from sklearn.preprocessing import StandardScaler
import hdbscan
def expand(a, b):
d = (b - a) * 0.1
return a-d, b+d
if __name__ == "__main__":
N = 1000
centers = [[1, 2], [-1, -1], [1, -1], [-1, 1]]
data, y = ds.make_blobs(N, n_features=2, centers=centers, cluster_std=[0.5, 0.25, 0.7, 0.5], random_state=0)
data = StandardScaler().fit_transform(data)
# 数据1的参数:(epsilon, min_sample)
params = ((0.2, 5), (0.2, 10), (0.2, 15), (0.3, 5), (0.3, 10), (0.3, 15))
# 数据2
# t = np.arange(0, 2*np.pi, 0.1)
# data1 = np.vstack((np.cos(t), np.sin(t))).T
# data2 = np.vstack((2*np.cos(t), 2*np.sin(t))).T
# data3 = np.vstack((3*np.cos(t), 3*np.sin(t))).T
# data = np.vstack((data1, data2, data3))
# # # 数据2的参数:(epsilon, min_sample)
# params = ((0.5, 3), (0.5, 5), (0.5, 10), (1., 3), (1., 10), (1., 20))
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
plt.figure(figsize=(12, 8), facecolor='w')
plt.suptitle('HDBSCAN聚类', fontsize=16)
for i in range(6):
eps, min_samples = params[i]
model = hdbscan.HDBSCAN(min_cluster_size=10, min_samples=min_samples)
model.fit(data)
y_hat = model.labels_
core_indices = np.zeros_like(y_hat, dtype=bool)
core_indices[y_hat != -1] = True
y_unique = np.unique(y_hat)
n_clusters = y_unique.size - (1 if -1 in y_hat else 0)
print(y_unique, '聚类簇的个数为:', n_clusters)
# clrs = []
# for c in np.linspace(16711680, 255, y_unique.size):
# clrs.append('#%06x' % c)
plt.subplot(2, 3, i+1)
clrs = plt.cm.Spectral(np.linspace(0, 0.8, y_unique.size))
for k, clr in zip(y_unique, clrs):
cur = (y_hat == k)
if k == -1:
plt.scatter(data[cur, 0], data[cur, 1], s=20, c='k')
continue
plt.scatter(data[cur, 0], data[cur, 1], s=60*model.probabilities_[cur], marker='o', c=clr, edgecolors='k', alpha=0.9)
plt.scatter(data[cur & core_indices][:, 0], data[cur & core_indices][:, 1], s=60, c=clr, marker='o', edgecolors='k')
x1_min, x2_min = np.min(data, axis=0)
x1_max, x2_max = np.max(data, axis=0)
x1_min, x1_max = expand(x1_min, x1_max)
x2_min, x2_max = expand(x2_min, x2_max)
plt.xlim((x1_min, x1_max))
plt.ylim((x2_min, x2_max))
plt.grid(b=True, ls=':', color='#808080')
plt.title(r'$\epsilon$ = %.1f m = %d,聚类数目:%d' % (eps, min_samples, n_clusters), fontsize=13)
plt.tight_layout()
plt.subplots_adjust(top=0.9)
plt.show()
执行图例。
数据1的图例,从图例可以看出聚类的数目都是4,不会出现不合理的只有1~2个聚类:
数据2的图例,从图例可以看出聚类的数目是2~3,不会出现不合理的只有1个聚类的情况:
8. 谱聚类
谱聚类同样可以如密度聚类处理不规则数据分布,如圆环形数据。
算法集位于: sklearn.cluster.SpectralClustering
代码:
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
import sklearn.datasets as ds
import matplotlib.colors
from sklearn.cluster import SpectralClustering
from sklearn.metrics import euclidean_distances
def expand(a, b):
d = (b - a) * 0.1
return a-d, b+d
if __name__ == "__main__":
matplotlib.rcParams['font.sans-serif'] = ['SimHei']
matplotlib.rcParams['axes.unicode_minus'] = False
t = np.arange(0, 2*np.pi, 0.1)
data1 = np.vstack((np.cos(t), np.sin(t))).T
data2 = np.vstack((2*np.cos(t), 2*np.sin(t))).T
data3 = np.vstack((3*np.cos(t), 3*np.sin(t))).T
data = np.vstack((data1, data2, data3))
n_clusters = 3
m = euclidean_distances(data, squared=True)
plt.figure(figsize=(12, 8), facecolor='w')
plt.suptitle('谱聚类', fontsize=16)
clrs = plt.cm.Spectral(np.linspace(0, 0.8, n_clusters))
for i, s in enumerate(np.logspace(-2, 0, 6)):
print(s)
af = np.exp(-m ** 2 / (s ** 2)) + 1e-6
model = SpectralClustering(n_clusters=n_clusters, affinity='precomputed', assign_labels='kmeans', random_state=1)
y_hat = model.fit_predict(af)
plt.subplot(2, 3, i+1)
for k, clr in enumerate(clrs):
cur = (y_hat == k)
plt.scatter(data[cur, 0], data[cur, 1], s=40, c=clr, edgecolors='k')
x1_min, x2_min = np.min(data, axis=0)
x1_max, x2_max = np.max(data, axis=0)
x1_min, x1_max = expand(x1_min, x1_max)
x2_min, x2_max = expand(x2_min, x2_max)
plt.xlim((x1_min, x1_max))
plt.ylim((x2_min, x2_max))
plt.grid(b=True, ls=':', color='#808080')
plt.title(r'$\sigma$ = %.2f' % s, fontsize=13)
plt.tight_layout()
plt.subplots_adjust(top=0.9)
plt.show()
可以发现,当σ足够高,如1.0时,高斯分布越来越平,即不再衰减。
或许选择的最终特征,就没有降维打击能力,则谱聚类,退化到近似于K-Means聚类。
图例:
课堂问答
- 问:聚类算法调参是要可视化出来看效果的么?
答:其实是需要的,可视化效果会相对直观。