二代三代转录组测序分析实战班
转录组大家都很熟悉了,我们之前也有几篇介绍:
- 转录组分析的正确姿势
- 39个转录组分析工具,120种组合评估(转录组分析工具哪家强-导读版)
- 120分的转录组考题,你能得多少
年前开了一期二代转录组线下研讨班,效果挺好,大家学的很扎实,不少朋友又继续参加了后续其它内容的培训课程。在一起学习,一起聊天,互相有些收获;能得到持续的认可,是我们最好的动力。
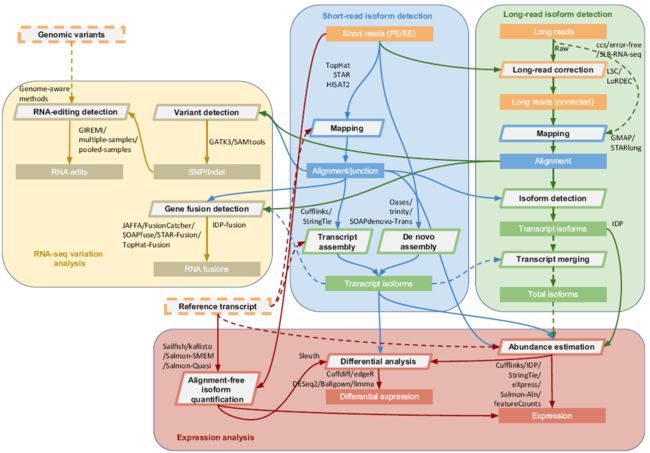
上次转录组培训后,还有不少新朋友希望能再开一期。5月份又恰逢一个在美国做三代测序分析 (PacBio和Nanopore)的朋友回国,机会难得,就决定在5月12日-20日在北京鼓楼再开一期,前两天二代转录组测序分析,后两天三代转录组测序分析。
这么多的内容看似与我们之前推崇的内容要少,学会是王道,有些不符。但多次培训下来,发现大家的接受能力都比较强,而且在微生物组扩增子培训时, 我们探索了一个更好的方式,保证在内容多的时候,也可以不同层次的朋友学会。
我以前推崇学习就要敲代码,我们在教授基础课时都是现场讲解思路、现场敲代码、现场调试,演示整个程序的编写、调试过程,给大家感受写程序的过程。而且助教老师和学员们会临时提出很多问题和需求,现场一起去解决,在这个过程中都能学到很多新知识和新用法。我们第一期转录组也是这么进行的,当时设置的内容比较少,整个学习下来很扎实。
但后来发现,这个对基础课如(Linux, R, Python)效果更好;而后面的高级分析课,尤其是对基础薄弱的朋友,更重要的是能理解整个分析过程,运行流程得到分析结果,把流程带走应用到自己的数据。而且大家把时间花在敲代码上,效率就会低一些 (不过如果想做生信,这一步总是要做的,回去对着视频和脚本,自己敲,有问题在群里提问,就事半功倍了)。
我们在扩增子课程中第一次探索了提供整个分析流程的方式,一步步讲解一遍流程的细节,一步步运行体验流程,再重头讲一遍流程,反复至少三遍 (之所以说至少,是因为中间也会对多个细节进行多次讲解和讨论),从理论和操作都对流程和结果有了深入理解。而且我们做了很好的封装,如果应用于自己的数据,只需修改下对应的实验设计文件,就可以获得最终的分析图表。
这样不同基础的朋友都可以有自己的收获:
- 基础薄弱些的学会了可以按照要求提供输入获得所有结果,并进行解读;
- 基础好一些的朋友,可以阅读流程里面的脚本怎么写的,跟自己之前的做些比较。分析技能学到一定程度都是有个相互比照,
互相阅读代码,才能更好进步; - 基础再好的,可以在这个基础上,提出更多需求,一起写作拓展流程;
在正在进行的ChIP-seq课程中,软件安装基于Conda,运行环境近乎一键部署;提供了整套分析命令,封装上没有扩增子课程做的好,但总体易用,简单改下样品名字即可用。这个课程也是目前课上讨论比较热烈的课程,一边运行着流程,一边讲解流程并讨论新的解决方式,整个过程下来感觉很充实,头脑风暴+现场实现。数据处理中流程是一方面,细节把握也很重要,这个需要不断讨论才能了解更深。
有了这些基础和尝试,这次同时提供二代和三代转录组分析,并在之前的基础上增加可变剪接分析。相信内容增多,效果也会更好。而且培训后,还可以继续观看培训视频,反复学习。不怕学不会,就怕不去学。
(如果基础薄弱,报名付款成功后,可免费领取基础程序课,做好准备工作, 让程序成为我们的得力工具而不是学习新知识的绊脚石。)
课程简介
请详细阅读课程简介,如果以下内容您全部或部分精通,欢迎报名应聘讲师。
一、转录组的应用、设计和案例分享
- 转录组学研究技术介绍
- 转录组学实验设计和测序原则、注意事项
- 转录组学文章案例分析
- 在线基因表达资源数据库
二、转录组分析流程实战
- 测序数据质量评估和清洗
- 基于比对的差异基因分析
- 不基于比对的差异基因分析
- 转录本组装和选择性剪接分析
- 目标基因富集分析
三、转录组高级分析
- WGCNA基因共表达分析
- WGCNA基因、表型关联分析
- Cytoscape 共表达网络绘制
- 转录组常见图形在线绘制
四、三代测序技术概述
- PacBio和Oxford Nanopore测序的原理
- 三代测序的特点和应用
- 三代测序在转录组研究的优势和案例分享
五、三代测序基本分析流程
- 原始测序序列去除接头和错误序列
- 提取环形一致序列读长(CCS reads)
- CCS reads分类(包括全长和非全长CCS reads)
- CCS reads聚类(根据CCS reads序列的相似性)获得最终的转录本集合
- 最终转录本比对回基因组
- 转录本定量和可变剪接分析
(不同版本会有细微差别,具体流程以实战专家讲述为主。)
如果您有其它关注的问题,也请报名时提出,把这次课程变成您的定制讲解。
主讲教师
主讲老师包括爱荷华大学、中科院微生物所、遗传发育所、基因组所、生物物理所等多名本领域一线技术专家。
助教团队
十余名科学院、清华、北大博士(含在读),轮值讲师和助教,辅助学员学习和矫正培训过程中不足的点。
培训时间
2018-05-12 到 2018-05-13 (主要是二代测序)
2018-05-19 到 2018-05-20 (主要是三代和二三混合)
独创线下集中授课2天+自行练习5天+再集中讲解答疑2天+后期学习群的四段式教学,并提供学习视频,教、学、练、答结合,真正实现独立分析大数据。
每天早9点到晚5点,半封闭式教学
报到时间:上课当天。
授课地点
北京市西城区鼓楼明德大厦 (北京市旧鼓楼大街47号院2号楼2010)。
课程价格
- 限时优惠4199 元/人
之后恢复原价6999元/人 (住宿自行解决,提供培训期间午餐) - 名额有限,每次课程报名满30人后自动关闭报名通道
- 提供易汉博基因科技实习机会或工作机会
促销优惠活动
- 座位按报名并成功缴费顺序从前到后龙摆尾式排序
- 赠送价值188元线上生信基础课程一门,目前的《应用Python处理生物信息数据和作图》、《生物信息作图系列R、Cytoscape及图形排版》和《生物信息中的Linux应用》任选其一。(http://bioinfo.ke.qq.com)
- 多人(N,10>N>1)组团报名并同时缴费,每人还可获得价值N百元的礼品(充值或购物卡)。
*注意事项
- 需自备笔记本电脑,系统不限(推荐使用Windows 10)。课程实践根据需要,我们会提供云计算平台。
- 培训班所有数据,文档为内部资料,仅供参阅,未经允许不得翻印外传登刊。
- 上课期间禁止录音,录像。
- 成功付款的学员,若临时有紧急事情不能到来的,可申请延期,更换后续培训班;也可申请退款。
- 若开课2周 (含) 前申请退款可退还85%费用;开课3个工作日 (含) 前申请退款退还70%的费用。
- 不可先延期再退款。
更多课程的详细介绍,请扫描下方二维码。
![]()
复制以下链接
http://www.ehbio.com/Training/ 或
点击阅读原文跳转报名页
成为实验中不可或缺的人,赶快报名吧!
- NGS基础 - FASTQ格式解释和质量评估
- NGS基础 - 高通量测序原理
- NGS基础 - 参考基因组和基因注释文件
- NGS基础 - GTF/GFF文件格式解读和转换
- 本地安装UCSC基因组浏览器
- 测序数据可视化 (一)
- IGV基因组浏览器可视化高通量测序数据
- 测序文章数据上传找哪里
- GO、GSEA富集分析一网打进
- GSEA富集分析 - 界面操作
- 去东方,最好用的在线GO富集分析工具
- 生信软件系列 - NCBI使用