MATLAB-绘制网络拓扑结构
MATLAB-绘制网络拓扑结构
- ①星形拓扑结构
- 一、定义
- 二、.m文件
- 三、 运行结果
- ②树形拓扑结构
- 一、定义
- 二、.m文件
- ==1.第一种方式==
- ==2.第二种方式==
- 三、运行结果
- ==1.第一种方式==
- ==2.第二种方式==
- 总结
参考文档连接:
1.拓扑结构的介绍
2.Matlab绘制树形图
3.Matlab–biograph() 函数
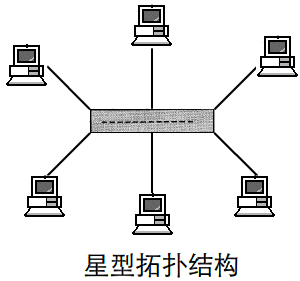
①星形拓扑结构
一、定义
星型拓扑结构是一个中心,多个分节点。它结构简单,连接方便,管理和维护都相对容易,而且扩展性强。网络延迟时间较小,传输误差低。中心无故障,一般网络没问题。中心故障,网络就出问题,同时共享能力差,通信线路利用率不高。
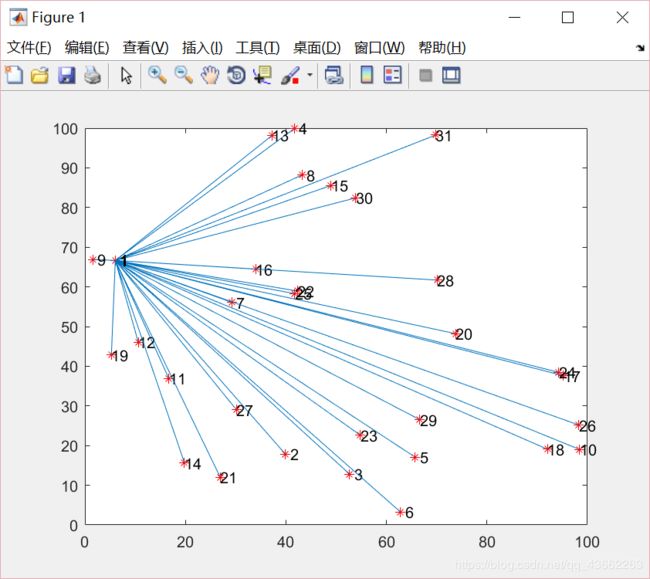
二、.m文件
%% 随机生成31个点
x=100*rand(31,1);
y=100*rand(31,1);
plot(x,y,'r*');
hold on;
%% center代表中心节点一个,children代表子节点30个
for center=1:1
for children=2:31
line([x(center,1),x(children,1)],[y(center,1),y(children,1)]) %将中心节点与所有的子节点连线
text(x(center,1),y(center,1),sprintf('%2.0f',center)) %备注出中心节点的序号
text(x(children,1),y(children,1),sprintf('%2.0f',children)) %备注出子节点的序号
end
end
三、 运行结果
②树形拓扑结构
一、定义
树形拓扑结构从总线拓扑演变而来。形状像一棵倒置的树,顶端是树根,树根以下带分支,每个分支还可再带子分支,树根接收各站点发送的数据,然后再广播发送到全网。好扩展,容易诊断错误,但对根部要求高。

二、.m文件
1.第一种方式
利用biograph函数生成树
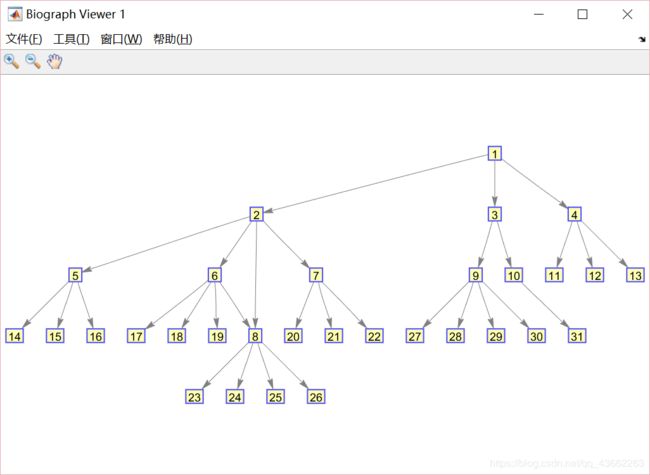
%% 创建31个节点
for n=1:31
ChannelName{n} = sprintf('%2.0f',n);
end
disp(ChannelName);
%% 画树状拓扑图
tree = [1 2;1 3;1 4;2 5;2 6;2 7;
3 9;3 10;4 11;4 12;4 13;5 14;5 15;5 16;
6 17;6 18;6 19;7 20;7 21;7 22;2 8;
8 23;8 24;8 25;8 26;9 27;9 28;9 29;9 30;10 31;6 8;];
cm = zeros(31);
for i = 1:31
cm(tree(i,1),tree(i,2)) = 1; %CM是这个图的邻接矩阵
end
bg1 = biograph(cm,ChannelName);
view(bg1);
- biograph函数生成一个bioinformatics图对象。
- BG = biograph(CM,IDs) 语句设置节点的序号名称。①IDs可以使一个元胞数组,数组中每个元素表示一个名字,数组长度与CM矩阵行列长度一致。IDs也可以是一个字符数组(此时各个节点的名字长度相同)。IDs必须是唯一的,不能重复。②参数CM是这个图的邻接矩阵(CM可以是稀疏矩阵形式表示也可以是一般方阵形式)。
- 具体注解见参考文档链接
2.第二种方式
利用treeplot、treelayout函数
(如果你们用第一种方式biograph函数,运行出现’biograph’ requires Bioinformatics Toolbox 错误,出现这个错误是因为你的matlab没有Bioinformatics Toolbox工具包)
%用treeVec放树节点一共33个节点,数组每位存放的的内容是对应节点它的父节点
%如数组前三位,0 1 1,
% 0代表节点1的父节点为0
% 0后面的1代表节点2的父节点为1
% 1后面的1代表节点3的父节点为1,依次类推
treeVec = [0 1 1 1 1 1 2 2 2 2 3 3 3 3 4 4
4 4 5 5 5 5 6 6 6 6 6 7 7 9 9 10 10];
%treeplot 在给定父指针向量的情况下绘制树形图
treeplot(treeVec);
count = size(treeVec,2);
%treelayout设置树或森林的布局
[x,y] = treelayout(treeVec);
x = x';
y = y';
%显示节点名
name1 = cellstr(num2str((1:count)'));
text(x(:,1),y(:,1),name1,'VerticalAlignment','bottom','HorizontalAlignment','right');
三、运行结果
1.第一种方式
biograph函数生成树
2.第二种方式
总结
- 虽然想要的拓扑结构图大致呈现出来了,但是算法有点暴力,不是很便捷,效率不是很高,尤其体系在树形拓扑结构图那,因为所需的邻接矩阵是手动设置的,那对于有大量节点需求的应用是不适合的,运用起来会很繁琐。
- 欢迎大家来交流,提供更好的解法,去改进脚本!