机器学习(十)-逻辑回归实践篇之乳腺癌肿瘤预测
逻辑回归实践篇之乳腺癌肿瘤预测
- 1 项目描述
- 2 项目分析
- 2.1 数据预处理
- 2.2 数据可视化
- 2.3 拟合模型(非训练型)
- 2.4 拟合模型(样本训练)
- 2.4 完整源代码
- 3 参考博客
注: 本篇博客为《Python机器学习及实践:从零通往Kaggle竞赛之路》一书逻辑回归案例的笔记,欢迎与我交流数据挖掘、机器学习相关话题。
1 项目描述
“良/恶性乳腺癌肿瘤预测”的问题属于二分类任务。
待预测的类别分别是良性乳腺癌肿瘤和恶性乳腺癌肿瘤。
通常使用离散的整数来代表类别, 0代表良性,1代表恶性。
2 项目分析
2.1 数据预处理
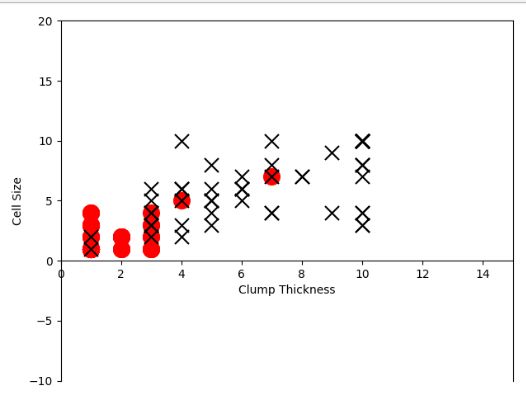
本项目案例中只取这两个特征,通过图像展示肿瘤样本在二维特征空间的分布情况,如下图所示。X代表恶性肿瘤,O代表良性肿瘤。
def load_data():
"""
加载数据集中的数据
:return:
"""
# 将训练集读取进来并存至变量df_train
df_train = pd.read_csv('./data/breast-cancer-train.csv')
# 将测试集读取进来并存至变量df_test
df_test = pd.read_csv('./data/breast-cancer-test.csv')
# 选取Clump Thickness(肿瘤厚度)和Cell Size(细胞尺寸)作为特征,构建测试集中的正负分类样本
df_test_negative = df_test.loc[df_test['Type'] == 0][['Clump Thickness', 'Cell Size']]
df_test_positive = df_test.loc[df_test['Type'] == 1][['Clump Thickness', 'Cell Size']]
return df_train, df_test, df_test_negative, df_test_positive
if __name__ == '__main__':
df_train, df_test, df_test_negative, df_test_positive = load_data()
print(df_train)
print(df_test)
print(df_test_negative)
print(df_test_positive)
2.2 数据可视化
本项目案例中只取这两个特征,通过图像展示肿瘤样本在二维特征空间的分布情况,如下图所示。X代表恶性肿瘤,O代表良性肿瘤。
def configure_plt(plt):
"""
配置图形的坐标表信息
"""
# 获取当前的坐标轴, gca = get current axis
ax = plt.gca()
# 设置x轴, y周在(0, 0)的位置
ax.spines['bottom'].set_position(('data', 0))
ax.spines['left'].set_position(('data', 0))
# 设置坐标轴的取值范围
plt.xlim((0, 15))
plt.ylim((-10, 20))
# 绘制x,y轴说明
plt.xlabel('Clump Thickness')
plt.ylabel('Cell Size')
return plt
def draw_pic():
"""
绘制恶性肿瘤和良性肿瘤的图形
:return:
"""
df_train, df_test, df_test_negative, df_test_positive = load_data()
import matplotlib.pyplot as plt
# 绘制图中的良性肿瘤样本点,标记为红色的o
plt.scatter(df_test_negative['Clump Thickness'], df_test_negative['Cell Size'], marker='o', s=200, c='red')
# 绘制图中的恶心肿瘤样本点,标记为黑色的x
plt.scatter(df_test_positive['Clump Thickness'], df_test_positive['Cell Size'], marker='x', s=150, c='black')
plt = configure_plt(plt)
# 显示图
plt.show()
if __name__ == '__main__':
draw_pic()
2.3 拟合模型(非训练型)
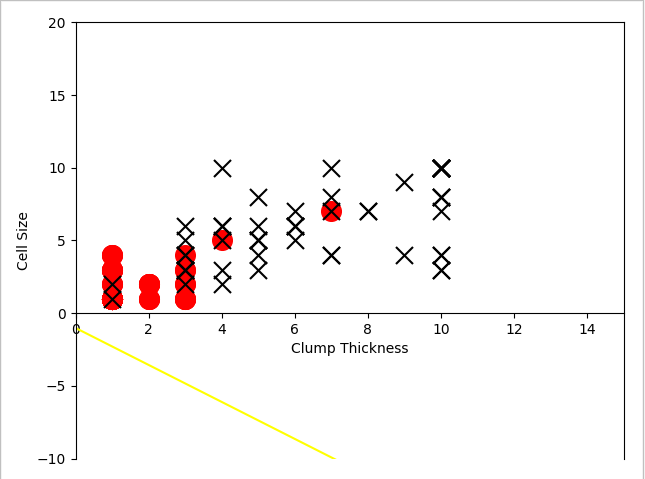
随后我们随机初始化一个二类分类器,这个分类器用一条直线来划分良/恶性肿瘤。决定这条直线的走向有两个因素:直线的斜率和截距。这些被我们称为模型的参数,也是分类器需要通过学习从训练数据中得到的。最初,随机初始化参数的分类器的性能表现如下图所示:
def init_model():
"""
初始化模型的参数,
随机初始化一个二类分类器,这个分类器用一条直线来划分良/恶性肿瘤。决定这条直线的走向有两个因素:直线的斜率和截距。
:return:
"""
# 利用numpy中的random函数随机采样直线的截距和系数
intercept = np.random.random([1])
coef = np.random.random([2])
lx = np.arange(0, 12)
ly = (-intercept - lx * coef[0]) / coef[1]
return lx, ly
def draw_pic():
"""
绘制恶性肿瘤和良性肿瘤的图形
:return:
"""
df_train, df_test, df_test_negative, df_test_positive = load_data()
import matplotlib.pyplot as plt
# 绘制图中的良性肿瘤样本点,标记为红色的o
plt.scatter(df_test_negative['Clump Thickness'], df_test_negative['Cell Size'], marker='o', s=200, c='red')
# 绘制图中的恶心肿瘤样本点,标记为黑色的x
plt.scatter(df_test_positive['Clump Thickness'], df_test_positive['Cell Size'], marker='x', s=150, c='black')
# **************************************************新加的部分******************
# 绘制初始化模型参数的随机直线
lx, ly = init_model()
plt.plot(lx, ly, c='yellow')
plt = configure_plt(plt)
# 显示图
plt.show()
if __name__ == '__main__':
draw_pic()
2.4 拟合模型(样本训练)
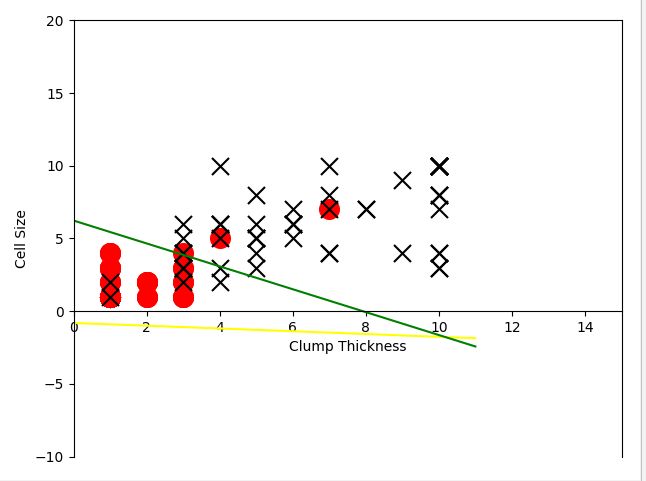
随后我们使用一定量训练样本,分类器所表现的性能有了大幅度的提示,如下图:
def train_model(df_train, df_test):
"""
使用一定量训练样本来训练模型
:return:
"""
import warnings
warnings.filterwarnings('ignore')
lr = LogisticRegression()
# 使用前10条训练样本学习直线的系数和截距
lr.fit(df_train[['Clump Thickness', 'Cell Size']], df_train['Type'])
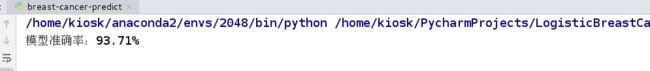
print('模型准确率:%.2f%%' %(lr.score(df_test[['Clump Thickness', 'Cell Size']], df_test['Type'])*100 ))
intercept = lr.intercept_
coef = lr.coef_[0, :]
lx = np.arange(0, 12)
# 原本这个分类面应该是lx*coef[0] + ly*coef[1] + intercept=0 映射到2维平面上之后,应该是:
ly = (-intercept - lx * coef[0]) / coef[1]
return lx, ly
def draw_pic():
"""
绘制恶性肿瘤和良性肿瘤的图形
:return:
"""
df_train, df_test, df_test_negative, df_test_positive = load_data()
import matplotlib.pyplot as plt
# 绘制图中的良性肿瘤样本点,标记为红色的o
plt.scatter(df_test_negative['Clump Thickness'], df_test_negative['Cell Size'], marker='o', s=200, c='red')
# 绘制图中的恶心肿瘤样本点,标记为黑色的x
plt.scatter(df_test_positive['Clump Thickness'], df_test_positive['Cell Size'], marker='x', s=150, c='black')
# 绘制初始化模型参数的随机直线
lx, ly = init_model()
plt.plot(lx, ly, c='yellow')
## **************************************************新加的部分******************
# 训练之后的模型数据
lx, ly = train_model(df_train, df_test)
plt.plot(lx, ly, c='green')
plt = configure_plt(plt)
# 显示图
plt.show()
if __name__ == '__main__':
draw_pic()
2.4 完整源代码
"""
文件名: breast-cancer-predict.py
创建时间: 2019-04-26 10:
作者: lvah
联系方式: [email protected]
代码描述:
项目描述:
“良/恶性乳腺癌肿瘤预测”的问题属于二分类任务。
待预测的类别分别是良性乳腺癌肿瘤和恶性乳腺癌肿瘤。
通常使用离散的整数来代表类别, 0代表良性,1代表恶性。
"""
import pandas as pd
import numpy as np
from sklearn.linear_model import LogisticRegression
def load_data():
"""
加载数据集中的数据
:return:
"""
# 将训练集读取进来并存至变量df_train
df_train = pd.read_csv('./data/breast-cancer-train.csv')
# 将测试集读取进来并存至变量df_test
df_test = pd.read_csv('./data/breast-cancer-test.csv')
# 选取Clump Thickness(肿瘤厚度)和Cell Size(细胞尺寸)作为特征,构建测试集中的正负分类样本
df_test_negative = df_test.loc[df_test['Type'] == 0][['Clump Thickness', 'Cell Size']]
df_test_positive = df_test.loc[df_test['Type'] == 1][['Clump Thickness', 'Cell Size']]
return df_train, df_test, df_test_negative, df_test_positive
def init_model():
"""
初始化模型的参数,
随机初始化一个二类分类器,这个分类器用一条直线来划分良/恶性肿瘤。决定这条直线的走向有两个因素:直线的斜率和截距。
:return:
"""
# 利用numpy中的random函数随机采样直线的截距和系数
intercept = np.random.random([1])
coef = np.random.random([2])
lx = np.arange(0, 12)
ly = (-intercept - lx * coef[0]) / coef[1]
return lx, ly
def train_model(df_train, df_test):
"""
使用一定量训练样本来训练模型
:return:
"""
import warnings
warnings.filterwarnings('ignore')
lr = LogisticRegression()
# 使用前10条训练样本学习直线的系数和截距
lr.fit(df_train[['Clump Thickness', 'Cell Size']], df_train['Type'])
print('模型准确率:%.2f%%' %(lr.score(df_test[['Clump Thickness', 'Cell Size']], df_test['Type'])*100 ))
intercept = lr.intercept_
coef = lr.coef_[0, :]
lx = np.arange(0, 12)
# 原本这个分类面应该是lx*coef[0] + ly*coef[1] + intercept=0 映射到2维平面上之后,应该是:
ly = (-intercept - lx * coef[0]) / coef[1]
return lx, ly
def configure_plt(plt):
"""
配置图形的坐标表信息
"""
# 获取当前的坐标轴, gca = get current axis
ax = plt.gca()
# 设置x轴, y周在(0, 0)的位置
ax.spines['bottom'].set_position(('data', 0))
ax.spines['left'].set_position(('data', 0))
# 设置坐标轴的取值范围
plt.xlim((0, 15))
plt.ylim((-10, 20))
# 绘制x,y轴说明
plt.xlabel('Clump Thickness')
plt.ylabel('Cell Size')
return plt
def draw_pic():
"""
绘制恶性肿瘤和良性肿瘤的图形
:return:
"""
df_train, df_test, df_test_negative, df_test_positive = load_data()
import matplotlib.pyplot as plt
# 绘制图中的良性肿瘤样本点,标记为红色的o
plt.scatter(df_test_negative['Clump Thickness'], df_test_negative['Cell Size'], marker='o', s=200, c='red')
# 绘制图中的恶心肿瘤样本点,标记为黑色的x
plt.scatter(df_test_positive['Clump Thickness'], df_test_positive['Cell Size'], marker='x', s=150, c='black')
# 绘制初始化模型参数的随机直线
lx, ly = init_model()
plt.plot(lx, ly, c='yellow')
## **************************************************新加的部分******************
# 训练之后的模型数据
lx, ly = train_model(df_train, df_test)
plt.plot(lx, ly, c='green')
plt = configure_plt(plt)
# 显示图
plt.show()
if __name__ == '__main__':
draw_pic()
3 参考博客
Python机器学习及实践——简介篇3(逻辑回归)