DADA2中文教程v1.8
文章目录
- DADA2 R包中文使用指南
- 写在前面
- 开始之前
- 准备
- 数据获取
- 定义原始文件路径
- 序列文件质量检测
- 反向序列的质量
- 序列过滤和裁剪
- 错误率
- 去除重复序列
- 基于错误模型进一步质控
- 序列拼接
- 生成ASV表
- 去除嵌合体
- 统计上述分析步骤
- 序列物种注释
- 相关文件保存
- DADA2准确性评估
- 基于R语言DADA2的后续分析
- 译者简介
- 猜你喜欢
- 写在后面
DADA2 R包中文使用指南
DADA2 Pipeline Tutorial
本文翻译自DADA2英文使用指南:http://benjjneb.github.io/dada2/tutorial.html
写在前面
我们将在一个小时内对DADA2(ver 1.8)包进行一个初步系统的学习。本教程使用的是已拆分好的双端illumina测序下机数据,barcode或者接头序列已经被去除。通过本流程我们将得到ASV 表格(amplicon sequence variant table),类似于我们熟悉的OTU table 。如同之前介绍过的DADA2特性,ASV table通过将每条序列及其数量信息记录下来,得到具有比传统聚类得到的OTU table更高分辨率的微生物分类信息。通过注释序列得到物种信息,下游我们将使用目前十分流行的R包phyloseq做下游微生物群落相关分析,尽请期待。
开始之前
本流程假设您的测序数据符合以下规范:
- 样品已被拆分好,即每个样品一个fq/fastq文件(或者双端成对fq文件);
- 已经去除非生物核酸序列,比如:引物(primers),接头(adapters or barcodes),linker等;
- 如果样品是下机的双端测序,其应具有双端测序的相匹配的两个fq文件。
注意: 如果您的数据并不符合这些规范,那么需要您在开始本流程前解决这些问题,有关这类问题的建议,请参阅官方网站常见问题解答( http://benjjneb.github.io/dada2/faq.html ).
准备
首先我们需要载入dada2包,如果您在此之前并没有安装成功,参见dada2安装指南(http://benjjneb.github.io/dada2/dada-installation.html) 本来我准备跳过这个步骤,但是年底我一直在回家的途中,拿的是一台新买的超极本给大家写教程,机器上并没有配置好dada2包,因此,这里和没有准备好这一环境的朋友们一起安装。
下面附上安装代码:
# 国内用户清华镜像站,加速国内用户下载
site="https://mirrors.tuna.tsinghua.edu.cn/CRAN"
# 检查是否存在Biocondoctor安装工具,没有则安装
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager",repo=site)
# 加载安装工具,并安装dada2
library(BiocManager)
BiocManager::install("dada2", version = "3.8")
# 需要显示版本信息
library(dada2)
packageVersion("dada2")
备注:dada2的其他版本(ver. 1.2 1.4 1.8)均可同步实现该流程。
数据获取
官方地址:https://www.mothur.org/w/images/d/d6/MiSeqSOPData.zip
本翻译教程全套流程文件亦包含所下载的原始数据
测试数据说明:这些肠道微生物测序样品数据来自断奶后的小鼠和一个是对照模拟群落(Illumina MiSeq,16S rRNA基因的2x250,V4区域),
定义原始文件路径
定义工作路径,显示文件夹内容,这里我将作者的linux路径更换为win下的路径;
# 设置数据所在目录,请设置为你下载解压的目录
# CHANGE ME to the directory containing the fastq files after unzipping.
path <- "C:/Users/woodc/Desktop/home/markdown/blog/wentao/MiSeq_SOP"
list.files(path)
通过字符串操作函数提取单个样品测序文件信息
# list.files返回指定目录中的文件名
# pattern指定返回指定类型的文件
# full.names = TRUE返回带有路径的完整文件名
# 返回测序正向文件完整文件名
fnFs <- sort(list.files(path, pattern="_R1_001.fastq", full.names = TRUE))
# 返回测序反向文件完整文件名
fnRs <- sort(list.files(path, pattern="_R2_001.fastq", full.names = TRUE))
# Extract sample names, assuming filenames have format: SAMPLENAME_XXX.fastq
# basename(fnFs)提取文件名(不要目录)
# strsplit按`_`进行分割字符,返回列表
# sapply批量操作,这里批量提取列表中第一个元素,即样本名
# 提取文件名中`_`分隔的第一个单词作为样品名
sample.names <- sapply(strsplit(basename(fnFs), "_"), `[`, 1)
序列文件质量检测
绘制前4个样本的质量示意图
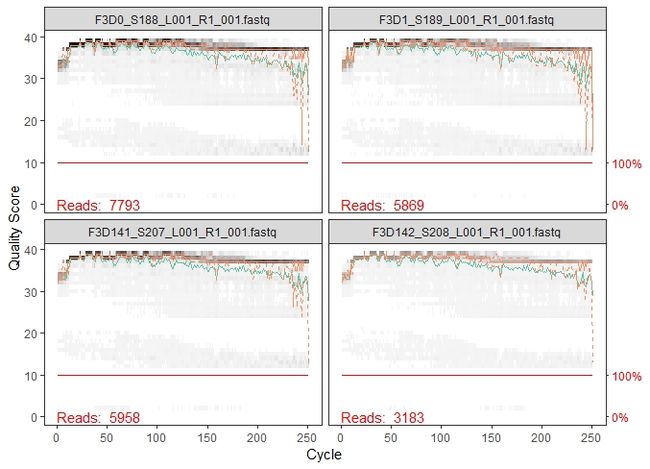
plotQualityProfile(fnFs[1:4])
这是一张热图,基于碱基的质量信息的频率分布所得,每个位置的中位数质量得分由绿线表示,质量得分的四分位数由橙色线表示。红线显示扩展至该位置序列长度的比例(这对于其他测序技术更有用,如454测序。因为Illumina读数通常都是相同的长度,因此是扁平的红线)。正向序列质量很好。我们通常建议修剪最后几个核苷酸序列,最后几个碱基错误率往往比较高。我们将截取/保留前240位的正向序列(剪去最后10个核苷酸)。
反向序列的质量
现在我们可视化反向序列的质量概况
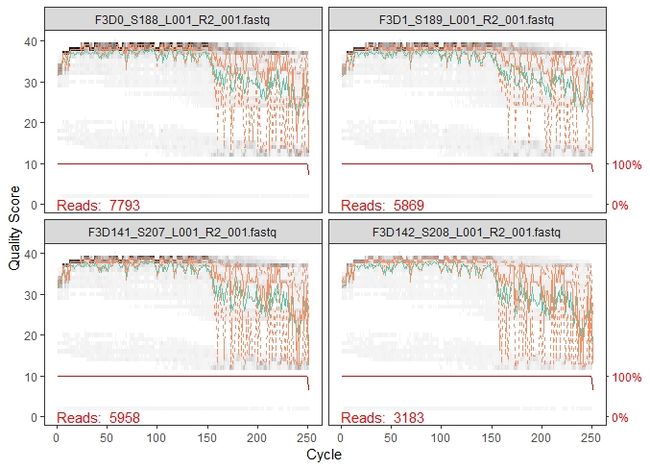
plotQualityProfile(fnRs[1:4])
反向序列质量明显较差,特别是在最后,这在Illumina测序中很常见。但这对于我们的DADA2 流程影响不大。虽然DADA2将质量信息整合到其误差模型中,但是这使得算法对低质量序列具有鲁棒性,所以我们从平均质量值突降的位置进行剪切会提高算法对稀有序列的敏感性。因此,我们对反向序列文件裁剪去至160bp。
注意:当我们在处理自己的数据时,不仅仅需要根据质量突降的位置进行裁剪位置的选择,首先我们必须确定测序的双端序列必须拥有足够的重叠(overlap),至少保证裁剪之后在 20 bp 以上,以保证拼接效率。
序列过滤和裁剪
首先对序列进行质量过滤剪切。我们通过filterAndTrim命令对正向序列和反向序列分别裁剪至240和160 bp(truncLen=c(240,160)),最大N的数量设置为0(maxN=0),maxEE参数表示允许在条reads中期望错误的最大值(参见“一种比平均质量更好的过滤策略”:https://academic.oup.com/bioinformatics/article/31/21/3476/194979 );truncQ为过滤最最质量的阈值,默认为2,将去除低于此质量的序列;rm.phix默认为TRUE,将去除比对上参考PhiX基因组的序列,在扩增子中常用;compress,默认输出压缩格式的结果;multithread为默认单线程,可以改为T或整数提高速度,但确保你电脑有足够的内存和计算资源。
# Place filtered files in filtered/ subdirectory
# 将过滤后的文件存于filtered子目录,设置输出文件名
filtFs <- file.path(path, "filtered", paste0(sample.names, "_F_filt.fastq.gz"))
filtRs <- file.path(path, "filtered", paste0(sample.names, "_R_filt.fastq.gz"))
# 过滤文件输出,输出和参数,统计结果保存于Out
out <- filterAndTrim(fnFs, filtFs, fnRs, filtRs, truncLen=c(240,160),
maxN=0, maxEE=c(2,2), truncQ=2, rm.phix=TRUE,
compress=TRUE, multithread=T)
head(out)
显示过滤前后的结果统计
reads.in reads.out
F3D0_S188_L001_R1_001.fastq 7793 7113
F3D1_S189_L001_R1_001.fastq 5869 5299
F3D141_S207_L001_R1_001.fastq 5958 5463
F3D142_S208_L001_R1_001.fastq 3183 2914
F3D143_S209_L001_R1_001.fastq 3178 2941
F3D144_S210_L001_R1_001.fastq 4827 4312
注意事项1:以上设置的过滤参数并不是不变的,如果我们需要缩短过滤时间,maxEE参数设置更小一些,想要保留多数的reads,那就需要对maxEE参数设置的更大些,尤其是反向测序数据(例如maxEE=c(2,5))。另外,在设置trunclen参数时一定要注意在裁剪之后的序列能保留足够的长度(最低20bp)来用于拼接。
注意事项2:对于ITS测序结果,序列长度变化较大,可以考虑不进行裁剪,但是要确保引物在这之前已经被去除干净。
错误率
下面我们来了解错误率
DADA2算法使用机器学习构建参数误差模型,认为每个扩增子测序样品都具有不同的误差比率。该learnErrors方法通过交替估计错误率和对参考样本序列学习错误模型,直到学习模型同真实错误率收敛于一致。与许多机器学习方法一样,算法有一个初始假设:数据中的最大可能错误率就是只有最丰富的序列是正确的,其余都是错误。(在我这台小笔记本上运行用了6分钟)
这是DADA2中运行最耗费计算资源的一步
errF <- learnErrors(filtFs, multithread=TRUE)
errR <- learnErrors(filtRs, multithread=TRUE)
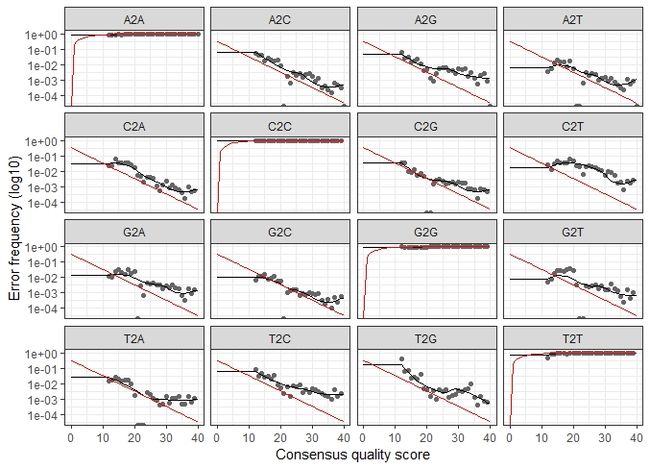
plotErrors(errF, nominalQ=TRUE)
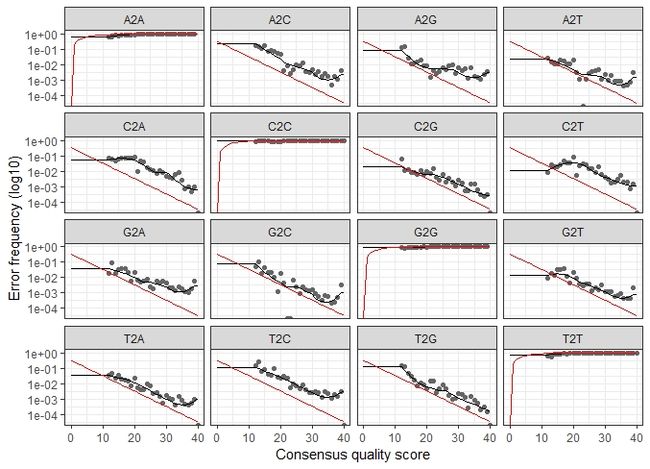
plotErrors(errR, nominalQ=TRUE)
可视化结果表示了每种可能的错误(A→C,A→G,…),图中点表示观察得到的错误率。黑线表示通过算法学习评估得到的错误率。红色的曲线表示由Q-score的定义下预期的错误率。这里估计的错误率(黑线)同观察到的错误率(点)拟合程度很好,并且错误率随着预期的质量而下降。这和我们的认知相符。
注意事项:DADA2核心算法亦是参数学习,计算量非常可观,目前我们测得的数据大小为本例子文件的十倍甚至一百倍,面对如此巨大的数据和需要消耗的计算资源,这一模型的展示便不适合我们实际较大的数据量,可以通过增加nbase参数调整拟合程度以减少计算量。
去除重复序列
与usearch去冗余步骤类似,仅仅保留重复序列中的一条序列,大量节省计算资源。对于在此使用小笔记本为大家翻译教程的我十分受用。
在这里值得注意的是DADA2保留了去重序列的质量信息,这些质量信息取自重复序列的均值。这一信息文件将作为参考错误模型用于后续序列处理,以提高了DADA2算法准确性。
derepFs <- derepFastq(filtFs, verbose=TRUE)
derepRs <- derepFastq(filtRs, verbose=TRUE)
注意事项:如果您的数据量很大,可能需要使用别的策略更为妥当(big data使用DADA的流程参考:http://benjjneb.github.io/dada2/bigdata.html 目前链接无法打开,可能作者在更新)
基于错误模型进一步质控
dadaFs <- dada(derepFs, err=errF, multithread=TRUE)
dadaRs <- dada(derepRs, err=errR, multithread=TRUE)
dadaFs[[1]]
DADA2算法从第一个样本中的1979个独特序列推断出128个真实序列。dada-class返回对象还有许多(参见help(“dada-class”)参考资料),包括关于每个去噪序列质量的多个评价指标,本教程不做解释。
注意事项1:在教程中,所有样本都同时加载到内存中。如果您正在处理接近或超过可用内存(RAM)的数据集(如今我们的电脑内存普遍4或8G,据我测试,8G内存可处理18个样品,每个样品大小为20M,也就是两个实验组),样品数量较多时,我们需要逐个处理样本:请参阅 DADA2大数据工作流程( http://benjjneb.github.io/dada2/bigdata.html )。
注意事项2:DADA2同时支持454和IonTorrent数据,但是建议对其中一些参数进行修改,可以通过R语言中?setDadaOpt来调取帮助文件,探索这些参数的修改方法。(如有需求,欢迎留言交流)
序列拼接
我们现在将正向和反向序列文件合并在一起以获得完整的序列。通过将去噪的正向序列与相应的去噪反向序列的反向互补序列比对,然后构建合并的“overlap”进行合并。默认情况下,仅当正向和反向序列重叠至少12个碱基并且在重叠区域中彼此相同时才输出合并序列。
mergers <- mergePairs(dadaFs, derepFs, dadaRs, derepRs, verbose=TRUE)
# Inspect the merger data.frame from the first sample
head(mergers[[1]])
mergers对象格式为R语言的数据框,数据框中包含序列及其丰度信息,未能成功合并的序列被删除。
生成ASV表
我们开始构建 amplicon sequence variant(ASV)表,;类似于我们传统的OTU表一样。
seqtab <- makeSequenceTable(mergers)
dim(seqtab)
# 查看序列长度分布
# Inspect distribution of sequence lengths
table(nchar(getSequences(seqtab)))
去除嵌合体
Dada核心质控方法纠正了一些错误,但嵌合体仍然存在。幸运的是,去噪后序列的准确性使得识别嵌合体比处理模糊OTU时更简单。
seqtab.nochim <- removeBimeraDenovo(seqtab, method="consensus", multithread=TRUE, verbose=TRUE)
dim(seqtab.nochim)
sum(seqtab.nochim)/sum(seqtab)
嵌合体的数量历来被大家讨论过很多次,因为不同实验,不同样品等等很多的因素都于嵌合体数量有关,这里我们得到的嵌合体数量大概为21%,但是这些嵌合体占据的序列数量大部分都是很少的,在这里这些嵌合体仅仅占我们全部序列数量的3.6%。
注意事项: 在进行嵌合体去除操作后大部分序列应该被保留下来。也有可能大部分序列作为嵌合体被去除,但这样情况并不多见。如果这种情况发生,我们只有返回上游进行重新处理序列,大部分这种情况都是由于引物序列未能去除干净导致的。
统计上述分析步骤
以上步骤序列可算是进行了重重筛选,这些序列保留及其去除的数量信息需要做一个记录才好。
# 求和函数
getN <- function(x) sum(getUniques(x))
# 合并各样本分步数据量
track <- cbind(out, sapply(dadaFs, getN), sapply(dadaRs, getN), sapply(mergers, getN), rowSums(seqtab.nochim))
# If processing a single sample, remove the sapply calls: e.g. replace sapply(dadaFs, getN) with getN(dadaFs)
# 修改列名
colnames(track) <- c("input", "filtered", "denoisedF", "denoisedR", "merged", "nonchim")
# 行名修改为样本名
rownames(track) <- sample.names
# 统计结果预览
head(track)
预览前6个样本各步过滤和处理前后数据量统计
input filtered denoisedF denoisedR merged nonchim
F3D0 7793 7113 6976 6979 6540 6528
F3D1 5869 5299 5227 5239 5028 5017
F3D141 5958 5463 5331 5357 4986 4863
F3D142 3183 2914 2799 2830 2595 2521
F3D143 3178 2941 2822 2867 2552 2518
F3D144 4827 4312 4151 4228 3627 3488
序列物种注释
这里我们的16s序列注释,采用下载Silva参考数据库进行训练和注释,DADA2包为此目的提供了朴素贝叶斯分类器方法实现物种注释。该assignTaxonomy函数将一组要分类的序列和具有已知分类的参考序列训练集作为输入,并输出的注释文件通过bootstrap检验。
首先从页面 http://benjjneb.github.io/dada2/training.html 下载相应数据库,有16S可选 Silva 132/128/123,RDP trainset 16/14, Greengene 13.8;真菌ITS必选UNITE。这里我们使用Silva 132,需要下载序列训练集和物种注释两个文件。
保存文件至工作目录。其它位置请修改下面代码中path变量
taxa <- assignTaxonomy(seqtab.nochim, paste0(path, "/silva_nr_v132_train_set.fa.gz"), multithread=TRUE)
taxa <- addSpecies(taxa, paste0(path, "/silva_species_assignment_v132.fa.gz"))
taxa.print <- taxa
# 另存物种注释变量,去除序列名,只显示物种信息
# Removing sequence rownames for display only
rownames(taxa.print) <- NULL
head(taxa.print)
不出所料,在这些粪便样本中最丰富的是Bacteroidetes,但是仅仅有少数被注释到种的水平,因为通常不可能从16S基因的一段进行确定的物种分配,并且可能因为参考数据库中对小鼠肠道微生物群的覆盖率极低。
另外:这里提到最近开发的IdTaxa物种注释分类方法也可通过 DECIPHER Bioconductor包获得。IDTAXA算法的论文表示其分类性能优于朴素贝叶斯分类器。这里我们包含一段代码区域,允许你使用IdTaxa函数替代assignTaxonomy(并且它也更快!)。经过训练的分类器可从 http://DECIPHER.codes/Downloads.html 获得(可能需要)。下载SILVA SSU r132文件以继续。(本教程未进行相关操作,各位有需求亲留言交流)
# 安装DECIPHER进行物种注释
BiocManager::install("DECIPHER", version = "3.8")
library(DECIPHER); packageVersion("DECIPHER")
# 转换ASV表为DNAString格式
dna <- DNAStringSet(getSequences(seqtab.nochim)) # Create a DNAStringSet from the ASVs
# 相关数据下载详见DECIPHER教程,并修改为下载目录
# http://DECIPHER.codes/Downloads.html 获得(可能需要)
load(paste0(path, "/SILVA_SSU_r132_March2018.RData")) # CHANGE TO THE PATH OF YOUR TRAINING SET
ids <- IdTaxa(dna, trainingSet, strand="top", processors=NULL, verbose=FALSE) # use all processors
ranks <- c("domain", "phylum", "class", "order", "family", "genus", "species") # ranks of interest
# Convert the output object of class "Taxa" to a matrix analogous to the output from assignTaxonomy
taxid <- t(sapply(ids, function(x) {
m <- match(ranks, x$rank)
taxa <- x$taxon[m]
taxa[startsWith(taxa, "unclassified_")] <- NA
taxa
}))
colnames(taxid) <- ranks; rownames(taxid) <- getSequences(seqtab.nochim)
taxa.print <- taxa <- taxid# Removing sequence rownames for display only
rownames(taxa.print) <- NULL
head(taxa.print)
# 注:此处我没有运行成功,读入RData后R环境崩溃了,个人感觉R语言处理大文件非常不擅长
注意事项:如果您的数据没有被适当注释,例如您的细菌16S序列被分配为大量Eukaryota NA NA NA NA NA, 可能核苷酸序列方向与参考数据库的方向相反。告诉dada2尝试反向互补方向进行匹配,assignTaxonomy(…,tryRC=TRUE)看看这是否可以修复注释信息。如果使用DECIPHER进行分类,请尝试IdTaxa (…, strand=“both”)。
相关文件保存
setwd(path)
seqtable.taxa.plus <- cbind('#seq'=rownames(taxa), t(seqtab.nochim), taxa)
# ASV表格导出
write.table(seqtab.nochim, "dada2_counts.txt", sep="\t", quote=F, row.names = T)
# 带注释文件的ASV表格导出
write.table(seqtable.taxa.plus , "dada2_counts.taxon.species.txt", sep="\t", quote=F, row.names = F)
# track文件保存
write.table(track , "dada2_track.txt", sep="\t", quote=F, row.names = F)
DADA2准确性评估
本教程的最后我们来评估DADA2的准确性
在群落中我们包含了一个模拟的微生物群落,这是对目前已知的20株菌的混合测序样品。并且已经提供了这些菌的真实注释信息,我们使用DADA2进行推断和真正的注释信息进行比较,从而评估其注释准确性。
这一人工群落包含20株菌DADA2确定了20个 ASV,所有这些ASV都与预期的群落成员的参考基因组完全匹配。此样本的DADA2分析之后的残留错误率为0%。
unqs.mock <- seqtab.nochim["Mock",]
unqs.mock <- sort(unqs.mock[unqs.mock>0], decreasing=TRUE) # Drop ASVs absent in the Mock
cat("DADA2 inferred", length(unqs.mock), "sample sequences present in the Mock community.\n")
mock.ref <- getSequences(file.path(path, "HMP_MOCK.v35.fasta"))
match.ref <- sum(sapply(names(unqs.mock), function(x) any(grepl(x, mock.ref))))
cat("Of those,", sum(match.ref), "were exact matches to the expected reference sequences.\n")
Dada2 piplines到此结束
我们得到了ASV表格,类似于传统OTU 表格,注释信息,传统的代表序列文件在这里的表现形式在ASV 表中的行名。我们除了没有构建进化树,基于扩增子的前处理就到此结束了。
基于R语言DADA2的后续分析
这里我们在R语言中有一整套的解决方案,尽请期待后续:
++基于R语言phyloseq包的扩增子下游分析策略++。
译者简介
个人简历:文涛,2016年就读于南京农业的大学。在资源院沈其荣教授课题组,研究方向为根际微生物生态,具体为植物介导下根际小分子代谢组同土壤微生物群落在防控土传病害方面的相互作用。2018年9月转博,期间个人主要完成了一篇发表于Microbiome的文章内容(三作,一二做作为小导师及其合作关系)。题目为:Root exudates drive the soil-borne legacy of aboveground pathogen infection(https://doi.org/10.1186/s40168-018-0537-x) ;本人一作已投文章一篇,在写文章一篇,两篇文章主要数据均为代谢组和扩增子测序获得。
技能:掌握扩增子测序传统数据处理及其下游分析;会使用Qiime2,usearch, R等工具进行新一代测序数据处理及其部分下游分析。。想通过coding的学习或者大家的帮助将分析过程流程化。会基于R语言的非靶向代谢组学下游分析技术。
现状:目前在跟进硕士内容,并加入根际功能方面的内容(宏基因组),其中水稻相关的根际宏基因组样品与12月送样,希望熟练掌握宏基因组数据处理流程。同时希望完善代谢组学分析技术和基于扩增子和代谢组的大数据整合技术。
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA