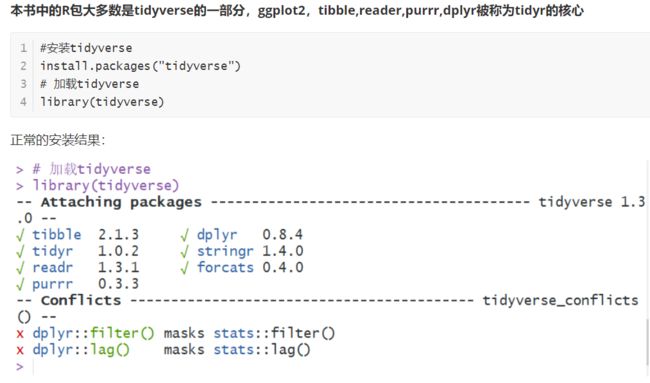
今天学习与R包相关的内容今天起了个大早,因为英语老师在美国有时差
一、配置Rstudio的下载镜像
R的配置文件 .Rprofile
在刚开始运行Rstudio的时候,程序会查看许多配置内容,其中一个就是.Renviron,它是为了设置R的环境变量(这里先不说它);而.Rprofile就是一个代码文件,如果启动时找到这个文件,那么就替我们先运行一遍(这个过程就是在启动Rstudio时完成的) 来自:https://mp.weixin.qq.com/s/XvKb5FjAGM6gYsxTw3tcWw生信星球
file.edit('~/.Rprofile') #首先用file.edit()来编辑文件,这时候Rstudio会自动打开一个新的窗口
然后在新打开的窗口输入以下两行代码:
# options函数就是设置R运行过程中的一些选项设置
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/")) #对应清华源
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/") #对应中科大源
# 当然可以换成其他地区的镜像
然后点击保存,save,最后重启以下Rstudio,再运行一下:
options()$repos和options()$BioC_mirror 就发现已经配置好了,就很方便地省了手动运行的步骤
二、R包安装和加载
R包安装命令是install.packages(“包”)或者BiocManager::install(“包”)。取决于你要安装的包存在于CRAN网站还是Biocductor,存在于哪里?下面介绍2种R包安装方式(划重点啦):
1、第一种方式,CRAN安装R包:install.packages()函数
这种方式是R自带的函数,直接安装包了,这个是最简单的,而且不需要考虑各种包之间的依赖关系。
install.packages("ggplot2") ##直接输入包名字即可
library("ggplot2") #加载一下即可使用
2、第二种方式,Bioconductor安装R包:BiocManager::install()
> if (!requireNamespace("BiocManager", quietly = TRUE))#用来进行一个特定的行为,如当建议包找不到时抛出一个错误。
> install.packages("BiocManager") # 首先要安装BiocManager包
> BiocManager::install("Biobase") # 再用BiocManager安装
参考:https://www.jianshu.com/p/98cb8607a731
- R包的加载:
library(包)或require(包)两个都可以
总结:安装加载三部曲(以dplyr包为例)
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
install.packages("dplyr") #先下载包
library(dplyr) #加载一下即可使用
三、dplyr五个基础函数
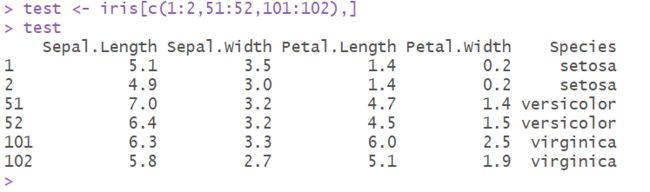
- 示例数据直接使用内置数据集iris的简化版:
test <- iris[c(1:2,51:52,101:102),] #取鸢尾花数据集的第1-2、51-52、101-102行
1.mutate(),新增列
2.select(),按列筛选
-
(1)按列号筛选
*(2)按列名筛选
- 3.
filter(),筛选行
- 4.
arrange(),按某1列或某几列对整个表格进行排序
- 5.
summarise():汇总
- 对数据进行汇总操作,结合
group_by使用实用性强- dplyr提供了一个分组函数group_by,把分组依据相同的数据组合成行,相当于ddply中的
group_vars- ungroup用于移出数据框的分组信息。配合上管道符号,可以方便地进行分组概述和分组计算。该函数还可以结合
group_by()函数实现分组聚合,group_by()函数语法:group_by(.data, ..., add = FALSE)
例子:
summarize(group_by(df2tbl,x), sum(y))
四、dplyr两个实用技能
1:管道操作 %>% (cmd/ctrl + shift + M)
(加载任意一个tidyverse包即可用管道符号)
小插曲:tydiverse有哪些包?(来自Lisa的疑问)
- 首先dplyr提供了一个符号%>%,该符号将左边的对象作为第一个参数传递到右边的函数中,这样就实现类似unix管道的编程风格,代码更易读.
- %>%来自dplyr包的管道函数,其作用是将前一步的结果直接传参给下一步的函数,从而省略了中间的赋值步骤,可以大量减少内存中的对象,节省内存.
2:count统计某列的unique值
五、dplyr处理关系数据
- 即:将2个表进行连接,注意:不要引入factor
- R语言环境变量的设置 环境设置函数为
options()
> rm(list = ls()) #一键清空所有变量
> options(stringsAsFactors = F) #对读入数据的string的处理
> test1 <- data.frame(x = c('b','e','f','x'),
+ z = c("A","B","C",'D'),
+ stringsAsFactors = F)
> test1
x z
1 b A
2 e B
3 f C
4 x D
> test2 <- data.frame(x = c('a','b','c','d','e','f'),
+ y = c(1,2,3,4,5,6),
+ stringsAsFactors = F)
> test2
x y
1 a 1
2 b 2
3 c 3
4 d 4
5 e 5
6 f 6
1.內连inner_join,取交集
先普及一下知识:
我们知道,数据库中经常需要将多个表进行连接操作,如左连接、右连接、内连接等,这里dplyr包也提供了数据集的连接操作,具体如下:
left join(左联接) 返回包括左表中的所有记录和右表中联结字段相等的记录
right join(右联接) 返回包括右表中的所有记录和左表中联结字段相等的记录
inner join(等值连接) 只返回两个表中联结字段相等的行
> inner_join(test1, test2, by = "x")
x z y
1 b A 2
2 e B 5
3 f C 6
结果说明:
很明显,这说明inner join并不以谁为基础,它只显示符合条件的记录.
2.左连left_join
> left_join(test1, test2, by = 'x')
x z y
1 b A 2
2 e B 5
3 f C 6
4 x D NA
> left_join(test2, test1, by = 'x')
x y z
1 a 1
2 b 2 A
3 c 3
4 d 4
5 e 5 B
6 f 6 C
结果说明:
left join是以A表的记录为基础的,A可以看成左表,B可以看成右表,left join是以左表为准的.
换句话说,左表(A)的记录将会全部表示出来,而右表(B)只会显示符合搜索条件的记录,B表记录不足的地方均为NULL.
3.全连full_join
> full_join( test1, test2, by = 'x')
x z y
1 b A 2
2 e B 5
3 f C 6
4 x D NA
5 a 1
6 c 3
7 d 4
4.半连接:返回能够与y表匹配的x表所有记录semi_join
> semi_join(x = test1, y = test2, by = 'x')
x z
1 b A
2 e B
3 f C
5.反连接:返回无法与y表匹配的x表的所有记录anti_join
> anti_join(x = test2, y = test1, by = 'x')
x y
1 a 1
2 c 3
3 d 4
6.简单合并
划重点:在相当于base包里的
cbind()函数和rbind()函数;注意,bind_rows()函数需要两个表格列数相同,而bind_cols()函数则需要两个数据框有相同的行数
#数据准备:准备三个test
> test1 <- data.frame(x = c(1,2,3,4), y = c(10,20,30,40))
> test1
x y
1 1 10
2 2 20
3 3 30
4 4 40
> test2 <- data.frame(x = c(5,6), y = c(50,60))
> test2
x y
1 5 50
2 6 60
> test3 <- data.frame(z = c(100,200,300,400))
> test3
z
1 100
2 200
3 300
4 400
#进行合并操作:
> bind_rows(test1, test2) #需要列数相同
x y
1 1 10
2 2 20
3 3 30
4 4 40
5 5 50
6 6 60
> bind_cols(test1, test3) #需要行数相同
x y z
1 1 10 100
2 2 20 200
3 3 30 300
4 4 40 400
★,°:.☆( ̄▽ ̄)/$:.°★ 。今天的学习到此结束,掌握的很多,需要消化的也很多~
参考的网站有:
https://www.cnblogs.com/nxld/p/6060534.html
https://www.cnblogs.com/assasion/p/7768931.html
https://blog.csdn.net/weixin_34233679/article/details/86265275
https://cloud.tencent.com/developer/article/1430436
当然还有公众号:生信星球学习小组的相关内容 ♥
明天再见啦~ヾ(•ω•`)o
——来自猪莎