文章名称:Genome sequencing sheds light on the contribution of structural variants to Brassica oleracea diversification
发表杂志:bioRxiv
发表单位:北京农林科学院等
发表时间:2020 年10月
甘蓝包括几种形态多样、具有重要经济价值的蔬菜作物。我们展示了花椰菜和卷心菜(结球甘蓝)两种形态的甘蓝的高质量染色体规模的基因组组装结果,通过两者比较确定了约120K的高置信度结构变异(SVs)。利用这些SVs对271份甘蓝种质进行群体分析,可以明显区分出不同的形态类型,表明SVs与甘蓝种群分化有关。研究表明,在花椰菜和甘蓝之间受选择SVs影响的基因在胁迫和刺激反应、分生组织及花发育相关功能富集。此外,还鉴定了受选择的SVs影响并参与从营养生长到生殖性生长的转换的基因,这些基因定义了花球的起始、花序分生组织增殖和花球的形成、维持和扩大,为了解花球发育的调控网络奠定了基础。本研究揭示了SVs在甘蓝不同形态类型多样性中的重要作用,新组装的基因组和SVs为今后的研究和育种提供了丰富的资源。
一、研究背景
甘蓝包括几种不同的优势蔬菜作物,2018年全球总产量近1亿吨(http://www.fao.org/faostat).)。该物种的极端多样性是独一无二的,它的形态类型是为了扩大代表收获产品的不同器官而选择的,例如,花椰菜(B. oleracea var. botrytis)和西兰花(B. oleracea var. italica)的花序、结球甘蓝(B. oleracea var. capitata)的头状花序(顶芽)、抱子甘蓝(B. oleracea var. gemmifera)的侧生叶芽、芥蓝(B. oleracea var.alboglabra)的叶子、擘蓝(B.oleracea var. gongylodes)的块茎。在过去的几年中,已经为不同形态的甘蓝属植物生成了参考基因组序列,包括芥蓝、结球甘蓝、花椰菜和西兰花等,这些基因组序列极大地方便了遗传变异分析,从而更好地了解甘蓝的遗传多样性、种群结构以及进化和驯化。
结构变异(SVs),包括插入、缺失、重复和易位,在植物基因组中大量存在,比单核苷酸多态性(SNPs)更有可能引起表型变化。许多SVs已被鉴定为不同作物重要农艺性状的因果遗传变异,例如花椰菜中Or基因第三个外显子中4.7kb插入导致橙色花球;甘蓝型油菜中BnaA9.CYP78A9上游区域的3.7kb插入导致的长角果和大种子;油菜中BnaFLC.A10启动子区域621bp插入有助于油菜对冬季栽培环境的适应。以前对甘蓝属植物的全基因组变异分析主要集中在SNPs和小Indels,而基因组SVs在很大程度上被忽视,这主要是由于在遗传变异鉴定中使用短序列读数的限制。通过将短测序读数映射到参考基因组来进行SV检测容易导致高水平的假阴性和假阳性,特别是对于高度重复的植物基因组,如甘蓝的基因组。因此,到目前为止,甘蓝不同形态类型中SVs的种群动态仍然很大程度上没有被研究。
最近,通过直接比较高质量的染色体水平的基因组组装结果和/或将使用PacBio或Nanopore测序技术产生的长读数映射到参考基因组的方法已被证明对于大型和复杂植物基因组中的SV检测是非常准确的。在这项研究中,我们使用PacBio长读取技术和高通量染色体构象捕获(Hi-C)技术,为花椰菜和结球甘蓝产生了高质量的染色体规模的基因组组装。通过基因组直接比对和长读长比对,在这两个基因组之间共鉴定出119,156个高置信度SVs。我们进一步收集271份(本研究中产生的163份甘蓝种质和先前报道的108份甘蓝种质)不同形态类型甘蓝种质的基因组重测序数据,并利用这些数据对这些种质的119,156个高置信度SVs进行基因分型。对甘蓝不同形态类型的等位基因频率进行研究,主要比较花椰菜和结球甘蓝群体间的等位基因频率,结合基因表达分析,我们证实了SVs对花椰菜花球形成的调控作用。
二、研究方法
1. 基因组
(1)样本情况
花椰菜(B. oleracea var. botrytis)样本Korso_1401是从IPK Gatersleben基因库获得的来自Korso的高度自交系,花球白色致密,成熟时间长(>95d)。
结球甘蓝(B. oleracea var. capitate)样本OX-heart_923,来自江苏省农业科学院蔬菜研究所的绿色尖头晚抽薹自交系。
(2)测序策略
PacBio测序,Korso样本共24个cell,9个PacBio RSII平台,15个PacBio Sequel平台);OX-heart样本15个cell(PacBio Sequel平台)。
Illumina测序,HiSeq 2500平台,PE150测序策略。
Hi-C测序,Illumina HiSeq X Ten平台,PE150测序策略。
BioNano光学图谱,Saphyr平台。
2. 重测序
(1)样本情况
163份甘蓝属不同种质(花椰菜89份,甘蓝65份,花椰菜9份)
(2)测序策略
Illumina HiSeq 2500平台,PE150 测序策略(117份)或PE100测序策略(46份)。
3. 转录组
(1)样本情况
采集Korso样本(根、茎、叶、球、芽、花和角果)和OX-heart样本(根、茎、叶、头状花序、芽、花和角果)7个不同组织,进行了转录组测序。此外,还采集了Korso样本处于以下发展阶段的顶端分生组织(SAM):营养、过渡(花球起始)、花球形成(花球直径~1厘米)、早熟(花球直径10厘米)和枝条伸长(成熟)。每个样本进行两到三个生物学重复。
(2)测序策略
二代转录组,Korso样本和OX-heart样本,Illumina HiSeq X Ten;
三代转录组,Korso样本,PacBio Sequel平台。
三、研究结果
1. 花椰菜和结球甘蓝基因组的从头组装
对花椰菜自交系样本Korso_1401(以下简称Korso)和结球甘蓝样本OX-heart_923(以下简称OX-heart)进行基因组测序。每个样本均产生约70.0 Gb的 PacBio序列,覆盖约120倍的Korso和OX-heart基因组,其估计大小分别为566.9 Mb和587.7 Mb。用PacBio序从头组装成Contigs,使用PacBio长序列和Illumina短序列(每个样本约100 Gb)校正组装的Contigs中的错误。用242.2 GB的BioNano光学图谱数据辅助Korso基因组组装。用Hi-C数据分别对Korso和OX-heart进行辅助组装。最终组装得到的Korso和OX-heart的基因组Contig个数分别为615个和973个,累积长度分别为549.7 Mb和565.4 Mb,N50大小分别为4.97Mb和3.10Mb。结果表明,Korso、OX-heart与西兰花HDEM组装结果的Hi-C热图之间有良好一致性。
对组装质量进行评估,结果发现,约99.8%的Illumina基因组数据可以被映射回组装的基因组上,98.0%的RNA-Seq读数可以被映射回组装的基因组上。BUSCO结果表明,97.2%和96.5%的核心保守植物基因在Korso和OX-heart的基因组中被完整组装。总而言之,这些结果证明了Korso和OX-heart基因组组装的质量较高。
2. 基因组注释与比较基因组
约60.7%的Korso和62.0%的OX-heart被注释为重复元件,其中Gypsy和Copia反转录转座子代表了这两个基因组中最丰富的家族。从Korso和OX-heart和B.rapa (V3.0)基因组中提取全长长末端重复反转录转座子(LTR-RTS),对这些完整LTR-RT的插入时间估计揭开了分别发生在大约20万年和150万年前的Korso和OX-heart两个LTR-RT的爆发。相反,在B. rapa中,大多数LRT-RT是最近形成的,超过30%的完整LTR-RT小于20万年,而在Korso和OX-heart中,这一比例分别为16.3%和15.9%。
高质量的Korso和OX-heart组装结果使我们能够精确识别着丝粒位置。在两个基因组中确定的着丝粒在每条染色体上的位置与之前用荧光原位杂交(FISH)分析确定的着丝粒位置一致。不出所料,重复序列在着丝粒区域得到了丰富。不同的重复元件家族在染色体上表现出明显不同的模式,如Copia类型的LTR主要位于着丝粒上,而Gypsy类型的LTR主要位于着丝粒周围区域(图1A)。
使用结合了从头预测,基于转录本和基于同源性的预测的综合策略,分别从Korso和OX-heart基因组预测了总共60,640和62,232个高可信度蛋白质编码基因。在这些预测基因中,转录组证据支持了70.9%和76.4%,其他植物中有91.0%和90.0%具有同源物。BUSCO显示Korso和OX-heart预测基因的完整性分别为96.1%和94.9%。
对Korso、OX-heart、B. rapa 和A. thaliana基因组的共线性分析证实了芸苔属植物的全基因组三倍化事件(WGT)和随后的亚基因组分化事件。基于这些共线关系,我们确定了Korso和OX-heart基因组中的三倍型区域,并根据其保留的基因密度将它们划分为三个亚基因组(图1)。正如先前研究中报道的一致,Korso和OX-heart的分为三个亚基因组,LF(最不分离的),MF1(中等分离的)和MF2(最分离的),在二倍化过程中显示出相同的重复基因的偏向保留模式。不同亚基因组中的重复基因拷贝显示出不同的基因表达模式,位于LF的拷贝通常比MF1和MF2中的拷贝有更高的表达水平。
我们比较了四个甘蓝种质(花椰菜Korso、结球甘蓝OX-heart、西兰花HDEM和类似甘蓝的快速循环TO1000)、另外三个油菜(B.rapa、B.ngra)和甘蓝C亚组的预测基因的蛋白质序列,以及其他五个十字花科植物(Aethionema arabicum、Arabiopsis thaliana、Capsella Rubella、Thellungiella salsugina和Schrenkiella parual)的预测基因的蛋白质序列。利用1,638个单拷贝同源基因构建了系统发育树,结果表明甘蓝与花椰菜和花椰菜的共同祖先的进化距离约为1.68 百万年,甘蓝与甘蓝C亚组供体的进化距离约为2.27 百万年,而芸薹属(Brassica)与其他十字花科植物的进化距离约为16.18 百万年(图1B),表明甘蓝与花椰菜和花椰菜的共同祖先存在约1.68 百万年的差异,甘蓝与甘蓝C亚组供体的差异约为2.27 百万年,而Brassica与其他十字花科物种的差异约为16.18 百万年(图1B)
a,Korso和OX-heart基因组的特征。i,染色体表意文字。红色,绿色,蓝色和灰色分别表示LF,MF1,MF2亚基因组和着丝粒区域。 ii。 GC含量。 iii,基因密度。 iv,重复密度。 v,Copia型LTR密度。 vi,Gypsy型LTR密度。 vii,Korso和OX-heart基因组之间的共线性分析。 b,基于1638个单拷贝直系同源基因的14种植物/变种的系统发育树及其估计的发散时间(百万年前)。
3. Korso和OX-heart基因组间的SVs
通过利用Korso和OX-heart的高质量基因组组装结果,能够通过直接基因组比较与PacBio长读长结合来鉴定高可信度SVs。 Korso和OX-heart组装结果显示出很高的共线性,这表明它们之间的平衡重排(倒位和易位)并不深刻。因此,在本研究中,我们重点研究了不平衡的SVs,主要是indel。在Korso和OX-heart的基因组之间共鉴定出119,156个SVs,大小在10 bp至667 kb之间,明显偏向相对较短的SVs。
基因和启动子区域中的SVs可以影响相应基因的功能或表达。 SV区分别占Korso和OX-heart总基因组大小的14.5%和15.0%,分别占基因区的10.0%和11.3%,以及编码序列的5.9%和6.6%,提示在基因中,特别是在编码区域中,功能限制了SVs的出现,而在启动子区域中没有发现SV的明显限制。Korso(58.5%)和OX-heart(58.6%)中超过一半的注释基因在其基因或启动子区域中受到至少一种SV的影响,并在多种生物学过程(例如细胞成分组织)中功能丰富,对压力和刺激的反应,信号转导,细胞分化,胚胎发育,基因表达和表观遗传调控以及花和分生组织发育。
我们在油菜中检测到多个先前描述的SVs,包括BoFLC3中两个与亚热带花椰菜适应有关的indel,以及BoFRIa的两个indels与花椰菜和卷心菜的冬季一年生或二年生的习性有关。
4. 不同形态甘蓝中SVs的种群动态
花椰菜和结球甘蓝是甘蓝属植物的两种极端形态类型,识别它们独特表型(如花序和叶状头)形成背后的基因组变异,将为了解这些重要性状的分子调控提供新的见解,并为促进育种提供重要信息。我们在Korso和OX-heart中鉴定的高品质SVs,为研究不同形态分化的甘蓝样本中SVs的动态变化提供了有价值的参考。为此,我们对163份甘蓝进行了基因组重测序,其中包括89份花椰菜、65份卷心菜和9份西兰花。我们还收集了Cheng等人(2016)报道的另外108个甘蓝的重测序数据,包括15个花椰菜,39个卷心菜,24个西兰花,18个大头菜,4个中国甘蓝,2个卷甘蓝,2个羽衣甘蓝,2个球芽甘蓝和2个野生甘蓝的重测序数据。在271份中,211份测序深度在10倍以上。根据基因组测序序列与Korso和OX-heart基因组的比对,在271份样本中对119156份高质量参考SVs进行了基因分型。为了评估我们的SV基因分型的准确性,我们分别对Korso和OX-heart的参考SVs进行了基因分型,方法是将它们的Illumina短读分别映射到这两个基因组上。超过86%的SVs可以进行基因分型,只有0.1%的SVs进行了错误基因分型,表明我们的基因分型具有较高的敏感性和准确性。每个品系的SVs基因分型率在41.3%至80.2%之间,其中187份(69.0%)和254份(93.7%)品系的基因分型率分别大于70%和60%。总的来说,在271份材料中,有89,882的SVs (75.4%)成功地进行了基因分型。
甘蓝型油菜不同组之间的SV等位基因频率变化主要是由于不同的理想性状驯化和适应不同环境的结果。不出所料,带有纯合子Korso等位基因的SV基因座在花椰菜种中普遍存在,平均占每个样本的82.3%,而在卷心菜中,纯合的OX-heart等位基因则普遍存在,平均频率为61.7%(图2a)。使用SV的系统发育和主成分分析(PCA)分析将花椰菜,卷心菜,西兰花和大头菜的种质清楚地分为不同的组(图2b和2c),这与我们基于相同271种种质的SNP数据揭示的模式一致,并且之前的报告基于119个种质一致(Cheng等,2016),进一步支持了我们的SV检测和基因分型非常可靠。
为了确定可能与花椰菜或甘蓝的特定性状相关的SVs,我们提取了总共49,904个SVs,它们在花椰菜和甘蓝的种群之间具有等位基因频率显着不同的频率(图3a)。在这些SV中,49,285(98.8%)基因型的等位基因频率花椰菜显著高于卷心菜,只有550个基因型的等位基因频率卷心菜高于花椰菜。这些可能选择的SVs分布在整个染色体上而没有明显的热点。受选择的SV在整个基因组中的普遍存在与两个高度专业化的油菜形态型之间相对较大的发散时间(〜1.68 百万年)以及它们的独立进化和驯化历史相符(图1b)。
在Korso和OX-heart基因组中,分别有21,111和21,400个基因在其基因或启动子区域中与至少一个受选择的SV重叠,在CDS区域中有6,059和6,344个与受选择的SV重叠。使用GO富集分析显示受选择的SV相关的基因在信号转导,刺激反应,细胞分化,细胞周期,胚胎发育,细胞生长和细胞死亡以及花发育有关的通路富集(图3b),显示出花椰菜和卷心菜不同表型的潜在关联。
a,具有不同基因型的SVs在不同形态型的种质中的百分比。 b,基于SVs的甘蓝种质的主成分分析。 c,使用SVs对271个甘蓝种的最大似然树和基于模型的聚类。树的分支颜色表示与b中不同的形态颜色一致。 K,祖宗血统的数量。
a,散点图,显示卷心菜和花椰菜中SV等位基因频率。b,与SV重叠的在甘蓝和花椰菜组之间具有明显的等位基因频率差异的基因GO富集分析。
5. 受选择的SVs为花椰菜花球形成的进化提供了洞察力
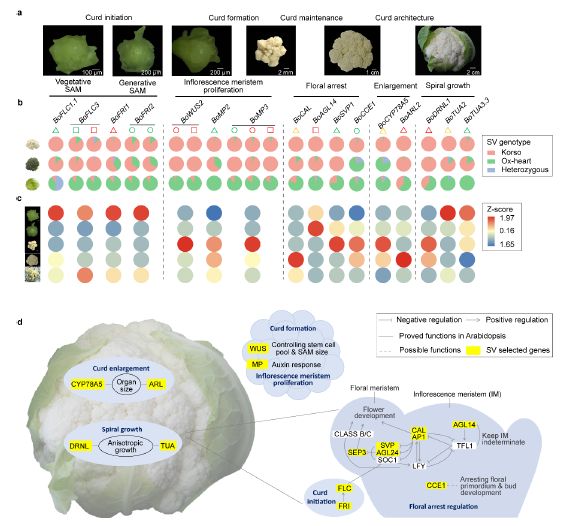
花椰菜的花球由螺旋叠代模式的初生花序分生组织组成,在这里,我们检索到总共294个基因,这些基因在其启动子或基因区域中带有受选择的SV,并且据报道,其拟南芥中的同源物在开花时间和花卉发育,分生组织维持和测定,器官大小控制以及芽或花序结构中起作用。此外,从营养枝顶端分生组织(SAM)到扩大的花球的五个阶段进行了RNA-Seq分析,以揭示SV在花球形成和发育中的潜在作用。
6. 从营养发育向生殖发育的转变
花球起始的第一阶段与从营养发育到生殖发育的转变相对应(图4a)。花椰菜及时地过渡到花蕾的形成阶段对于花球的形成是必不可少的,而对于卷心菜,需要较长的营养阶段才能使叶头正常发育。 MADS盒转录因子FLC是春化和自主途径中的开花时间整合子,起着抑制开花的作用。多项研究证明了FLC旁系同源物在不同的油菜形态型中的开花时间中的作用。在Korso的BoFLC1.1启动子中发现了3,371 bp的插入片段(SV_b_92666a),该序列在强烈的差异选择下出现,分别存在于99%和88%的花椰菜和西兰花中,而仅在9%的卷心菜中(图4b)。BoFLC1.1及其两个串联旁系同源物(BoFLC1.2和BoFLC1.3)以及BoFLC3在过渡阶段均被显着下调(图4c)。Korso的等位基因BoFLC3基因在第一个内含子中包含263 bp缺失(SV_w_24534)和49 bp插入(SV_w_24533)。在拟南芥和十字花科作物中已经报道了FLC第一内含子的结构对开花时间的影响。我们发现在这两个SV基因座上,Korso等位基因在花椰菜(86.7%和86.4%)和西兰花(96.9%和92.9%)中占优势,而在卷心菜(9.7%和8.7%)中则很少见(图4b)。
FLC功能由FRI激活,已被确定为花椰菜花球诱导的温度依赖性时机QTL区域中的候选基因。在Korso和OX-heart基因组中都鉴定出两个FRI同源物BoFRI1和BoFRI2。BoFRI1启动子区域的743 bp缺失(SV_b_96002)是Korso等位基因的特征。大部分花椰菜(98.0%)具有纯合子Korso基因型,而大多数卷心菜(87.0%)具有纯合子OX-heart基因型(图4b)。对于BoFRI2,在其编码区中鉴定出两个插入片段(12和21 bp,SV_w_31837和SV_w_31838),都显示出花椰菜和卷心菜的基因型频率有显着差异(图4b)。已经发现这两个插入缺失与冬季一年或两年一次的花椰菜和卷心菜习性有关。FES和SUF可与FRI形成推定的转录激活物复合物,以促进FLC表达。与卷心菜相比,BoFES1.1和BoSUF4.2在花椰菜中具有受选择的SV,其表达在过渡期显着下调,与BoFLC1s和BoFLC3相似。参与调控FLC表达的其他基因,包括参与表观遗传修饰的基因,例如PRC1和PRC2复杂成分BoVIN3,BoVIL2.3,BoVRN1.1,也包含受选择的SV。这些结果共同表明,FLC相关的自主途径和春化途径可能受到花椰菜和卷心菜之间差异SV的影响,从而导致它们转换到生殖阶段的时机不同。
a,顶端分生组织(SAM)和花球在花球发展的不同阶段的示例图。b,花椰菜,西兰花和卷心菜中SV与候选基因重叠的等位基因频率。三角形,圆形和正方形分别表示启动子,CDS和内含子区域中的SV,它们的不同颜色(绿色,红色和黄色)分别表示Korso中与OX-heart相比不同类型的SV:插入,缺失和替换。c,显示在花球发育不同阶段的候选基因表达的热图。d,花球形成和发育的调控网络。黄色背景的基因为花椰菜和卷心菜之间受选择的SV基因。
7. 花序分生组织增殖
花球起始后的主要过程是形成花球的未确定的花序分生组织的连续规则螺旋增殖。干细胞维持和分生组织扩散在这一过程中起着关键作用。WUSCHEL充当生长素反应变阻器,以维持拟南芥中的顶端干细胞。在Korso的BoWUS2的第一个内含子中鉴定出12 bp的读框内缺失(SV_w_83072)和21 bp的插入(SV_w_83073)。所有采样的花椰菜和花椰菜种质均具有两个SV的纯合子Korso基因型,而甘蓝中的Korso等位基因很少(4%)(图4b)。BoWUS2的表达从营养形成到花球形成显着上调,在花球形成阶段的表达最高(图4c),这意味着这两个SV可以在花球形成中发挥作用。
MP / ARF5与ANT和AIL一起在依赖植物生长素的器官启动和叶序模式中发挥关键作用。我们鉴定了在BoMP2,BoMP3,BoANT,BoAIL5,BoAIL6和BoAIL7的启动子和其同源基因的基因区域中选择的SV。花椰菜BoMP2的启动子中23 bp的插入(SV_w_71238)在花椰菜中处于强选择状态(花椰菜和卷心菜种分别为96.1%和0%)。花椰菜中BoMP3基因CDS区中11 bp插入和23 bp缺失(SV_w_92482和SV_w_92481)和内含子区14 bp缺失(SV_w_92433)受到强烈选择(花椰菜分别为95.6%,96.1%,96.2%和卷心菜分别为12%,14%,13.3%(图4b)。与BoWUS2一样,在花球形成阶段也观察到了BoMP2和BoMP3的最高表达(图4c)。
8. 花序维持和花停滞
花椰菜的花球由成千上万的花序分生组织组成,花序分生组织在发育中被阻滞。在分生组织决定(FMI)基因BoCAL的启动子区域鉴定出大的变异(SV_b_70950)(在OX-heart中约11.4 kb,在Korso中约7.7 kb),受到强烈的选择。几乎所有花椰菜(99.0%)和大多数西兰花(87.5%)品种都具有Korso等位基因,而大多数卷心菜品种(79.2%)在此基因座上都具有OX-heart等位基因(图4b),表明其花球形成中的潜在作用。
其他一些FMI基因,包括BoAP1.2,BoFUL1,BoFUL3和BoSEP3,也受到受选择的SVs的影响,并且在营养,过渡和花球形成阶段均表达相对较低,但在花球膨大时表达显着提高。拟南芥的研究表明,花分生组织决定(IMI)基因TFL1和FMI基因之间的拮抗相互作用调节了发育命运的转变。与FMI基因相比,BoTFL1.2的表达模式几乎相反,表明其抑制作用。虽然在BoTFL1.2中未鉴定出受选择的SV,但在强选择下发现其正调控子BoAGL14的内含子有13 bp缺失(SV_w_84836)(图4b),而BoAGL14表现出与BoTFL1.2相同的表达模式,表明BoTFL1.2和BoAGL14在阻止花序形成和维持,从而形成花球和维持花球中潜在重要的作用。SVP是花卉过渡的关键负调控因子。在Korso、98.1%的其他花椰菜和所有西兰花种质中发现了BoSVP1启动子中的420 bp(SV_w_74120)插入,而在卷心菜种质中仅25.2%(图4b)。BoSVP1从营养期到过渡期显着上调,并在整个花球形成过程中保持高表达水平(图4c),表明其在花芽发育中的阻遏作用,如拟南芥中报道。
据报道,花椰菜花球特异性基因BoCCE1在分生组织发育/停止过程中具有潜在的作用。在这里,我们确定了一个1,505 bp的插入片段(SV_b_67089a),覆盖了整个Korso中的BoCCE1基因体。该插入片段的基因分型显示,BoCCE1基因存在于大多数花椰菜种质中(97.1%),但在大多数卷心菜(86.5%)和西兰花(78.1%)种质中均不存在,这表明BoCCE1在花椰菜芽中可能具有花停滞作用。与花椰菜芽相比,它们在发育后期被阻滞(图4b,c)。
9. 花序膨大和螺旋状生长
和器官大小调节、细胞分裂和扩增、胞周期等相关基因可以调节花球的重量。CYP78A5(KLU)已在拟南芥中鉴定,可防止增殖停滞并促进器官生长。花椰菜BoCYP78A5的高表达仅在花球中检测到,尤其是在花球形成和膨大阶段(图4c)。BoCYP78A5启动子中的2775-bp取代(SV_b_76292)存在于98.1%的花椰菜种中,而仅存在于8.2%的卷心菜种中(图4b),可能有助于BoCYP78A5的花球特异性表达。BoARL2(或CDAG1)已被证明在促进花椰菜花球大小中起作用。BoARL2在花球中高度表达,在Korso的花球扩大阶段表达最高(图4c)。在BoARL2的启动子中检测到一个269 bp的缺失(SV_w_38468),并存在于所有花椰菜和大多数西兰花(96.9%)的种中,而只有41.7%的卷心菜种(图4b)。
花序的螺旋排列是花椰菜花球的典型特征。DRNL基因的转录标志着花序分生组织外围区域的侧方器官开始分化。在BoDRNL1的启动子中发现了258 bp的缺失(SV_w_30645),该启动子存在于所有花椰菜和西兰花品种中,而仅在卷心菜品种中占40.6%(图4b)。BoDRNL1特别在花球中表达,在花球形成和膨大阶段表达最高(图4c),这表明其在决定花球结构中的潜在作用。在alpha-微管蛋白基因BoTUA2和四个BoTUA3基因中也鉴定出了受选择的SVs(图4b),其在在拟南芥中的同源基因导致螺旋生长。
四、总结
油菜B. oleracea物种包括许多重要的蔬菜作物,它们表现出异常高的形态多样性,其中花椰菜和卷心菜代表两种极端形态。在这项研究中,我们通过整合PacBio的长读长和Hi-C数据,为花椰菜和结球甘蓝组装了高质量的染色体级基因组序列,这为油菜作物的未来研究和改良提供了重要资源,为全面探索油菜表型多样性提供基础。
SVs在植物表型变化的遗传调控中起着至关重要的作用,并且通常是许多重要性状的致病性遗传变异,这些重要性状是作物驯化和育种的目标。但是,农作物中SVs的种群分析远远落后于SNP的种群,这主要是由于准确识别SVs的技术困难。当前广泛使用的SVs检测方法依赖于短测序reads和参考基因组的比对,这容易导致高假阳性率和高假阴性率。PacBio和Nanopore等长读长测序技术的最新进展有助于SVs的检测。但是,由于读取长度受限制,因此无法检测到一些较大的SV(例如插入)。在本研究中,通过直接比较花椰菜和卷心菜的高质量参考基因组,结合长读比对,识别出约120K个高置信度SV,其中许多大于100 kb。在包含271个代表不同B. oleracea形态的种质的群体中,此SVs参考集的基因分型以及对这些SVs在不同形态类型群体中(主要是花椰菜,西兰花和卷心菜)的等位基因频率差异的研究揭示了许多在某些形态型下处于选择状态的SVs,其中许多影响基因与其相应的独特表型的行成。
花椰菜的花球由成千上万的花序分生组织组成,这些分生组织螺旋状排列在较短的扩大的花序分支上。这使得花椰菜成为分析花序发育和极端器官发生的遗传机制的理想模型。在花椰菜中受选择的SVs影响许多基因。结合对花球发育过程中表达谱的分析,我们鉴定了数十种关键的SVs和相关基因,它们与花椰菜的独特花球表型具有潜在的关联。这些包括在花球发育的不同发育阶段中起作用的基因。第一阶段是花球起始,涉及从营养阶段到生殖阶段的转变,影响开花时间调节的基因(例如FLC和FRI)受到影响。花球形成的关键步骤是花序增殖,其中WUS和MP等基因具有花椰菜和西兰花特定的SVs。花椰菜花球的特征还在于花分生组织的停滞,与几个花序决定基因(例如CAL,AP1和SEP3)以及受选择的SVs影响的潜在负调控基因(例如AGL14,SVP和CCE1)相匹配。在花椰菜花球结构中起作用的几个基因也受到所选SVs的影响,这些包括在器官大小控制(例如CYP78A5和ARL)和花序螺旋组织(例如DRNL和TUA)中可能发挥作用的基因(图4d)。我们的分析表明,SVs对花椰菜独特的花球表型有重要贡献,并阐明了花椰菜花球形成的调控网络。
参考文献
1. Ning Guo, Shenyun Wang, LeiGao,et al. Genome sequencing sheds light on the contribution of structuralvariants toBrassica oleraceadiversification.bioRxiv,2020.