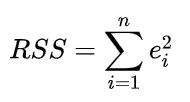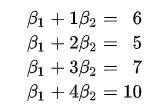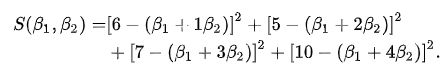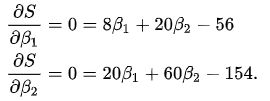线性回归与最小二乘法
欢迎关注”生信修炼手册”!
线性回归模型是使用最广泛的模型之一,也最经典的回归模型,如下所示
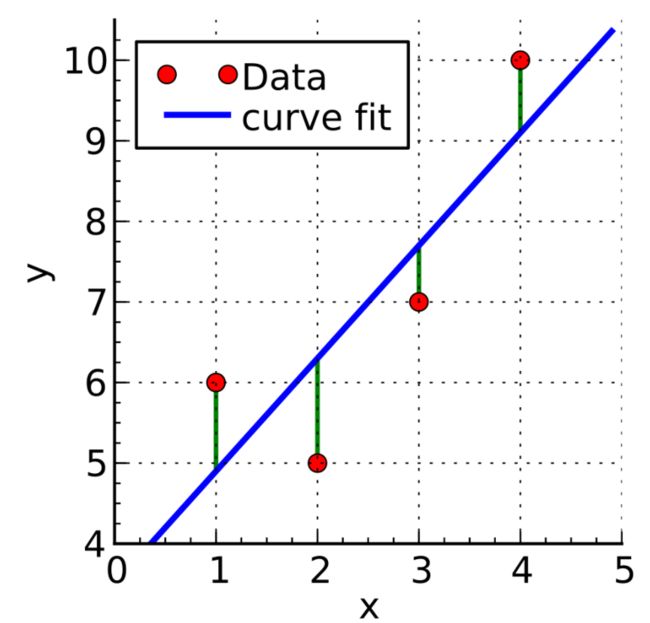
x轴表示自变量x的值,y轴表示因变量y的值,图中的蓝色线条就代表它们之间的回归模型,在该模型中,因为只有1个自变量x,所以称之为一元线性回归,公式如下
我们的目的是求解出具体的参数值,可以穿过这些点的直线可以有多条,如何选取呢?此时就需要引入一个评价标准。在最小二乘法中,这个评价标准就会误差平方和,定义如下
其中e表示通过回归方程计算出的拟合值与实际观测值的差,通过维基百科上的例子来看下实际的计算过程
如上图所示,有4个红色的采样点,在每个点都可以得到(x, y)的观测值,将4个采样点的数据,带入回归方程,可以得到如下结果
计算全部点的误差平方和,结果如下
对于上述函数,包含了两个自变量,为了求解其最小值,可以借助偏导数来实现。通过偏导数和函数极值的关系可以知道,在函数的最小值处,偏导数肯定为0,所以可以推导出如下公式
对于上述两个方程构成的方程组,简单利用消元法或者代数法就可以快速求出两个参数的值
实际上,更加通过的方法是通过矩阵运算来求解,这种方法不仅适合一元线性回归,也适合多元线性回归,其本质是利用矩阵来求解以下方程组
计算过程如下
>>> data = np.array([[1, 1], [1, 2], [1, 3], [1, 4]])
>>> data
array([[1, 1],
[1, 2],
[1, 3],
[1, 4]])
>>> target = np.array([6, 5, 7, 10]).reshape(-1, 1)
>>> target
array([[ 6],
[ 5],
[ 7],
[10]])
# 先对data矩阵求逆矩阵
# 再计算两个矩阵的乘积
>>> np.matmul(np.matrix(data).I, target)
matrix([[3.5],
[1.4]])通过一个逆矩阵与矩阵乘积操作,就可以方便的求解参数。在scikit-learn中,使用最小二乘法的代码如下
>>> data = np.array([1, 2, 3, 4]).reshape(-1, 1)
>>> data
array([[1],
[2],
[3],
[4]])
>>> target = np.array([6, 5, 7, 10]).reshape(-1, 1)
>>> target
array([[ 6],
[ 5],
[ 7],
[10]])
>>> reg = linear_model.LinearRegression()
>>> reg.fit(data, target)
LinearRegression()
>>> reg.intercept_
array([3.5])
>>> reg.coef_
array([[1.4]])intercept_表示回归方程的截距,coef_表示回归方程的系数。
最小二乘法的求解过程简单粗暴,但是也存在一定限制,首先,根据方程组能够求解可以知道,样本数目必须大于等于特征的个数;其次,当输入的特征很多,大于10000时,矩阵运算非常的费时。
最小二乘法肯定可以求解出线性方程的解,但是其解只是在线性模型假设的前提下得到的最优解,如果数据不符合线性模型,此时用最小二乘法依然可以得到结果,但是显然是一个非常差的拟合结果,为了更好的评估线性回归拟合效果的好坏,我们还需要一个评估指标R square, 公式如下
这个值也称之为拟合优度,从定义可以看出,其范围在0到1之间,越靠近1,说明拟合效果越好。在scikit-learn中,提供了计算拟合优度的函数,用法如下
>>> from sklearn.metrics import mean_squared_error, r2_score
>>> predict = reg.predict(data)
>>> mean_squared_error(target, predict)
1.0500000000000003
>>> r2_score(target, predict)
0.7对于线性回归而言,离群值对拟合结果影响很大,在预处理阶段,要注意过滤离群值点;同时,我们会根据回归系数对变量的重要性进行排序,此时要注意各个变量的单位是不一样的,在预处理阶段需要进行归一化。
·end·
—如果喜欢,快分享给你的朋友们吧—
原创不易,欢迎收藏,点赞,转发!生信知识浩瀚如海,在生信学习的道路上,让我们一起并肩作战!
本公众号深耕耘生信领域多年,具有丰富的数据分析经验,致力于提供真正有价值的数据分析服务,擅长个性化分析,欢迎有需要的老师和同学前来咨询。
更多精彩
KEGG数据库,除了pathway你还知道哪些
全网最完整的circos中文教程
DNA甲基化数据分析专题
突变检测数据分析专题
mRNA数据分析专题
lncRNA数据分析专题
circRNA数据分析专题
miRNA数据分析专题
单细胞转录组数据分析专题
chip_seq数据分析专题
Hi-C数据分析专题
HLA数据分析专题
TCGA肿瘤数据分析专题
基因组组装数据分析专题
CNV数据分析专题
GWAS数据分析专题
2018年推文合集
2019年推文合集
写在最后
转发本文至朋友圈,后台私信截图即可加入生信交流群,和小伙伴一起学习交流。
扫描下方二维码,关注我们,解锁更多精彩内容!
![]()
一个只分享干货的
生信公众号