论文解读:《基于知识的BERT:一种像计算化学家一样提取分子特征的方法》
论文解读:《Knowledge-based BERT: a method to extract molecular features like computational chemist》
- 1.文章概述
- 2.背景
- 3.数据
-
- 3.1 数据预处理
- 3.2 用于分子特性预测的小型药物数据集
- 3.3 Malaria (疟疾)数据集
- 3.4 CHIRAL1 数据集
- 4.方法
-
- 4.1 预训练任务
- 4.2 模型构建
-
- 4.2.1 K-BERT的预训练
- 4.2.2 K-BERT 的微调
- 4.2.3 数据增强
- 4.3 模型评估
- 5.结果
-
- 5.1 K-BERT 的性能比较
- 5.2 预训练可以显着增强模型提取分子特征的能力
- 5.3 对比学习预训练任务使 K-BERT 能够更好地“理解”SMILES
- 5.4 预训练使 K-BERT 能够生成通用分子指纹
- 5.5 K-BERT-FP 可以捕获 MACCS 无法捕获的分子大小特征
- 5.6 K-BERT-FP 可以通过进一步的预训练来捕捉手性特征
- 6.结论
- 7.关键点
文章地址:https://academic.oup.com/bib/article-abstract/23/3/bbac131/6570013?redirectedFrom=fulltext&login=false
DOI:https://doi.org/10.1093/bib/bbac131
期刊:BRIEFINGS IN BIOINFORMATICS
2021年影响因子/JCR分区:13.994/Q1
出版日期:2022年4月18日
代码:https://github.com/wzxxxx/Knowledge-based-BERT
1.文章概述
基于机器学习算法的分子性质预测模型已成为在药物发现早期阶段对没有前途的先导分子进行分类的重要工具。与主流的基于描述符和图的分子性质预测方法相比,基于 SMILES 的方法无需人类专家知识即可直接从 SMILES 中提取分子特征,但它们需要强大的特征提取算法和更大的训练数据量,这使得基于 SMILES 的方法不太受欢迎。作者展示了预训练在促进重要药物特性预测方面的巨大潜力。分别利用基于原子特征预测、分子特征预测和对比学习预训练任务,开发了一种新的预训练方法K-BERT,可以像化学家一样从SMILES中提取化学信息。 在15 个药物数据集上进行了试验,计算结果表明,K-BERT 优于成熟的基于描述符(XGBoost)和基于图(Attentive FP 和 HRGCN+)的模型。此外,作者还发现对比学习预训练任务使 K-BERT 能够更好地“理解” SMILES,而不仅限于使 SMILES规范化。此外,K-BERT 生成的通用指纹 K-BERT-FP 在 15 个药物数据集上表现出与 MACCS 相当的预测能力,还可以捕获传统二进制指纹无法捕获的分子大小和手性信息。最后的结果说明了 K-BERT 在药物发现中,对于分子特性预测的实际应用有着巨大潜力。
2.背景
药物开发是一个漫长、复杂且成本高昂的过程,平均而言,将一种新药推向市场需要 10 多年和近 26 亿美元的时间。由于具有环保、高效和低成本的优势,基于机器学习(ML)算法的分子性质预测模型已成为在药物发现的早期阶段对没有前途的候选药物进行分类的重要工具。
在分子表示方面,主流的基于ML的分子性质预测方法大致可以分为三类:基于描述符的、基于图的和基于SMILES的。
- 基于描述符的方法通过预定义的分子描述符/指纹的固定长度特征向量来表征分子,一直是分子性质预测中最流行的方法。然而,基于描述符的模型质量对用于训练的专家设计的分子特征非常敏感,并且描述符/指纹的生成需要广泛的人类专家知识。
- 基于图的方法将分子表征为具有节点(原子)和边(键)的分子图,而不是固定长度的特征向量,它们可以从原子和键属性定义的简单初始特征中自动提取分子特征以及通过图神经网络(GNN)框架的分子拓扑结构。
因此,基于图的模型可以针对特定任务自动学习分子中每个原子的表示,这在一定程度上避免了手动提取描述符/指纹时任务相关信息的丢失。然而,基于图的方法严重依赖数据量,当数据集较小时,其性能甚至不如基于描述符的方法。此外,GNN 容易出现过度平滑的问题,因此 GNN 的层数一般只有 2-4 层,这限制了其特征提取能力。因此,基于图的方法在某些任务上取得了优异的性能,但并未在药物发现上带来突破性进展。 - SMILES 是一种用于表示分子的简化化学语言。因此,基于 SMILES 的分子性质预测可以看作是一个自然语言处理(NLP)问题。基于 SMILES 的方法无需人类专家知识即可直接从 SMILES 中提取分子特征,而无需依赖任何手动生成的特征。由于分子的结构和化学信息隐含在 SMILES 中,不像分子描述符/指纹和分子图那样明确,因此基于 SMILES 的方法对特征提取能力和数据量有更高的要求。这使得基于 SMILES 的方法并不如前两者流行。
在这项研究中,作者基于 BERT 构建了基于 SMILES 的预训练模型,并探索了基于 SMILES 的预训练模型可以为药物发现中重要药物特性的预测带来什么。为了使模型能够“理解”化学,并学习从分子中提取重要特征,将基于描述符和基于图形的方法中手动生成的信息设置为预训练任务。预训练模型被命名为基于知识的 BERT (K-BERT)。 K-BERT 的第一个预训练任务是从分子图中预测原子的初始特征,以便模型能够识别原子之间更细微的差异(词汇表),而不是简单地识别原子类别。第二个预训练任务是从分子描述符/指纹中预测分子的分子特征,使模型能够学习从分子中提取预定义的分子特征。第三个预训练任务是对比学习,使同一分子的不同 SMILES 的嵌入更加相似,从而使 K-BERT 能够“理解” SMILES。使用 15 个药物数据集来评估 K-BERT 的性能,结果表明 K-BERT 优于基于描述符的方法和基于图的方法 。结果表明,预训练可以显着增强模型提取分子特征的能力,并使 K-BERT 能够生成通用指纹。生成的指纹 K-BERT-FP 在 15 个数据集上取得了与 MACCS 指纹相当的性能,并且可以捕获 MACCS 无法捕获的分子的大小和手性特征。
3.数据
3.1 数据预处理
CHEMBL 中的小分子用于 K-BERT 的预训练。所有的分子都被规范化了,混合物、无机物和RDKit不能处理的分子都被去除了。最后,将近 180 万个分子用于预训练。
3.2 用于分子特性预测的小型药物数据集
作者测试了K-BERT在15个成药性小数据集上的表现,数据集分子数都在2000以下。具体数据集如下:Pgp-substrate (Pgp-sub)、human intestinal absorption (HIA)、 human oral bioavailability 20% (F20%)、human oral bioavailability 30% (F30%)、CYPsubstrate (CYP1A2-sub、CYP2C19-sub、CYP2C9-sub、CYP2D6-sub和CYP3A4-sub)、half-life (T1/2)、 drug-induced liver injury (DILI)、FDA maximum recommended daily dose (FDAMDD)、skin sensitization (SkinSen)、carcinogenicity (Carcinogenicity)和respiratory toxicity (Respiratory)。
3.3 Malaria (疟疾)数据集
Malaria 数据集是搜集自Malaria Treatment Response Portal的一个子集,里面的分子都包含手性信息。针对恶性疟原虫的生物活性类别(1 表示活跃,0 表示不活跃),用于 100 000 种化合物的多样化集合,其 3D 地形特征源自天然产物中常见的立体化学和骨骼元素,但在典型筛选集合中的代表性不足。这个数据集被用于评估K-BERT是否能够学习到手性信息。
3.4 CHIRAL1 数据集
CHIRAL1数据集是 Lyu 等人报道的多巴胺受体D4对接筛选数据的一个子集。CHIRAL1中的每个分子只有一个四面体中心,根据中心的手性分为R和S。在本研究中,共有204778个分子用于进一步的预训练,使得K-BERT能学习到手性信息。
4.方法
4.1 预训练任务
作者在BERT基础上提出了新的预训练策略,让模型能够直接从SMILES中提取分子特征。作者提出了三个预训练任务:原子特征预测任务、分子特征预测任务和对比学习任务。原子特征预测任务允许模型学习基于图的方法中手动提取的信息(初始原子信息),分子特征预测任务允许模型学习基于描述符的方法中手动提取的信息(分子描述符/指纹),而对比学习任务允许模型使同一分子的不同 SMILES 字符串的embedding更相似,从而使 K-BERT 能够识别同一分子的不同SMILES字符串。
(1)预训练任务1:原子特征预测任务
对RDKit计算所得的分子中每个重原子的原子特征进行预测。原子特征将包括度、芳香性、氢、手性和手性类型等,因此可看作是一个多任务分类任务。
(2)预训练任务2:分子特征预测任务
对RDKit计算所得的分子特征进行预测。本研究采用MACCS指纹,同样也可将该任务视为多任务分类任务(可以更换为其他的指纹/描述符)。
(3)预训练任务3:对比学习
对于canonical SMILES输入,通过SMILES随机化得到多种不同的SMILES形式。该预训练任务的目标是最大化同一分子不同SMILES字符串嵌入的余弦相似度,最小化不同分子间嵌入的相似度,使得模型能够更好地“理解”SMILES。
4.2 模型构建
4.2.1 K-BERT的预训练
输入特征:如图 1 所示,每个SMILES都是用Schwaller等人提出的标记化方式来标记成不同的token。然后将token(如’O’、‘Br’和‘[C@H]’)编码成K-BERT的输入。
预训练:每个重原子的原子特征和每个分子的分子特征都通过RDKit计算,并分别用于预训练任务1和2。使用RDKit 计算 CHEMBL 中每个分子的一个规范(典型)的SMILES和4个随机生成的 SMILES,用于预训练任务3。CHEMBL中大约 180 万个分子被用于预训练K-BERT,目标为最小化3个预训练任务的损失函数。
4.2.2 K-BERT 的微调
如图1D所示,K-BERT中有6个transformer encoder,作者从预训练模型中载入前5个transformer encoder的参数,第6层transformer encoder和预测层重新随机初始化。然后在下游任务数据上,重新训练模型。
4.2.3 数据增强
每个分子的SMILES,通过RDKit随机扩充到5个不同的SMILES。在训练集中,每个SMILES都被当做是单独(不同的)分子。而在测试集和验证集中,同一分子的不同SMLES都被看成是该分子,5个不同SMILES的预测结果均值作为该分子的预测结果。
4.3 模型评估
每个数据集通过随机拆分以 8:1:1 的比例分为训练集、验证集和测试集。验证集用于模型部分,测试集用于验证 K-BERT 的性能。
回归任务通过确定系数 (R2)、平均绝对误差 (MAE) 和均方根误差 (RMSE) 进行评估。分类任务通过受试者工作特征曲线下面积(ROC-AUC)和马修斯相关系数(MCC)进行评估。
5.结果
5.1 K-BERT 的性能比较
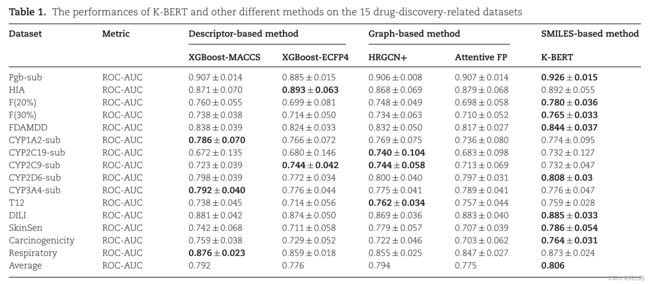
作者首先评估了K-BERT在15个成药性数据集上的表现。在原子特征预测任务、分子特征预测任务和对比学习任务上对 K-BERT 进行了预训练,得到了相应的预训练模型。然后,通过微调将预训练的模型转移到 15 个下游任务中。使用两种基于图的方法 HRGCN+ 和 Attentive FP 、两种基于描述符的方法XGBoost-MACCS和XGBoostECFP4进行比较。K-BERT取得了优异的表现(表1),在8个数据集中取得了最好的结果。
5.2 预训练可以显着增强模型提取分子特征的能力
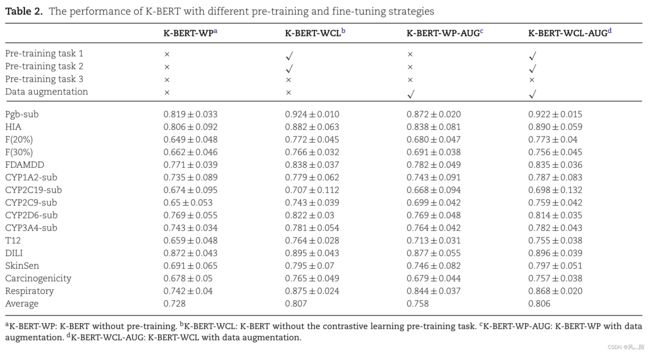
数据增强在基于SMILES的模型中表现出了极大的优势。本文发现,数据增强和预训练都是增强模型从SMILES中提取分子特征的能力。如表2所示,作者采用了不同的策略训练模型。由于这里会比较数据增强,而对比学习在预训练过程中有类似数据增强的操作,为了公平,表2中均未采用对比学习预训练任务。K-BERT-WCL显著优于K-BERT-WP,说明预训练能够提升模型提取分子特征的能力。同时,K-BERT-WP-AUG表现优于K-BERT-WP也能够说明,数据增强也能够帮助模型更好的理解SMILES进而提升模型性能。K-BERT-WCL和K-BERT-WCL-AUG表现差不多,这说明数据增强对已经过预训练的模型提升帮助非常有限。这也符合预期,通过预训练,模型已经能够较好的理解SMILES规则。这时候用同一分子的不同SMILES进行数据增强,相当于在对同一分子进行多次训练,自然难以提升模型性能。
5.3 对比学习预训练任务使 K-BERT 能够更好地“理解”SMILES
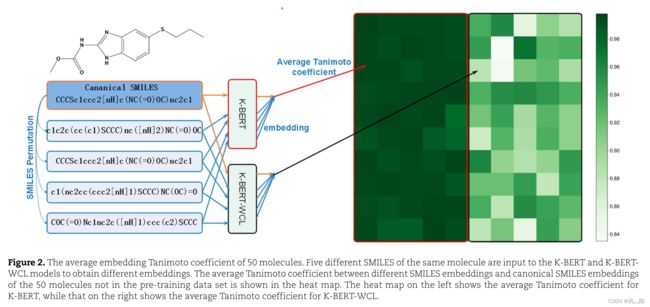
通过相同分子的不同 SMILES 计算了从 K-BERT-WCL 和 K-BERT 生成的嵌入的相似性;使用未出现在预训练数据集中的 50 个分子来评估同一分子的不同 SMILES 的嵌入相似性。对于每个分子,使用 RDKit 根据规范 SMILES 生成其他 4 个不同的 SMILES,然后使用 K-BERT -WCL 和 K-BERT 生成相应的embedding。 作者比较了同一分子不同SMILES通过模型生成的embedding的平均Tanimoto相似度。结果如图2所示,经过对比学习预训练任务,embedding的相似度得到了显著提升。这说明,对比学习能够帮助模型识别同一分子的不同SMILES字符串。
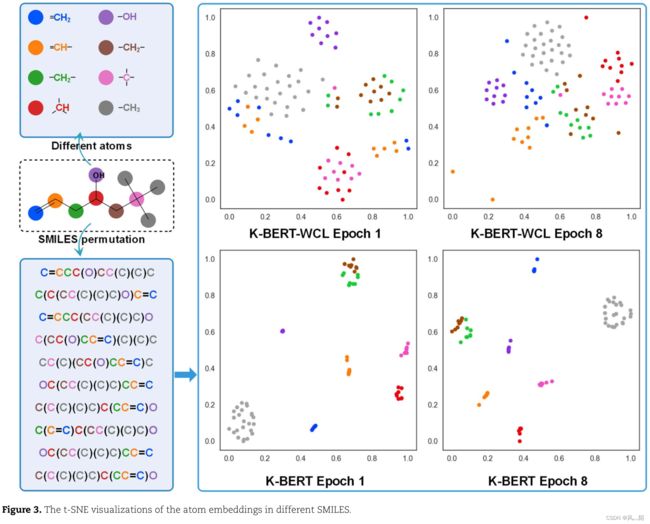
此外,作者以分子‘C=CCC(O)CC©©C’(不在预训练数据集中)为例,通过RDkit随机生成十个SMILES字符串,并对分子中的不同原子embedding进行了t-SNE可视化,结果如图3所示。结果显示,经过对比学习预训练任务,K-BERT 在区分不同 SMILES 中的相同原子标记方面优于 K-BERT-WCL,进一步证明对比学习预训练任务使 KBERT 能够更好地“理解”SMILES。在对比学习预训练任务的约束下,K-BERT 模型总能很好地区分不同 SMILES 中的相同原子标记(在 epoch 1 和 8 中),但 K-BERT WCL 不能。 K-BERT-WCL 在 epoch 8 的表现优于 epoch 1,说明随着预训练的推进,KBERT-WCL 模型可以逐渐理解同一分子的不同 SMILES。
5.4 预训练使 K-BERT 能够生成通用分子指纹
K-BERT生成的分子embedding能够作为一种通用的分子指纹K-BERT-FP(不局限于某一任务)。作者比较了K-BERT-FP和MACCS在成药性数据集上的表现(图4),结果显示K-BERT-FP和MACCS取得了可比较的预测能力。
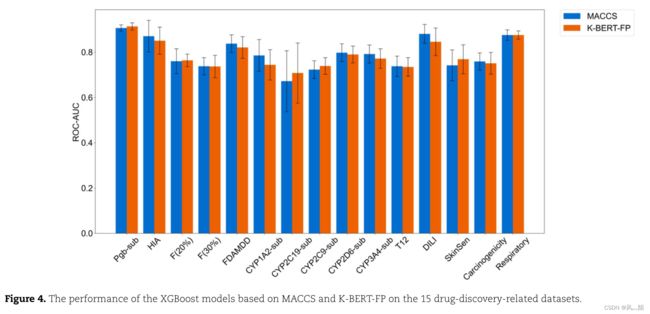
5.5 K-BERT-FP 可以捕获 MACCS 无法捕获的分子大小特征
作者为了证明K-BERT-FP不是MACCS的简单复制,而是能够捕捉到一些MACCS不能捕捉的信息(如分子大小信息)。作者首先比较了K-BERT-FP和MACCS对DrugBank数据集中分子的TMAPs可视化结果。结果如图5所示,K-BERT-FP和MACCS都能够较好的对DrugBank数据集中的分子进行可视化,K-BERT-FP并没有比MACCS更好的组织能力。这可能是由于大分子中的一些分子碎片信息,隐性的包含了分子大小信息,使得MACCS也能够反应分子大小信息。
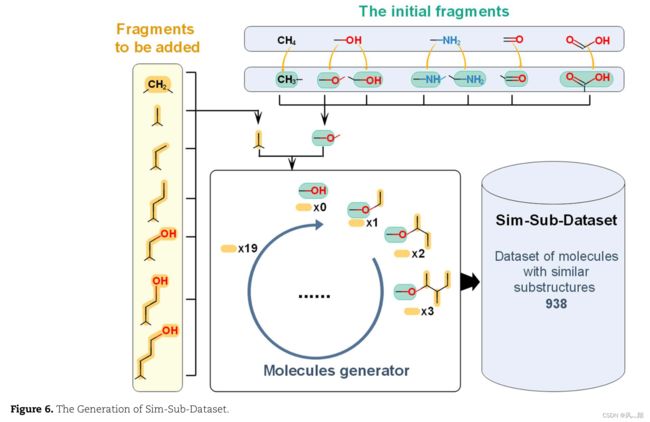
为了说明 K-BERT-FP 可以捕获 MACCS 无法捕获的分子的大小信息,我们构建了一个数据集,其中的分子包含相似的子结构但具有不同的分子大小。如图 6 所示,作者从甲烷、甲醇、甲胺、甲醛和甲酸中生成了 7 个初始片段,并选择了 7 个简单的重复片段。然后,在初始片段的基础上,循环添加20个不同的重复组,生成具有相似子结构的数据集(Sim-Sub-Dataset),这个数据集都是基于相似碎片重复生成的(图6)。由于MACCS只对表征是否包含某一分子碎片,而未表征分子碎片的数量,因此MACCS不能够反应此类分子的分子大小情况。
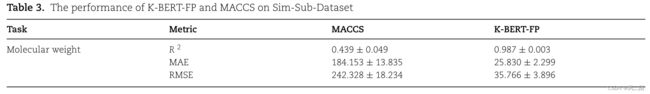
作者比较了K-BERT-FP和MACCS在预测该数据集分子权重的能力。结果如表3所示,K-BERT-FP显著优于MACCS,这说明K-BERT-FP能够捕捉到MACCS不能捕捉的信息。
5.6 K-BERT-FP 可以通过进一步的预训练来捕捉手性特征
作者首先比较了K-BERT和MACCS在表征CHIRAL1数据集上2500个手性异构体的能力。结果如图7A和图7B所示,K-BERT和MACCS并不能较好的区分CHIRAL1上的手性异构体。为了使得K-BERT-FP中包含手性信息,作者将K-BERT在手性数据集CHIRAL1上进行了进一步预训练,K-BERT-FP-CHIRAL1的分子预测预训练任务仍然是MACCS分子指纹,而K-BERT-FP-CHIRAL1-R-S的分子预测预训练任务改为预测分子的手性R/S。结果如图7C和图7D所示,K-BERT-FP-CHIRAL1-R-S能够很好的区分手性异构体。此外,同一组分子的异构体都能从另一组中找到,这说明K-BERT-FP-CHIRAL1-R-S在表征分子手性信息的同时,仍然蕴含着分子的结构信息。
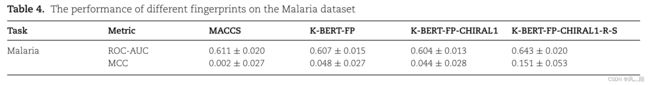
同时,作者比较了不同指纹在手性数据集Malaria上的预测表现(基于XGBoost建模)。计算结果显示,K-BERT-FP-CHIRAL1-R-S优于其他指纹,这说明通过定制化的预训练任务,K-BERT能够关注手性信息,进而提高模型对手性特征的提取能力。
6.结论
作者提出了一种能够像药物化学家一样提取分子特征的预训练策略K-BERT,K-BERT能够较好地从SMILES字符串中提取分子特征,且在成药性预测数据集上表现了较强的预测能力。此外,作者还发现K-BERT能够生成一种通用的分子指纹K-BERT-FP,且K-BERT-FP能够捕捉MACCS不能捕捉的分子大小信息。经过进一步预训练,K-BERT-FP还能够捕捉到手性的信息。这表明,通过对特定任务的了解,可以设置不同的预训练任务来使K-BERT-FP捕捉特定的分子特征信息。
7.关键点
- 提出了基于原子特征预测、分子特征预测和对比学习的三个新的预训练任务,以构建用于分子特性预测的基于知识的 BERT (K-BERT) 预训练模型。
- K-BERT 模型在 15 项小型制药任务中的 8 项中取得了最佳性能,突出了我们的预训练策略在药物发现方面的有效性和优势。
- 对比学习预训练任务使 KBERT 能够“理解”SMILES,而不仅限于规范 SMILES。
- K-BERT 生成的通用指纹 K-BERT -FP 在 15 个药物数据集上表现出与 MACCS 相当的预测能力,并且还可以捕获传统指纹无法捕获的分子大小。
- 通过更改预训练分子任务并在特定数据集中进行进一步预训练,K-BERT 可以生成包含某些特定分子特征(例如手性信息)的定制 K-BERT -FP。