解读16S扩增子分析表格+代码实现
16s分析结果详解
文章目录
-
- 16s分析结果详解
- OTU表解读
- 物种柱状图
- 韦恩图
- 稀释曲线
- Shannon-Winner曲线
- Rank-Abundance曲线
- Alpha多样性(样本内多样性)
-
- Chao1
- Shannon
- Ace
- Simpson
- Alpha多样性指数差异箱形图
- Beta多样性分析(样品间差异分析)
- PCoA分析
- PCA分析
- NMDS分析(非度量多维尺度分析)
- 排序分析
- 样本-物种丰度关联circos弦装图
- Ternary三元相图
- 相关系数图
- LDA差异贡献分析
- 物种进化树的样本群落分布图
OTU表解读
OTU(operational taxonomic units) 是在系统发生学研究或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系,种,属,分组等)设置的同一标志。通常按照 97% 的相似性阈值将序列划分为不同的 OTU,每一个 OTU 通常被视为一个微生物物种。相似性小于97%就可以认为属于不同的种,相似性小于93%-95%,可以认为属于不同的属。样品中的微生物多样性和不同微生物的丰度都是基于对OTU的分析。
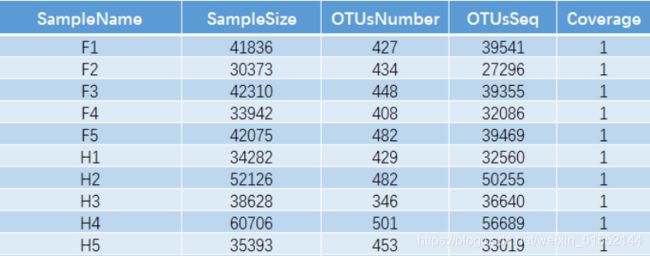
其中SampleName表示样本名称;SampleSize表示样本序列总数;OTUsNumber表示注释上的OTU数目;OTUsSeq表示注释上OTU的样本序列总数。
Coverage是指各样品文库的覆盖率,其数值越高,则样本中序列没有被测出的概率越低。该指数实际反映了本次测序结果是否代表样本的真实情况。
计算公式为:C=1-n1/N 其中n1 = 只含有一条序列的OTU的数目;N = 抽样中出现的总的序列数目。
下表是对每个样本在分类字水平上的数量进行统计,并且在表栺中列出了在每个分类字水平上的物种数目

其中SampleName表示样本名称;
Phylum表示分类到门的OTU数量;
Class表示分类到纲的OTU数量;
Order表示分类到目的OTU数量;
Family表示分类到科的OTU数量;
Genus表示分类到属的OTU数量;
Species表示分类到种的OTU数量。
物种柱状图
横坐标中每一个条形图代表一个样本,纵坐标代表该分类层级的序列数目或比例。同一种颜色代表相同的分类级别。图中的每根柱子中的颜色表示该样本在不同级别(门、纲、目等)的序列数目,序列数目只计算级别最低的分类,例如在属中计算过了,则在科中则不重复计算。
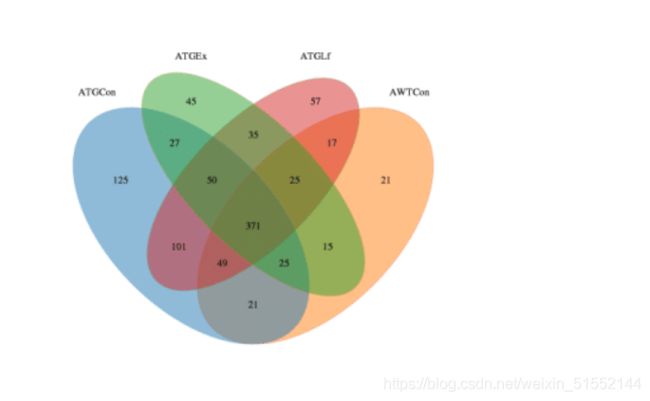
韦恩图
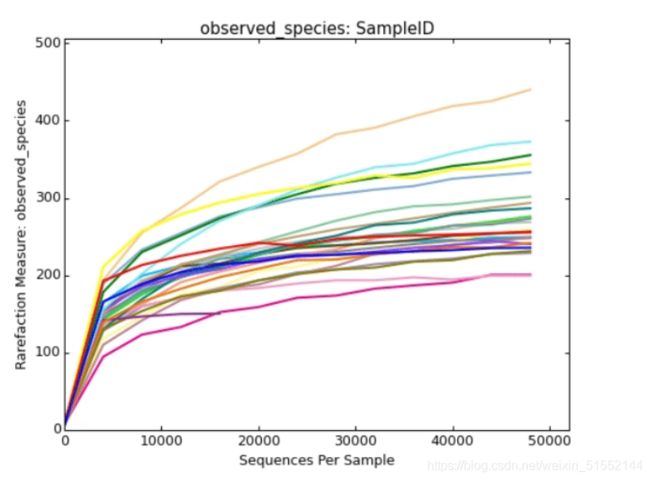
稀释曲线
是用来评价测序量是否足以覆盖所有类群,并间接反映样品中物种的丰富程度。
它是利用已测得16S rDNA序列中已知的各种OTU的相对比例,来计算抽取n个(n小于测得reads序列总数)reads时出现OTU数量的期望值,然后根据一组n值(一般为一组小于总序列数的等差数列)与其相对应的OTU数量的期望值做出曲线来。
当曲线趋于平缓或者达到平台期时也就可以认为测序深度已经基本覆盖到样品中所有的物种,增加测序数据无法再找到更多的OTU;
横坐标代表随机抽取的序列数量;纵坐标代表观测到的OTU数量。样本曲线的延伸终点的横坐标位置为该样本的测序数量。
Shannon-Winner曲线
Shannon-Wiener 曲线,是利用shannon指数来进行绘制的,反映样品中微生物多样性的指数,利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线,以此反映各样本在不同测序数量时的微生物多样性。
当曲线趋向平坦时,说明测序数据量足够大,可以反映样品中绝大多数的微生物物种信息。
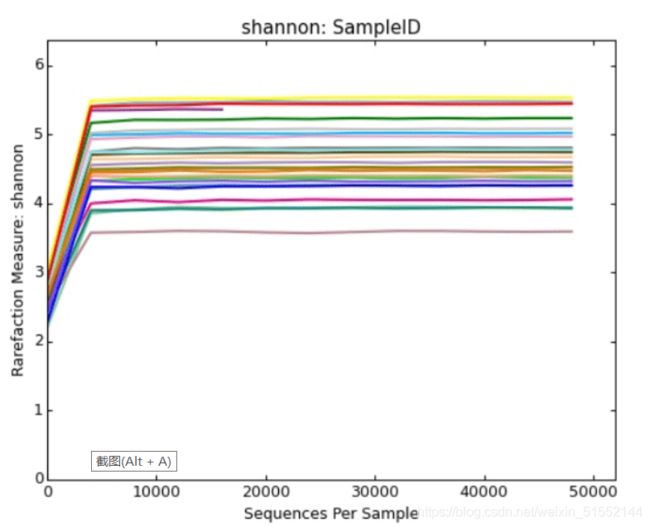
横坐标代表随机抽取的序列数量;纵坐标代表的是反映物种多样性的Shannon指数,样本曲线的延伸终点的横坐标位置为该样本的测序数量。
其中曲线的最高点也就是该样本的Shannon指数,指数越高表明样品的物种多样性越高。
其中,Sobs= 实际测量出的OTU数目;
ni= 含有i 条序列的OTU数目;N = 所有的序列 数。
Rank-Abundance曲线
该曲线用于同时解释样品多样性的两个方面,即样品所含物种的丰富程度和均匀程度。
Rank-abundance曲线是分析多样性的一种方式。构建方法是统计单一样本中,每一个OTU所含的序列数,将OTUs按丰度(所含有的序列条数)由大到小等级排序,再以OTU等级为横坐标,以每个OTU中所含的序列数(也可用OTU中序列数的相对百分含量)为纵坐标做图。
注:横坐标:OTU等级,“300”代表样本中按照丰度排列第300位的OTU;纵坐标:该等级OTU中序列数的相对百分含量,即属于该OTU的序列数除以总序列数,纵坐标轴上数字,例如“100”代表相对丰度为100%,“10”代表相对丰度为10%,依次类推。
- 物种的丰富程度由曲线在横轴上的长度来反映,曲线越宽,表示物种的组成越丰富;
- 物种组成的均匀程度由曲线的形状来反映,曲线越平坦,表示物种组成的均匀程度越高。
Alpha多样性(样本内多样性)
Alpha多样性是指一个特定区域或者生态系统内的多样性,常用的度量指标有
- Chao1 丰富度估计量(Chao1 richness estimator)
- 香农 – 威纳多样性指数(Shannon-wiener diversity index)
- 辛普森多样性指数(Simpson diversity index)等
计算菌群丰度:Chao、ace;
计算菌群多样性:Shannon、Simpson。
Simpson指数值越大,说明群落多样性越高;Shannon指数越大,说明群落多样性越高。
Chao1
用chao1 算法计算群落中只检测到1次和2次的OTU数估计群落中实际存在的物种数。Chao1 在生态学中常用来估计物种总数,Chao1值越大代表物种总数越多。
Schao1=Sobs+n1(n1-1)/2(n2+1)
其中Schao1为估计的OTU数,Sobs为观测到的OTU数,n1为只有一条序列的OTU数目,n2为只有两条序列的OTU数目。
Shannon
用来估算样品中微生物的多样性指数之一。它与 Simpson 多样性指数均为常用的反映 alpha 多样性的指数。Shannon值越大,说明群落多样性越高。
Ace
用来估计群落中含有OTU 数目的指数
Simpson
用来估算样品中微生物的多样性指数之一,Simpson 指数值越大,说明群落多样性越高。

Alpha多样性指数差异箱形图
Beta多样性分析(样品间差异分析)
Beta多样性度量时空尺度上物种组成的变化
PCoA分析
是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,PCoA 可以找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。
PCA分析
主成分分析(Principal component analysis)PCA 是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要的前几位特征值,采取降维的思想,PCA 可以找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。
PCA分析文章
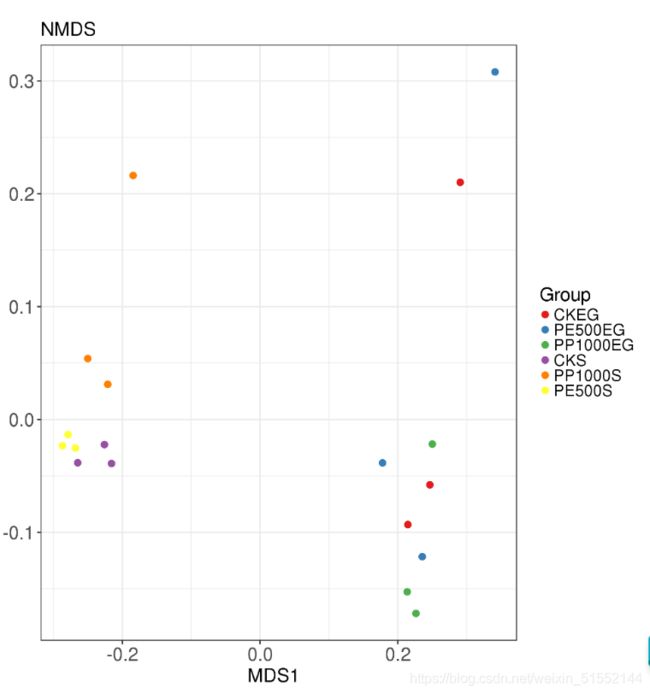
NMDS分析(非度量多维尺度分析)
NMDS(Nonmetric Multidimensional Scaling)常用于比对样本组之间的差异,可以基于进化关系或数量距离矩阵。
排序分析
- PCA,PcoA,NMDS分析都属于排序分析(Ordination analysis)。
排序(ordination)的过程就是在一个可视化的低维空间或平面重新排列这些样本。
目的:使得样本之间的距离最大程度地反映出平面散点图内样本之间的关系信息。
排序又分两种:非限制性排序和限制性排序。
1、非限制性排序(unconstrained ordination)
——只使用物种组成数据的排序
- (1) 主成分分析(principal components analysis,PCA)
- (2) 对应分析(correspondence analysis, CA)
- (3) 去趋势对应分析(Detrended correspondence analysis, DCA)
- (4) 主坐标分析(principal coordinate analysis, PCoA)
- (5) 非度量多维尺度分析(non-metric multi-dimensional scaling, NMDS)
2、限制性排序(constrained ordination)
——同时使用物种和环境因子组成数据的排序
- (1) 冗余分析(redundancy analysis,RDA)
- (2) 典范对应分析(canonical correspondence analysis, CCA)
样本-物种丰度关联circos弦装图
样本与物种的共线性关系circus 图是一种描述样本与物种之间对应关系的可视化圈图,该图不仅反映了每个样本的优势物种组成比例,同时也反映了各优势物种在不同样本之间的分布比例。
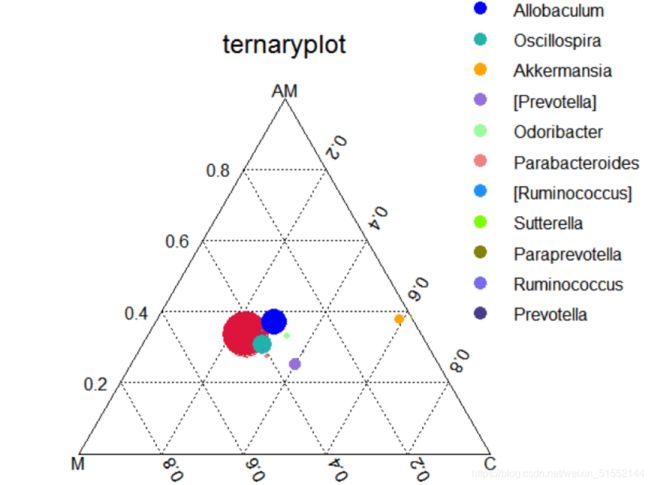
Ternary三元相图
三元相图是重心图的一种,它有三个变量,在一个等边三角形坐标系中,图中某一点的位置代表三个变量间的比例关系。这里表示三组样本之间优势物种的差异,通过三元图可以展示出不同物种在分组中的比重关系。
图解读:三角分别代表三个或三组样本,图中的圆分别代表排名最高哦的属水平的物种,三种颜色分别代表三组不同分组的优势物种,圆圈大小代表物种的相对丰度,圆圈理哪个顶点接近,表示此物种在这个分组中的含量较高。该分析仅限三个样本或三组样本之间分析比较。
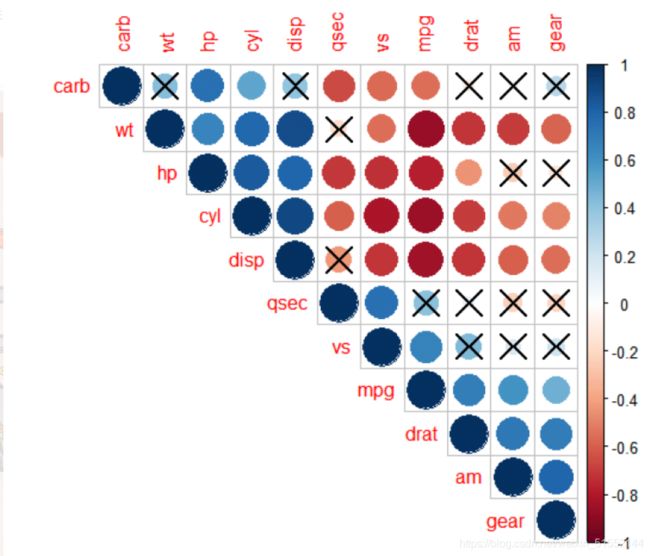
相关系数图
通过R 软件的corrplot 包绘制spearman 相关性热图,并通过该热图可以发现优势物种/样本之间重要的模式与关系。
图解读:蓝色系的为正相关,红色系的为负相关,×表示检验水平下无意义。越靠近颜色条两头,相关系数越大。所以说,我们可以通过实心圆的颜色和大小判断相关的方向和相关系数的大小。
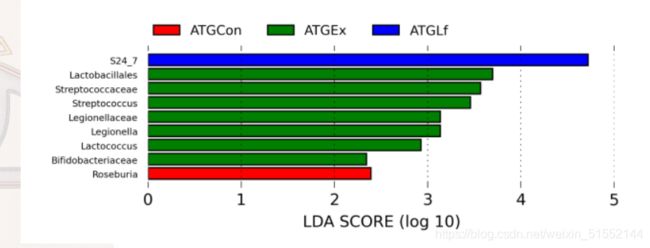
LDA差异贡献分析
LDA
LDA是有监督的,增加了种属之间的信息关系后,结合显著性差异标准测试和线性判别分析的方法进行特征选择。
不同颜色代表不同样本或组之间的显著差异物种。
组间差异显著物种又可以称作生物标记物(biomarkers),这个LDA分析主要是想找到组间在丰度上有显著差异的物种。
物种进化树的样本群落分布图
它是将不同样本的群落构成及分布以物种分类树的形式在一个环图中展示。数据经过分析后,将物种分类树和分类丰度信息通过软件GraPhlAn进行绘制
其目的是将物种之间的进化关系以及不同样本的物种分布丰度和最高分布样本的信息在一个视觉集中的环图中一次展示,其提供的信息量较其他图最为丰富。

REF
【1】刘鹏远, 陈庆彩, 胡晓珂. 渤海湾湾口表层沉积物中的核心细菌群落结构及其对环境因子的响应. 微生物学通报, 2018, 45(9): 1940-1955.