【冷冻电镜】Relion4.0——subtomogram教程
本文是根据原版教程的介绍进行的总结。不得不说Relion是用过的cryoEM和cryoET里最好用、教程最详细最贴心的,respect。就是安装的时候有些许麻烦,使用体验还是不错的。
教程指路:Relion4.0 Tutorial
安装注意事项
因为我之前安装过Relion3.1版本,所以为了使用最新版本,首先需要使用命令git checkout ver4.0更新到4.0版本的代码,然后重新按照教程cmake ..一遍再make,这里注意以前生成的命令所在的位置和现在的不要搞混了,我是把之前的删掉了又重新生成的。(之前保存到/usr/local/bin里面了,这次把相关的命令删了,直接将relion/build/bin所在的路径加到了环境变量PATH里,source一下bashrc,再打开relion的GUI,终于打开的是4.0 beta版本了!)
准备实验数据
这里教程提供了一套数据,来自EMPIAR-10164。大小5个G多,第一次下载的时候中止了,结果解压出来发现少了好多东西,所以一定要注意一下是否下载成功了哈。
指令:
wget ftp://ftp.mrc-lmb.cam.ac.uk/pub/scheres/relion40_sta_tutorial_data.tar
tar -xf relion40_sta_tutorial_data.tar
里面应该有三个子文件夹:tomograms,masks,input。
tomograms里面有5个子文件夹。
预处理
用户需要对层析图进行外部预处理。完成 tomogram 的 movies frames alignment, tilts series alignment, particle picking。
- Tilt series alignment: 使用IMOD完成。
- Initial CTF parameters: CTFFind / CtfPlotter。
- Order list: 倾斜序列获取的时间顺序。
- Fractional electron dose: 每个倾斜图像的电子剂量。
建议为每个项目创建一个目标,来针对每个你想确定的结构。然后在项目目录中启动RELION的GUI。在项目目录中,应该创建一个单独的目录来存储所有IMOD重构的tomograms文件夹,可以命名为tomograms/,在里面应该在TS_1等文件夹中找到不同的层析图。
relion使用倾斜周对齐钱的原始倾斜序列对阵,并从IMOD导入对齐数据。tomogram文件夹中应该包含一个堆栈文件(.st/ .mrc/ .mrcs),newst.com和tilt.com脚本文件,其中包含了转换和倾斜角度文件名和层析图尺寸、偏移量和排除视图。
粒子坐标应该参考使用该信息重建的层析图像。
使用命令来开始项目:
relion --tomo&
导入tomograms
要做的第一步是将层析图像集导入管道。为此,我们必须首先创建一个包含所需信息的 .star描述文件,每个层析图一行。基本字段是倾斜系列堆栈文件名、CTF估计数据文件名和IMOD项目文件夹。例如,本教程中使用的星号文件tomograms_descr.star包含以下字段:
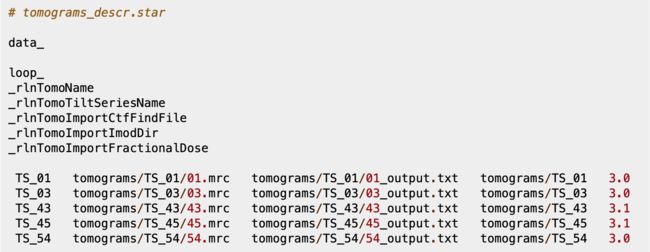
导入的数据集中断层图已经使用 IMOD 配准过,用 CTFFind 或者 CtfPlotter 估计过 CTF 。
rlnTomoName 标签是每个断层图的唯一标识符,如果这个描述文件中没有提供,它会自动创建(更多信息可以在 relion_tomo_import_tomograms 程序帮助中找到)。请注意,在此数据集中,电子剂量不是恒定的,因此应在描述文件中提供,而不是在导入层析成像时作为全局参数提供。
在GUI中,从左侧的任务类型浏览器选择Tomo import,并在Tomograms选项卡上填写以下参数:


可以在名为 Current job: Give_alias_here 的白色字段中为该作业提供一个有意义的别名(例如:tomograms)。
运行 Run! 以后会生成文件夹ImportTomo/job001/,以及到该目录的一个名为ImportTomo/tomograms 的符号链接,里面会生成 tomograms.star 文件。它包含一个具有一般层析图像属性的表,以及每个层析图的特定表,包括投影矩阵、散光散焦、累计辐射剂量和每个倾斜帧的变形等。(参见 relion_tomo_import_tomograms 程序帮助)。
导入坐标
构造伪子断层所需的最小数据是每个粒子的三维坐标集和对应的层析名称。像素坐标应该与newst.com tilt.com文件给出的原始像素大小(Bin1)的尺寸和偏移量相关(参见导入断层图)。在坐标导入过程中,检查所有粒子的层析名称是否存在于相关的层析集 tomograms.star中,并将相应的 data_optics表添加到输出 particles.star 文件(更多信息请参见 relion_tomo_import_particles 程序帮助和 particle set 描述)。
还是选择 Tomo import ,这次填写的时 Coordinates 标签页,而且要在Tomograms 标签页把 Import tomograms 设为 No。

在新创建的目录中会发现导入的粒子集 particles.star 和 optimiser_set.star 文件。如果你在一个不同的项目中预处理你的粒子,你可以使用相同的Tomo导入作业类型在Others选项卡中导入粒子星号文件、3D references、3D masks等。请注意,这不是运行relion的推荐方式,用户负责生成正确的星号文件。
生成 pseudo-subtomograms
在使用任何常规 RELION 程序(那些不是专门用于断层扫描的程序,如relion_refine)进行处理之前,我们首先需要构建单个的伪亚层析图像粒子,这相当于SPA工作流中的颗粒提取过程。
伪亚层析图像粒子文件不仅 与particle set 有关。事实上,它们也依赖于 tomogram set (倾斜序列对齐,离焦估计),如果粒子的运动已经被细化,它们还依赖于 trajectory set 。因此,每次运行 Tomo frame alignment 或 Tomo CTF refinement 作业类型时,都需要重构一组新的更新的伪subtomo粒子。对于特定的 tomo RELION 程序(前缀名relion_tomo_)这是不必要的,因为它们直接由原始的倾斜系列堆栈文件提供,因此引用的伪subtomo 文件将被忽略。
在本教程中,我们将首先构建较低分辨率的粒子,bin比例因子为4,获得一个de novo 3D 模型。
Running the job:
选择 Make pseudo-subtomo 的 IO 选项卡。请注意,输入文件可以通过单独的 optimisation set 和/或 particle set和 tomogram set 文件来覆盖 optimisation set 文件中的文件。这里,我们将在可用的地方使用优化集文件。
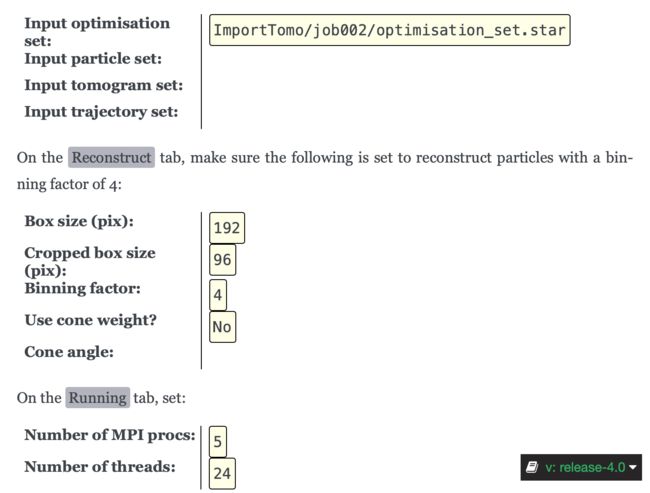
注意,tomo 相关程序的MPI版本是在一个 tomogram 基础上并行化的,也就是说,层析图将分布在多个处理器中。因此,如果MPI进程的数量大于断层扫描的数量,则无法提高性能(Number of MPI processes <= Number of tomograms)。使用上述设置,该任务在作者的系统上只花费了不到10分钟。
伪subtomo粒子 及其相关的CTF和多重模式将被存储到一个名为 PseudoSubtomo/job007/ subtomgrams/ 的新目录下的 MRC 文件中。你可以检查它以及更新的particles.star文件,一个新的 optimation_set.star 也会被创建。
De novo 3D model
RELION-4.0 使用了一个梯度驱动的算法从 pseudo-subtomos 生成一个全新的 3D initial model。在RELION-4.0版本中,该算法不同于 CryoSPARC 中的 SGD 算法。如果有一个合理的角度方向分布,该算法可能会产生一个合适的、低分辨率的模型,随后可以用于 3D classification 或 3D auto-refine。
本教程中使用的示例通常允许在挑选过程中获得垂直于球面的初始方向,这可以用作初始参考。然而,这里我们将展示如何在没有任何先验知识的情况下获得一个全新的模型。
在3D initial model 作业类型的 I/O 选项卡上选择PseudoSubtomo/job007/particles.star。CTF上的一切都已填好。如下填写Optimisation 选项卡(保留默认的角度和偏移采样):


这一步需要的时间比较长。(我后面的每轮迭代都需要40-50分钟了……原文中作者只需要一个下午茶的时间,枯了)
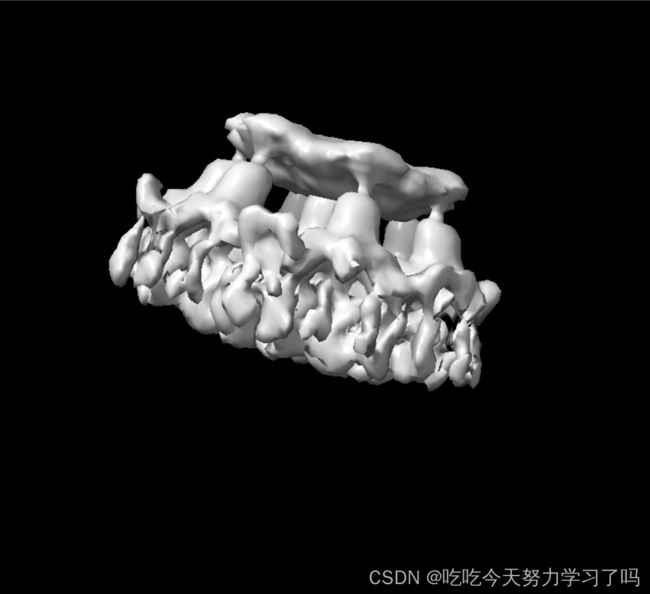
可以使用UCSF chimera这样的3D查看器查看梯度驱动算法(InitialModel/job008/run_it100_class001.mrc)的输出映射。应该确认对称点组是正确的对称轴也是正确的。如果是这样,对称的输出映射(InitialModel/job008/initial_model.mrc)应该类似于梯度驱动算法的输出映射。
在经过了100轮迭代以后,这是我生成的初始结构的结果,用ChimeraX进行可视化:
初始3D refinement
一旦有了初始参考模型(map),就可以使用三维自动细化(3D auto-refine)程序,以完全自动化的方式将数据集细化到高分辨率。该过程采用了 gold-standard 方法,也就是FSC,从独立细化的半重建中计算傅里叶壳相关系数来估计分辨率。
因为和单颗粒分析相比,伪自断层扫描文件需要更多的内存资源,所以建议分几个步骤来运行这个过程,从高的binning factor到1,以提高处理效率。由于初始模型是使用带bin因子4的伪子层析成像进行处理的,因此我们将使用这些相同的粒子开始3D细化。
选择3D auto-refine,按照原教程里的参数填写,该步骤我运行了半天时间(单个3090 GPU),一共迭代了12轮次达到收敛。原教程中作者使用4GPU,只用了一小时的时间。


在 Auto-sampling 选项卡上,通常可以保持默认值。请注意,采样选项卡上的方向采样率只会在前几个迭代中使用,从那里开始,算法将自动增加角度采样率,直到收敛。因此,对于所有小于八面体或二十面体对称的细化,我们通常使用默认的角度采样7.5度,并从采样1.8度进行局部搜索。只有对于更高的对称细化,我们使用3.7度采样,并从0.9度执行局部搜索。这里我们唯一要改变的是设置:

因为MPI节点分配给了一个领导人和两组在两个half-sets上做所有工作的追随者,最有效率的方式是使用奇数个MPI处理器,最小数量的MPI是3。在最后一次迭代时,内存需求可能会显著增加,因为将考虑到Nyquist之前的所有频率,所以对于比本测试数据集中更大的boxes,最好在集群节点上使用尽可能多的线程来运行。

分析结果:
该步骤每一轮迭代会产生两个run_it0**_half*_model.star文件和两个run_it0**_half*_class001.mrc文件,每一个都独立细化了一半的数据。当收敛后会生成一个单独的run_model.star和run_class001.mrc。因为在最后一次迭代中,两个独立的半重建被连接在一起,分辨率通常会在最后一次迭代中显著提升,因为程序将使用所有的数据输出到尼奎斯特频率。这种迭代也需要更多的内存和CPU。角度采样的自动增加是自动优化过程的一个重要方面,它基于一篇文章中解释的信噪比考虑来估计角度和平移赋值的准确性。程序不会使用比它认为必要的更精细的角度和平移采样率,因为那样并不会改善结果。估计的精度和使用的采样率都会存储下来(_optimiser.star,_model.star)。有关更多信息,请查看SPA教程的高分辨率3D细化步骤。
这项工作可能会达到奈奎斯特频率,因此为了达到更高的分辨率,我们将需要一组新的伪subtomo粒子和一个更小的分块因子(2或者1)的参考map。
由于在最初的3D细化中获得的细化地图覆盖了HIV壳和基质,我们需要确保在下一轮细化中使用的mask对齐并且只聚焦于壳。
我们建议将 reference 居中,因为 masks/ 文件夹中提供的蒙版已经居中。可以使用 IMOD 3dmod 这样的3D查看器查看输出的细化映射 (Refine3D/job009/run_class001.mrc) 和掩码(masks/mask_align_bin4.mrc),以估计两个映射之间以像素为单位的Z偏移量。在我们的例子中,它是2.75像素,但这可能是不同的,因为它取决于初始de novo模型。因此,可以通过命令行重新输入粒子:
relion_star_handler --i Refine3D/job009/run_data.star \
--o Refine3D/job009/run_data_z2.75.star --center --center_Z 2.75
为了评估 reference map中的衣壳是否与 mask 对齐,我们可以使用 Tomo reconstruct particle 工作类型来重构它,这将在下一步重构粒子中进行描述。
重建颗粒
从一组 particles 获得 3D reference map 的常用方法是在给定的方向上平均它们提取的伪subtomo图。然而,也可以通过平均倾斜序列中每个粒子的2D投影直接获得参考地图。
在这里,虽然在 Tomo reconstruct particle 中,我们提供了两种选择,我们强烈建议从2D倾斜系列投影重建。这节省了一个插值步骤,并且避免了在伪子层析图构造相关的偏差。它还创建了两个半图和参考图,节省了计算时间。
主要的区别在于:
- 根据 2D tilt series 平均使用
relion_tomo_reconstruct_particle程序,接收输入的优化集文件。另一方面,从3D pseudo-subtomos平均使用常规的relion_reconstruct程序,并要求显式提供输入粒子集文件名。 - 如果选择从2D倾斜序列中取平均值,也可以在同一job中估计FSC曲线。为此,应在 Average选项卡中提供 FSC溶剂掩模(FSC solvent mask)。
在本教程中,我们将使用较小的分块因子重建初始的3D细化后的3D参考地图,以保持细化以获得更高的分辨率。
在 Tomo reconstruct particle jobtype 中选择 IO 选项卡。请注意,我们在重新中心化参考映射后使用粒子集文件,这样我们就可以通过层析图信息直接设置 tomogram set 文件或 optimisation set,从任何前面的步骤中。然后,还应该具体提供 particle set文件。


请注意,从2D tilt series 中用 relion_tomo_reconstruct_particle 程序重构有3个线程参数。这里提供的线程数同时设置了–j 和 --j_out 参数,因此,为了避免超过可用内存资源,–mem 参数也应该设置使用大约 80-90%。在我们的系统中,我们在每个集群节点(64Gb)上运行1个MPI进程。
高分辨率refinement
一旦获得了高分块因子(低分辨率)的map,就可以继续将该数据集细化到更高的分辨率。
此时在本教程中,3d细化可以直接处理使用原始像素大小(bin=1)。但是,由于我们不再需要设置大值低通滤波器作为初始参考,因此建议通过减少分块因子来连续处理3D细化,以加快计算速度。这个初始的低通滤波器防止了对图中高频分量的bias,并在比初始滤波器更高的分辨率上保持了完全独立的细化的FSC。为了克服这个问题,可以使用 halfmap 文件作为 relion_refine 处理的每个半集的参考map,从而保持两个半集的 3D refinement 在整个工作流中独立。为此,当引用映射文件名包含*half?*.mrc,每个halfmap会被自动分配给它的半集。
然后,运行一个带有严格掩码mask /mask_fsc.com的后处理,你应该会得到一个3.6Å左右的分辨率。此时,这是我们在不应用任何特定的tomo细化的情况下可以达到的最佳对齐,如 Tomo CTF refinement 和 Tomo frame refinement 部分所示。
Masks & 后处理
在本教程中,我们使用了自定义mask,是基于几何形状的,使用dynamo包中的 dynamo_mask_GUI 等工具外部创建。对于一般的掩码创建,请参考SPA教程的掩码描述。
用在 subtomogram averaging 的锐化三维参考map和估算金标准FSC曲线步骤与SPA教程中描述的是一样的。
在输入 I/O 中的输入可以选择 optimisation set。输出会创建一个 postprocess_optimisation_set.star 文件,还有参考 FSC 文件。这即将作为 Tomo CTF refinement 和 Tomo frame alignment 的输入。
3D classification
该步骤和SPA教程中的相同。
Tomo CTF refinement
在这一步中,我们将展示如何应用与CTF估计相关的不同参数的细化。即倾斜系列投影散焦和散光、尺度和不对称像差和对称像差。有关参数的完整描述,请查看 relion_tomo_refine_ctf 程序。
在进行 tomo 的 refinement(Tomo CTF refinement 和 Tomo frame alignment)之前,必须运行 Tomo reconstruct particle 去估计出一个 reference map来作为它们的输入。主要原因是投影和重建算法完全匹配,我们要防止在3D auto-refine 工作中,平均地图中伪子层析图构建相关的误差而导致次优结果。
另一方面,reference half maps 的后处理过的FSC数据可以有选择地用于估计信噪比。在没有提供的情况下,这些程序内部计算它没有相位随机,所以信噪比会稍微乐观一些。由于FSC数据估计也集成在 Tomo reconstruct particle 作业中,我们建议在运行Tomo细化作业之前对其进行估计。
请注意,独立于在之前的 3D auto-refine 工作中用的分块因子水平,Tomo CTF refinement 和Tomo frame alignment 协议都以原始像素大小处理数据 (binning 1)。因此,reference map 应该总是在这个 binning 级别被重建,并且应该使用适当的参考和FSC掩模。
使用 Tomo reconstruct particle 的输出 optimisation set 文件作为输入。此时这个优化集中应该包括最初导入的的 tomogram set,从 3D auto-refine 得到的 run_data.star 作为 粒子集。 ReconstructParticleTomo/jobXXX/half<12>.mrc 作为 half maps,ReconstructParticleTomo/jobXXX/PostProcess/postprocess.star 作为 FSC data. 注意在之前的三维细化中使用到的参考mask也要被包含到优化集中。或者如果参考掩码设计为除1以外的binning因子,则应特别提供参考mask。


分析结果:检查输出文件夹 CtfRefineTomo/jobxxx 会发现新的 tomograms.star 和新的 particles.star,带有新的 CTF、scale和Zernike aberrations。
为了评估结果,建议使用新的参数来运行新的 Tomo reconstruct particle 作业,并进行FSC估计。请注意,这个参考映射也将被用作下一次 Tomo frame alignment 作业的输入。与之前的FSC估计相比,我们可以观察到在中高频范围有轻微的改善。
Tomo frame alignment
RELION 也在 2D tomography 上实现了类似贝叶斯抛光(Bayesian polishing)的功能。这个程序改进了将3D空间映射到倾斜系列的图像上的投影。可选地,还可以估计粒子的光束诱导运动轨迹和变形。有关参数的完整描述,请查看 relion_tomo_align 程序。
这里作为输入的优化集已经更新了上一个 Tomo CTF refinement 中的 reference mask文件字段,因此我们不需要覆盖任何其他输入文件。


请注意,每个粒子的运动估计显著增加了处理时间。在我们的系统中,这大约需要2个小时。
分析结果:
在输出文件夹 FrameAlignTomo/jobxxx 中可以看到新的 tomograms.star 和 particles.star 文件,包含了修正过的倾斜序列和颗粒位置,还有一个 trajectory set 文件叫做 motion.star ,里面包含了颗粒的轨迹。同样地,为了评估结果,建议使用新的参数运行一个新的Tomo reconstruct particle 作业,并进行FSC估计。与之前的FSC估计相比,我们应该看到一个明显的改进,分辨率大约为3.5Å。
Tomo refinement cycle
在运行两个tomo特定的细化步骤后,仍然建议运行一个新的 3D auto-refine 工作,以利用改进的 tomograms 和 particles。为此,我们需要构造一组新的 pseudo-subtomos 和 reference maps,如bin=1的伪子层析图和参考图小节所述。对于新的3D自动细化作业,与在bin 1处运行自动细化作业中的参数相同,除了这两个:

这个新的3D细化在我们的系统中花费了1天时间(4个GPU卡),结果为大约3.4Å的分辨率,完成了第一个改进周期。如果再执行一个新的包含Tomo CTF细化,Tomo帧对齐和3D自动细化的循环,应该能得到 3.3Å 左右的分辨率,在第三个周期后可以最终收敛到3.2Å。
以上就是教程中所展示的对于示例数据集的实验全过程。由于demo数据集的对称性较高,可以再对其他的数据集进行实验。
On-the-fly processing
RELION-4.0中,动态处理是基于scheme功能的。程序可以从Project目录的命令行启动:
relion_it.py &
然后会打开 GUI 界面,用户需要填写一些内容。
注意:该脚本依赖于预编译好的 Schemes/prep 和 Schemes/proc Schemes,在 RELION 源码中的 scripts 子目录下。环境变量 RELION_SCRIPT_DIRECTORY 需要指向该目录。也可以指向自己的其他版本的 Schemes。
(其他详细内容请查看原教程。)
A u t h o r : C h i e r Author: Chier Author:Chier


