MONAI(4)—一文看懂各种Transform用法(下)
上小结链接:https://mp.csdn.net/editor/html/113742194
6 裁剪&填充
【SpatialCropd, CenterSpatialCropd, CropForegroundd, RandCropByPosNegLabeld, SpatialPadd】
对于CT或者MRI图像来讲,图像是非常大的,又是一个三维图像,不可能全部输入网络中训练。要么把图像直接Resize到固定的尺寸,要么就是裁剪图像。monai提供了非常多的裁剪模式,包括中心裁剪,前景裁剪和随机裁剪等等,同时图像不够大的话,也可以进行填充。今天介绍几种经常用到的裁剪方式。
注意事项,这些裁剪方式都要求数据格式为通道优先格式(必须有通道维度),也就是说要放在Addchanneld后面使用
- SpatialCropd: 空间裁剪
这个函数是根据提供的空间中心和大小裁剪图像,或者,如果未提供中心和大小,则必须提供ROI的开始和结束坐标来裁剪图像。
参 数 介 绍
roi_center: int, ROI的中心体素坐标,如果是二维图像,则坐标是(x, y), 如果是三维,则坐标是(x, y, z)
roi_size: int, ROI的大小。很好理解,有了中心,再加上大小就可以裁减ROI了
roi_start / roi_end, int, 如果不提供上述两个参数,还可以自己指定ROI的开始结束坐标
from monai.transforms import SpatialCropD, CenterSpatialCropd, CropForegroundd, RandCropByPosNegLabeld,SpatialPadD
loader = LoadImaged(keys=["image", "label"], dtype=np.float32)
add_channel = AddChanneld(keys=["image", "label"])
# 重新加载一个图像
data_dict = loader(data_dicts[4])
datac_dict = add_channel(data_dict) # (1, 512, 512, 61)
crop0 = SpatialCropD(keys=["image", "label"], roi_center=(256,256,30), roi_size=(256,256,30))
# 中心坐标为图像的中点(256,256,30), 大小也是(256,256,30)
data_crop = crop0(datac_dict)
print(data_crop['image'].shape) # (1, 256, 256, 30) 可视化查看crop效果
image, label = datac_dict["image"][0], datac_dict["label"][0]
imagecrop, labelcrop = data_crop["image"][0], data_crop["label"][0]
plt.figure("visualize", (8, 8))
plt.subplot(2, 2, 1)
plt.title("image")
plt.imshow(image[:, :, 30], cmap="gray")
plt.subplot(2, 2, 2)
plt.title("label")
plt.imshow(label[:, :, 30])
plt.subplot(2, 2, 3)
plt.title("croped image")
plt.imshow(imagecrop[:, :, 15], cmap="gray")
plt.subplot(2, 2, 4)
plt.title("croped label")
plt.imshow(labelcrop[:, :, 15])
plt.show()按中心裁是裁不到脾脏的,于是我们可以手动输入起始点坐标
crop1 = SpatialCropD(keys=["image", "label"], roi_start=(0,0,40), roi_end=(256,256,60))
data_crop = crop1(datac_dict)
data_crop['image'].shape # (1, 256, 256, 20)
image, label = datac_dict["image"][0], datac_dict["label"][0]
imagecrop, labelcrop = data_crop["image"][0], data_crop["label"][0]
plt.figure("visualize", (8, 8))
plt.subplot(2, 2, 1)
plt.title("image")
plt.imshow(image[:, :, 40], cmap="gray")
plt.subplot(2, 2, 2)
plt.title("label")
plt.imshow(label[:, :, 40])
plt.subplot(2, 2, 3)
plt.title("croped image")
plt.imshow(imagecrop[:, :, 0], cmap="gray")
plt.subplot(2, 2, 4)
plt.title("croped label")
plt.imshow(labelcrop[:, :, 0])
plt.show()这样就裁剪到目标了。
其实后面的各种裁剪都是会在内部调用SpatialCropd的,只是坐标的获取方式不一样。
- CenterSpatialCropd: 中心裁剪
这个裁剪方式就非常简单了,看参数只需要提供ROI大小,自动给你中心裁剪,而不需要像SpatialCropd一下手动提供中心坐标。
crop2 = CenterSpatialCropd(keys=['image', 'label'], roi_size=(256,256,30))
data_crop = crop2(datac_dict)
print(data_crop['image'].shape) # (1, 256, 256, 30)- CropForegroundd: 裁剪前景
使用边框裁剪图像。通过在使用select_fn选择前景来生成边界框。在边框的每个空间尺寸中添加边距。如果整个医学图像中的有效部分很小,通常用于帮助训练和验证。
简单讲,可以自己定义裁剪的方式,比如按图像值>0的裁剪,按 label 中>0的裁剪等等。反正找出图像中有效的部分用于训练。
注意:CropForegroundd和CropForeground会有些区别,CropForeground不会有label,所以不可能按label的值去选择图像。所以分割的话,建议用字典形式,就可以裁剪出有阳性值的部分。
参 数 介 绍
source_key: str, 按image还是label裁剪图像.这里的key, 就是keys=['image', 'label']中的key。
selecct_fn: 按照什么原则选择前景。默认为选择大于0的值作为前景。比如,如果source_key="image", 则选择的是image>0的部分作为前景
channel_indices: 选择特定的通道
margin: int, 边缘填充的个数。通常用原来的值进行填充。默认为0,则不填充。
start_coord_key:str, 记录前景空间边界框的起始坐标的键(不了解)。
end_coord_key:str,记录前景空间边界框的结束坐标的键(不了解)。
- 举例,首先是一个用普通模式的小例子
from monai.transforms import CropForeground
image = np.array(
[[[0, 0, 0, 0, 0],
[0, 1, 2, 1, 0],
[0, 1, 3, 2, 0],
[0, 1, 2, 1, 0],
[0, 0, 0, 0, 0]]]) # 1x5x5, single channel 5x5 image
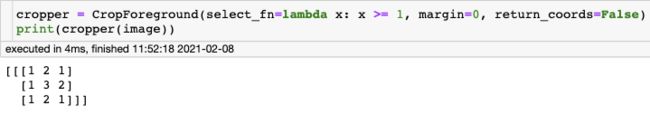
cropper = CropForeground(select_fn=lambda x: x >= 1, margin=0, return_coords=False)
print(cropper(image))就把大于0的值都取到了,假设设定margin=1,也就是周围填充一个像素。
举例2 裁剪image中大于300的像素点,边缘扩充2个像素点
crop3 = CropForegroundd(keys=['image', 'label'], source_key='image', select_fn=lambda x: x > 300, margin=2)
# 裁剪原则为: image中大于300的像素点,边缘扩充2个像素点
data_crop = crop3(datac_dict)
print(data_crop['image'].shape) # (1, 474, 404, 61)注意到其实裁剪出来的图像还是有很多0区域,具体为什么,感兴趣的可以研究一下,并在评论区告诉我一下,谢谢。
举例3 裁剪label中大于0的像素点,并扩充2个像素点
crop3 = CropForegroundd(keys=['image', 'label'], source_key='label', select_fn=lambda x: x > 0, margin=2)
# 裁剪原则为: label中大于0的像素点,边缘扩充2个像素点
data_crop = crop3(datac_dict)
print(data_crop['image'].shape) # (1, 124, 105, 15)可以看出,使用了label来裁剪图像,可以精准定位目标。
- RandCropByPosNegLabeld: 按阴性阳性比裁剪
如果你一幅图像很大,需要裁剪成多个子图,那这个方法就再合适不过了。像是腹部CT这种很大的三维图像,我通常都会使用这个方法,按照阴性阳性比裁剪成4个子图。
参 数 介 绍
label_key: str, 又是一个需要提供key的参数。代表label的键(我们的例子中就是"label"),用于查找前景和背景
spatial_size: [int, int], ROI的大小
pos: float, 与neg一起用于计算将前景体素而不是背景体素选为中心的概率的比率. 选中阳性值的概率为:pos / (pos + neg)
neg: float, 与pos同理。
num_samples: int, 返回多少个子图
image_key: str, 如果提供了image key, 就会使用label==0, 并且image>image_threshold (阈值)的部分作为阴性样本,所以裁剪中心将仅存在于有效图像区域上。
image_threshold: float, 如果使用了image_key, 则提供阈值
fd_indices_key: str, 根据提供的前景索引来裁剪图像,将忽略image_key和 image_threshold
bg_indices_key:str, 需要同fd_indices_key一起提供。典型的是通过FgBgToIndicesd获取索引并缓存结果。
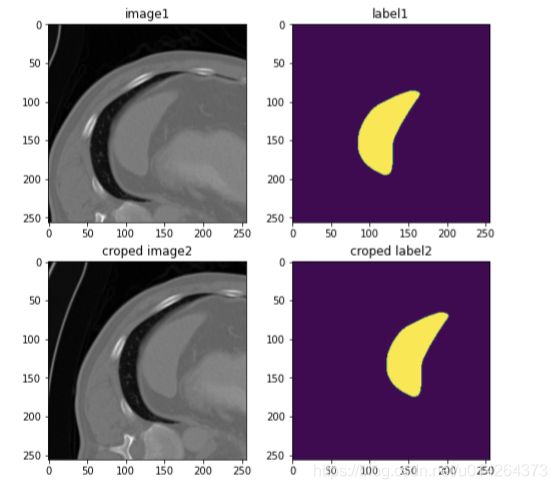
举例 从原图像中裁剪4个大小为(256,256,30)的子图,并且阳性比率为1/2
crop4 = RandCropByPosNegLabeld(keys=["image", "label"], label_key='label', spatial_size=(256,256,30),
pos=1.0, neg=1.0, num_samples=4, image_key='image', image_threshold=200.0)
data_crop = crop4(datac_dict)
print(f'there are {len(data_crop)} croped images')
print('the croped image shape is ', {data_crop[0]['image'].shape}) # 这里返回了4幅图像,所以需要索引可视化其中两幅图像
imagecrop1, labelcrop1 = data_crop[0]['image'][0], data_crop[0]['label'][0]
imagecrop2, labelcrop2 = data_crop[1]['image'][0], data_crop[1]['label'][0]
plt.figure("visualize", (8, 8))
plt.subplot(2, 2, 1)
plt.title("image1")
plt.imshow(imagecrop1[:, :, 15], cmap="gray")
plt.subplot(2, 2, 2)
plt.title("label1")
plt.imshow(labelcrop1[:, :, 15])
plt.subplot(2, 2, 3)
plt.title("croped image2")
plt.imshow(imagecrop2[:, :, 15], cmap="gray")
plt.subplot(2, 2, 4)
plt.title("croped label2")
plt.imshow(labelcrop2[:, :, 15])
plt.show()从图中可以看出,通过label来裁剪图像,每幅图像的差异不是很大。想要阳性更多,加大阳性的比例,想要阴性更多,加大阴性的比例。
- SpatialPadd: 空间填充
裁剪已经学了很多种了,有时还是需要用到填充的。
参 数 介 绍
spatial_size: [int, int], 填充后的空间大小
method: str, 填充的方法, 有两种。"symmetric"(对称填充)(默认方式), "end"(仅在末端填充)
mode: str, 填充值的获取方式,如"constant"(填充0, "edge"(用边缘值填充), "maximum "(最大值填充)
- 举例 填充值=0
pad = SpatialPadD(keys=["image", "label"], spatial_size=(512,512,60), method='symmetric', mode='constant')
data_pad = pad(data_crop[2]) # 把刚才裁剪的图像放大
print(data_pad['image'].shape) # (1, 512, 512, 60)
print(data_pad['image'][0,0,0,0]) # 填充的是0image, label = data_crop[2]["image"][0], data_crop[2]["label"][0]
imagepad, labelpad = data_pad["image"][0], data_pad["label"][0]
plt.figure("visualize", (8, 8))
plt.subplot(2, 2, 1)
plt.title("image")
plt.imshow(image[:, :, 15], cmap="gray")
plt.subplot(2, 2, 2)
plt.title("label")
plt.imshow(label[:, :, 15])
plt.subplot(2, 2, 3)
plt.title("pad image")
plt.imshow(imagepad[:, :, 30], cmap="gray")
plt.subplot(2, 2, 4)
plt.title("pad label")
plt.imshow(labelpad[:, :, 30])
plt.show()注意,填充的0在这里是灰色而不是黑色,因为黑色的值是比0还小的。
- 举例 边缘填充
由于我们直接放大了一倍,这种方式填充起来就更奇怪了。放大的小一些可以用边缘填充。
今天分享了这么多种填充和裁剪的方法,大家都学会了吗。我相信有人带着走,应该会轻松很多吧。剩下的一些变换就要靠大家自己去摸索啦,举一反三,把学习的方法掌握到。
比如还没有讲到的放射变换(RandAffined),方向变换(Orientationd),分辨率变换(Spacingd),高斯模糊(RandGuassianNoised)等都是可以研究的。