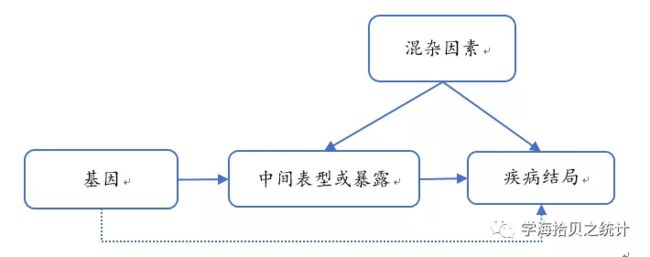
孟德尔随机化研究是一种近年来主要应用于流行病学病因推断上的一种数据分析方式。Katan1986 提出:不同基因型决定不同的中间表型,若该表型代表个体的某暴露特征,用基因型和疾病的关联效应能够代表暴露因素对疾病的作用,由于等位基因遵循随机分配原则,该作用不受传统流行病学研究中的混杂因素和反向因果关联所影响 (反向因果关联,指的是暴露和结局的时间顺序颠倒)。
目前的问题是在观察性研究中得到的结论往往是association而不是causation,而基于孟德尔独立分配定律(配子形成时等位基因随机分配到子代配子中),所以基因和疾病之间的关联不会受到出生后的环境、社会经济地位、行为因素等常见混杂因素的干扰,且因果时序合理,使效应估计值更接近真实情况。
原理:
基础研究证实,疾病发生均可追溯到基因水平,即基因型决定中间表型差异,该中间表型代表某暴露因素作用于该疾病。例如,研究饮酒量引起CHD发病的风险,ALDH2基因多态性决定血中乙醛浓度,后者可影响饮酒行为,从而改变饮酒量,所以血乙醛浓度这一中间表型可间接代表饮酒量。因此,研究基因型和疾病的关联可以模拟暴露因素和疾病的关联。
要推断得暴露因素与疾病结局的因果关系,需满足3个重要的前提条件:
所选基因与中间表型或暴露因素高度相关;
所选基因与混杂因素不相关;
所选基因与疾病结局间条件独立(也即将中间表型或暴露因素和基因型同时作为自变量联立分析时,基因型对疾病结局的效应不再存在)。
满足以上3个条件后,我们才能有理由说明基因是由中间表型介导作用于疾病,也即得到该中间表型或暴露是病因的推断。
MendelianRandomization包可以用来计算孟德尔随机化,它只需要汇总的数据就可以,即只需要回归系数与标准误。
# install package
if (!requireNamespace("MendelianRandomization"))
install.packages("MendelianRandomization")
library(MendelianRandomization)
该包自带两个数据集:
- ldlc, ldlcse, hdlc, hdlcse, trig, trigse, chdlodds, chdloddsse: 是28个SNP位点与LDL-cholesterol, HDL-cholesterol, triglycerides, and
coronary heart disease (CHD) risk表型关联分析的beta-coefficients
和 standard errors (Waterworth et al (2011) "Genetic variants influencing circulating lipid levels and risk of coronary artery
disease", doi: 10.1161/atvbaha.109.201020.); - calcium, calciumse, fastgluc, fastglucse:7个SNP与/CASR/ gene region关联分析,这7个SNP具有相关性,相关性系数存放在calc.rho变量里;
输入的数据包括两方面:
exposure暴露因素比如LDL-cholesterol的beta-coefficients
和 standard errorsoutcome结果比如coronary heart disease的beta-coefficients
和 standard errors
MRInputObject <- mr_input(bx = ldlc,
bxse = ldlcse,
by = chdlodds,
byse = chdloddsse)
MRInputObject # example with uncorrelated variants
MRInputObject.cor <- mr_input(bx = calcium,
bxse = calciumse,
by = fastgluc,
byse = fastglucse,
corr = calc.rho)
MRInputObject.cor # example with correlated variants
输入的数据类型为:
该包支持三种因果评估方法:
- the inverse-variance weighted method
IVWObject <- mr_ivw(MRInputObject,
model = "default",
robust = FALSE,
penalized = FALSE,
correl = FALSE,
weights = "simple",
psi = 0,
distribution = "normal",
alpha = 0.05)
IVWObject <- mr_ivw(mr_input(bx = ldlc, bxse = ldlcse,
by = chdlodds, byse = chdloddsse))
IVWObject
返回的结果为:
- the median-based method
- the MR-Egger method
一个在线的分析网站:http://app.mrbase.org/?state=vdC1XQF7PUQcIpCrIn82&code=4/BQEo30y3p7TdX0sqJbpB3YcSrHtaRMYOz2ixP-s9Gl4qeUWePqJYfWXhobEFXnZoaQ6DRI3YHh4ND-TuxGppXvk&scope=email+profile+https://www.googleapis.com/auth/userinfo.profile+https://www.googleapis.com/auth/userinfo.email
参考:1. 学海拾贝之统计 https://mp.weixin.qq.com/s?src=11×tamp=1542788289&ver=1257&signature=Yy9zB0oHHML4HTgC-mVVyE9L9ByN4LSih72iyF4wW-HFd-C44WTmKoSs5G9YJYo751cuW8fcaBQLqlYQmsx1XwiHxMNQwsljMS7ScP*fqjCFbwXsEHqrPHJGuzKi513I&new=1
- MendelianRandomization v0.3.0: an R package for performing
Mendelian randomization analyses using summarized data