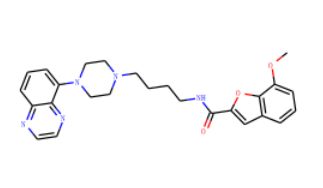
- python画出分子化学空间分布(UMAP)
Sakaiay
python
利用umap画出分子化学空间分布图安装pipinstallumap-learn下面是用一个数据集举的例子importtorchimportumapimportpandasaspdimportnumpyasnpimportmatplotlib.pyplotaspltimportseabornassnsfromsklearn.manifoldimportTSNEfromrdkit.Chemimport
- python版本3.7情况下安装rdkit,torch_geometric,numpy的过程
CWQF-T
pythonnumpy深度学习
前言在上一篇中,我是在anaconda自带的python3.9的环境下安装的torch_geometric,相对来说步骤比较少,一个下午就安装好了,但是在最近接触的一篇论文中需要用到rdkit这个包,我尝试在这个虚拟环境下安装,但是出现了如下提示,表示此包和python3.9不适配-rdkit->python[version='2.7.*|3.5.*|3.6.*|>=2.7,=3.5,=3.6,=
- rdkit安装
qq_46572534
深度学习pythonanaconda
不夸张,我用过最香的rdkit安装引用:https://anaconda.org/rdkit/rdkit
- rdkit 小分子库安装使用;PyRosetta大分子库安装
loong_XL
知识点CADD/AIDDpythonpycharm开发语言
1、rdkit安装使用使用教程参考:https://sites.google.com/view/ml-basics/dealing-with-moleculesgithub地址:https://github.com/rdkit/rdkit1)pip安装python-mpipinstallrdkitpython-c"fromrdkitimportChem;print(Chem.MolToMolBlo
- pthon3.8版本之后rdkit安装之后缺少dll文件的问题
Ai_Chem
python
pthon3.8版本之后rdkit安装之后缺少dll文件的问题描述原因解决方法描述出现:fromrdkit.Chem.rdmolfilesimport*ImportError:DLLloadfailedwhileimportingrdmolfiles:找不到指定的程序。原因使用下面两种方法都是安装的之前版本的rdkit,python3.8之后现在应该是叫rdkit-pypi//两种出错的安装方式c
- RDkit的安装
Alkaid路
conda
首先在官网找到安装教程https://www.rdkit.org/docs/Install.html:2.提示需要更新conda,输入y,回车;3.更新完毕后激活刚刚创建的虚拟环境;4.检验rdkit是否安装成功:输入python进入python环境;importrdkitrdkit.__version__安装成功
- 【RDKit】关于RDKit安装问题,python兼容性问题(最后有RDKit的正确安装方法)
DJ.马
RDKitpython开发语言
//两种出错的安装方式condainstall-cconda-forgerdkitcondainstall-crdkitrdkit使用上面两种方法安装会出现如下报错:fromrdkit.Chem.rdmolfilesimport*ImportError:DLLloadfailedwhileimportingrdmolfiles:找不到指定的程序。切记不要直接conda安装使用下面两种方法都是安装的
- Pycharm使用Conda创建的新环境,并使用terminal下载rdkit
贩卖梦境
RNN用于分子生成pythonanacondapycharm
1.配置anaconda环境变量已经下载安装好anaconda之后,没有添加系统变量,找不到位置,所有系统无法识别conda命令。添加对应的Anaconda环境变量:(以自己的anaconda安装路径为准)D:\programe\anaconda;D:\programe\anaconda\Library\mingw-w64\bin;D:\programe\anaconda\Library\usr\
- 一起学 RDKit Cookbook (1)
wufeil
药物设计rdkitpython机器学习深度学习
这部分内容来自于RDKIT的简单教程:https://www.rdkit.org/docs/Cookbook.htmlRDKIT算是化学生物的神器了,以前每一次都是即时查接口,现在按照这个简易教程走一遍,增加感觉。最好的办法就是全程过一遍。当然啦,在这些过程中,也有我自己对RDKit的一些理解吧。如果有不对的地方,请多多指正!一、画分子Chem.MolFromSmiles()输入的字符串是SMIL
- 机器学习模型的超参数优化用于分子性质预测
wufeil
药物设计机器学习深度学习python
在《预测化学分子的nlogP——基于sklearn,deepchem,DGL,Rdkit的图卷积网络模型》中简单介绍了sklearn模型的使用方法。现在来介绍一下,如何对sklearn模型进行超参数优化。要想获得更好的模型,离不开超参数优化。这里的目的是:示例使用交叉验证结合网格搜索,对机器学习模型进行超参数优化。一、导入相关模块主要是pandas和sklearn的模型及模型评价指标。import
- DeepPurpose 生物化学深度学习库;蛋白靶点小分子药物对接亲和力预测虚拟筛选
loong_XL
CADD/AIDD深度学习人工智能CADD药物对接药物筛选
参考:https://blog.csdn.net/c9Yv2cf9I06K2A9E/article/details/107649770https://github.com/kexinhuang12345/DeepPurpose##安装pipinstallDeepPurposerdkitDeepPurpose包括:数据:关联TDC库下载,是同一作者开发的https://blog.csdn.net/w
- rdkit&networkx | smiles与graph与邻接矩阵相互转换
ASKCOS
AIDDCADD化学生物
fromrdkitimportrdBase,Chemfromrdkit.ChemimportAllChem,Drawfromrdkit.Chem.DrawimportrdMolDraw2DfromIPython.displayimportSVGfrommatplotlib.colorsimportColorConverterimportnetworkxasnximportargparse
- rdkit&python | 标准化检查分子正确性
ASKCOS
AIDDCADD化学生物pythonrdkit
分子结构处理MolecularSanitization默认情况下,RDKit读入分子的时候会进行分子结构检查处理(MolecularSanitization)。结构处理的过程中会自动计算一些有用信息,如杂化、环信息等,保证分子的结构在化学上是合理的:价键合理。Sanitization的检查过程,包含11步。clearComputedProps:清除已有的分子、原子、键的计算性质,如Thisstep
- rdkit&python | 修正化合物中原子价态错误
ASKCOS
AIDDCADD化学生物
错误smiles价态纠正mol=Chem.MolFromSmiles('C[CH2+]1(C#N)CC1')RDKitERROR:[08:03:09]Explicitvalenceforatom#1C,5,isgreaterthanpermittedBOND_TYPE=[0,Chem.rdchem.BondType.SINGLE,Chem.rdc
- rdkit&nlp | smiles数据扩增与smiles标准化
ASKCOS
AIDDCADD化学生物python自然语言处理pytorch
fromrdkitimportChemfromrdkit.Chem.DrawimportIPythonConsolesmi='CC(=O)OC1=CC=CC=C1C(=O)O'mol=Chem.MolFromSmiles(smi)用doRandom产生随机smilesrandom_equivalent_smiles=Ch
- rdkit&python | 消除smiles中的立体结构信息
ASKCOS
AIDDCADD化学生物pythonrdkit
fromrdkitimportChemsmi='CCC/C=C/CC'm=Chem.MolFromSmiles(smi)Chem.MolToSmiles(m,isomericSmiles=False)‘CCC=CCCC’smi='CCCC[C@@H](I)C'm=Chem
- rdkit&python | 基于类药性规则对分子进行筛选
ASKCOS
AIDDCADD化学生物化学信息学
类药性类药性指化合物与已知药物的相似性。具有类药性的化合物并不是药物,但是具有成为药物的可能,这一类化合物称为类药性分子或药物类似物分子。类药性是一个模糊的概念,在药物研发中,类药性研究基于先导化合物之上,可以说类药性分子是高质量的先导化合物。Lipinski五规则类药五原则,是辉瑞公司资深药物化学家ChristopherA.Lipinski在1997年提出的筛选类药分子的基本法则,符合Lipin
- rdkit&python | 超全化合物分子清理方法
ASKCOS
AIDDCADD化学生物rdkit生物信息学化学信息学
分子结构处理MolecularSanitization默认情况下,RDKit读入分子的时候会进行分子结构检查处理(MolecularSanitization)。结构处理的过程中会自动计算一些有用信息,如杂化、环信息等,保证分子的结构在化学上是合理的:价键合理。Sanitization的检查过程,包含11步。clearComputedProps:清除已有的分子、原子、键的计算性质,如Thisstep
- chemoinformatics&python | 化学信息学软件包安装方法集合
ASKCOS
AIDDCADD化学生物RDKit化学信息学
rdkit化学信息学condainstall-cconda-forgerdkitcondainstall-crdkitrdkitpipinstallrdkit-pypi谷歌colab上安装rdkit!curl-Lordkit_installer.pyhttps://git.io/fxiPZimportrdkit_installer%timerdkit_installer.install()indi
- rdkit&python | 分子3D构象与结构优化与描述符计算
ASKCOS
AIDDCADD化学生物rdkitAIpython化学
导入库fromrdkit.ChemimportAllChemfromrdkit.Chem.DescriptorsimportrdMolDescriptorsfromrdkitimportChemfromrdkit.Chem.DrawimportIPythonConsolesmi="COC(=O)Cc1cc(F)cc2c1[C@@H](C)CO2"mol=Chem.MolFromSmile
- nginx django uwsgi配置记录
张大铁
1.配置python环境,MySQL,安装依赖pipinstalldjangosklearnpandasrdkit-pypimysqlclientuwsgi2.安装nginx在nginx下载,编译tarxvfnginx-1.21.3.tar.gzcdnginx-1.21.3/./configure#默认安装路径/usr/local/nginxmakemakeinstall3.配置nginx,启动n
- 【RDKit】化学指纹(fingerprint)
DJ.马
RDKitpython开发语言
转载自:Rdkit|化学指纹(fingerprint)_rdkit分子指纹_发呆的比目鱼的博客-CSDN博客Rdkit|化学指纹(fingerprint)化学指纹(fingerprint)RDKit中还有许多其他类型的分子指纹可供选择,以下是其中几个常见的分子指纹类型:Morgan指纹(Circularfingerprints):基于分子的拓扑结构和半径参数生成的二进制指纹,可以用于描述分子的结
- RDkit | 安装报错及使用
跳动的喵尾巴
Rpython问题汇总r语言python
关于RDKit的学习及介绍:RDKit安装基础教程:[GettingStartedwithRDKitinPython]RDkit四:数据处理过程中smiles编码的清洗统一化reticulate-RInterfacetoPython在RStudio中加载rdkit.Chem和rdkit.Chem.rdmolops时,报错,根据保存内容提示,是需要先安装python的,具体安装及设置参考:Pytho
- RDkit四:数据处理过程中smiles编码的清洗统一化
随便叫点什么……
RDkit数据挖掘人工智能
之前写过一篇博客介绍smiles编码,smart编码及摩根指纹(ECFP):(1条消息)RDkit:介绍smiles编码,smart编码及摩根指纹(ECFP)_随便叫点什么……的博客-CSDN博客smiles编码具有唯一性:每个SMILES编码对应唯一一个化学结构,刚刚接触smiles编码的时候一定会有这样的疑问,为什么同一个化学分子smiles编码有多种表示形式:c1cccc(c1OC(=O)C
- RDKit:基于分子文件输出分子结构
AspirinCode
#!/usr/bin/python3fromrdkitimportChemfromrdkit.ChemimportDrawspl=Chem.SDMolSupplier('molecules.sdf')#读入分子库mols=[]formolinspl:mols.append(mol)#获取前十个分子mols=mols[:10]#输出分子结构img=Draw.MolsToGridImage(mols,
- python argument types in rdkit.chem.rdmolfiles.moltosmiles(nonetype) did not match c++ signature
Mr John
python
运行rdkit时报如下错误:pythonargumenttypesinrdkit.chem.rdmolfiles.moltosmiles(nonetype)didnotmatchc++signature出现问题时首先分析可能的报错原因rdkit包出现错误,这时候需要卸除原来的rdkit包,并安装新的包待处理的mol文件格式有错误,如果报这个错误就需要查看文件的错误注意:切记不要一报错就认为是软件包
- rdkit bug: ArgumentError: Python argument types did not match C++ signature
药学人的AI世界
python
condaforge重装rdkit解决方案参考链接:ArgumentError:Pythonargumenttypesinrdkit.Chem.rdMolDescriptors.CalcCrippenDescriptors(NoneType)didnotmatchC++signature:·Issue#87·rdkit/conda-rdkit(github.com)
- `OSError: file error: bad input file` in RDkit with .sdf file
R$refresh
DebugOSError
在我用RDKit读取SDF文件时出现,但是本质上是因为路径不对。所以,在出现OSError:fileerror:badinputfile时,仔细检查输入文件的路径参考https://stackoom.com/question/3l8zb
- RDKit新手入门
AIDD Learning
RDKit机器学习笔记python机器学习
RDKit新手入门一、RDKit是什么?开源的化学信息工具包采用了商业友好的BSD协议核心数据结构和算法用C++实现通过Boost.Python技术对RDKit进行封装,提供Python2/Python3的接口通过SWIG技术提供Java和C#接口提供了大量对化学分子2D/3D的计算操作生成用于机器学习的分子描述符基于PostgreSQL搭建分子数据库KNIME中的化学信息计算支持(https:/
- RDKit(2023.09.1 )环系统模板
DrugAI
RDKit化学信息学与AIpythonRDKit化学信息学
环境Python3.9RDKit2023.09.1fromrdkitimportChemfromrdkit.ChemimportrdDepictorfromrdkit.ChemimportDrawfromrdkit.Chem.DrawimportIPythonConsoleIPythonConsole.ipython_useSVG=True%matplotlibinlineimportrdkitp
- Hadoop(一)
朱辉辉33
hadooplinux
今天在诺基亚第一天开始培训大数据,因为之前没接触过Linux,所以这次一起学了,任务量还是蛮大的。
首先下载安装了Xshell软件,然后公司给了账号密码连接上了河南郑州那边的服务器,接下来开始按照给的资料学习,全英文的,头也不讲解,说锻炼我们的学习能力,然后就开始跌跌撞撞的自学。这里写部分已经运行成功的代码吧.
在hdfs下,运行hadoop fs -mkdir /u
- maven An error occurred while filtering resources
blackproof
maven报错
转:http://stackoverflow.com/questions/18145774/eclipse-an-error-occurred-while-filtering-resources
maven报错:
maven An error occurred while filtering resources
Maven -> Update Proje
- jdk常用故障排查命令
daysinsun
jvm
linux下常见定位命令:
1、jps 输出Java进程
-q 只输出进程ID的名称,省略主类的名称;
-m 输出进程启动时传递给main函数的参数;
&nb
- java 位移运算与乘法运算
周凡杨
java位移运算乘法
对于 JAVA 编程中,适当的采用位移运算,会减少代码的运行时间,提高项目的运行效率。这个可以从一道面试题说起:
问题:
用最有效率的方法算出2 乘以8 等於几?”
答案:2 << 3
由此就引发了我的思考,为什么位移运算会比乘法运算更快呢?其实简单的想想,计算机的内存是用由 0 和 1 组成的二
- java中的枚举(enmu)
g21121
java
从jdk1.5开始,java增加了enum(枚举)这个类型,但是大家在平时运用中还是比较少用到枚举的,而且很多人和我一样对枚举一知半解,下面就跟大家一起学习下enmu枚举。先看一个最简单的枚举类型,一个返回类型的枚举:
public enum ResultType {
/**
* 成功
*/
SUCCESS,
/**
* 失败
*/
FAIL,
- MQ初级学习
510888780
activemq
1.下载ActiveMQ
去官方网站下载:http://activemq.apache.org/
2.运行ActiveMQ
解压缩apache-activemq-5.9.0-bin.zip到C盘,然后双击apache-activemq-5.9.0-\bin\activemq-admin.bat运行ActiveMQ程序。
启动ActiveMQ以后,登陆:http://localhos
- Spring_Transactional_Propagation
布衣凌宇
springtransactional
//事务传播属性
@Transactional(propagation=Propagation.REQUIRED)//如果有事务,那么加入事务,没有的话新创建一个
@Transactional(propagation=Propagation.NOT_SUPPORTED)//这个方法不开启事务
@Transactional(propagation=Propagation.REQUIREDS_N
- 我的spring学习笔记12-idref与ref的区别
aijuans
spring
idref用来将容器内其他bean的id传给<constructor-arg>/<property>元素,同时提供错误验证功能。例如:
<bean id ="theTargetBean" class="..." />
<bean id ="theClientBean" class=&quo
- Jqplot之折线图
antlove
jsjqueryWebtimeseriesjqplot
timeseriesChart.html
<script type="text/javascript" src="jslib/jquery.min.js"></script>
<script type="text/javascript" src="jslib/excanvas.min.js&
- JDBC中事务处理应用
百合不是茶
javaJDBC编程事务控制语句
解释事务的概念; 事务控制是sql语句中的核心之一;事务控制的作用就是保证数据的正常执行与异常之后可以恢复
事务常用命令:
Commit提交
- [转]ConcurrentHashMap Collections.synchronizedMap和Hashtable讨论
bijian1013
java多线程线程安全HashMap
在Java类库中出现的第一个关联的集合类是Hashtable,它是JDK1.0的一部分。 Hashtable提供了一种易于使用的、线程安全的、关联的map功能,这当然也是方便的。然而,线程安全性是凭代价换来的――Hashtable的所有方法都是同步的。此时,无竞争的同步会导致可观的性能代价。Hashtable的后继者HashMap是作为JDK1.2中的集合框架的一部分出现的,它通过提供一个不同步的
- ng-if与ng-show、ng-hide指令的区别和注意事项
bijian1013
JavaScriptAngularJS
angularJS中的ng-show、ng-hide、ng-if指令都可以用来控制dom元素的显示或隐藏。ng-show和ng-hide根据所给表达式的值来显示或隐藏HTML元素。当赋值给ng-show指令的值为false时元素会被隐藏,值为true时元素会显示。ng-hide功能类似,使用方式相反。元素的显示或
- 【持久化框架MyBatis3七】MyBatis3定义typeHandler
bit1129
TypeHandler
什么是typeHandler?
typeHandler用于将某个类型的数据映射到表的某一列上,以完成MyBatis列跟某个属性的映射
内置typeHandler
MyBatis内置了很多typeHandler,这写typeHandler通过org.apache.ibatis.type.TypeHandlerRegistry进行注册,比如对于日期型数据的typeHandler,
- 上传下载文件rz,sz命令
bitcarter
linux命令rz
刚开始使用rz上传和sz下载命令:
因为我们是通过secureCRT终端工具进行使用的所以会有上传下载这样的需求:
我遇到的问题:
sz下载A文件10M左右,没有问题
但是将这个文件A再传到另一天服务器上时就出现传不上去,甚至出现乱码,死掉现象,具体问题
解决方法:
上传命令改为;rz -ybe
下载命令改为:sz -be filename
如果还是有问题:
那就是文
- 通过ngx-lua来统计nginx上的虚拟主机性能数据
ronin47
ngx-lua 统计 解禁ip
介绍
以前我们为nginx做统计,都是通过对日志的分析来完成.比较麻烦,现在基于ngx_lua插件,开发了实时统计站点状态的脚本,解放生产力.项目主页: https://github.com/skyeydemon/ngx-lua-stats 功能
支持分不同虚拟主机统计, 同一个虚拟主机下可以分不同的location统计.
可以统计与query-times request-time
- java-68-把数组排成最小的数。一个正整数数组,将它们连接起来排成一个数,输出能排出的所有数字中最小的。例如输入数组{32, 321},则输出32132
bylijinnan
java
import java.util.Arrays;
import java.util.Comparator;
public class MinNumFromIntArray {
/**
* Q68输入一个正整数数组,将它们连接起来排成一个数,输出能排出的所有数字中最小的一个。
* 例如输入数组{32, 321},则输出这两个能排成的最小数字32132。请给出解决问题
- Oracle基本操作
ccii
Oracle SQL总结Oracle SQL语法Oracle基本操作Oracle SQL
一、表操作
1. 常用数据类型
NUMBER(p,s):可变长度的数字。p表示整数加小数的最大位数,s为最大小数位数。支持最大精度为38位
NVARCHAR2(size):变长字符串,最大长度为4000字节(以字符数为单位)
VARCHAR2(size):变长字符串,最大长度为4000字节(以字节数为单位)
CHAR(size):定长字符串,最大长度为2000字节,最小为1字节,默认
- [强人工智能]实现强人工智能的路线图
comsci
人工智能
1:创建一个用于记录拓扑网络连接的矩阵数据表
2:自动构造或者人工复制一个包含10万个连接(1000*1000)的流程图
3:将这个流程图导入到矩阵数据表中
4:在矩阵的每个有意义的节点中嵌入一段简单的
- 给Tomcat,Apache配置gzip压缩(HTTP压缩)功能
cwqcwqmax9
apache
背景:
HTTP 压缩可以大大提高浏览网站的速度,它的原理是,在客户端请求网页后,从服务器端将网页文件压缩,再下载到客户端,由客户端的浏览器负责解压缩并浏览。相对于普通的浏览过程HTML ,CSS,Javascript , Text ,它可以节省40%左右的流量。更为重要的是,它可以对动态生成的,包括CGI、PHP , JSP , ASP , Servlet,SHTML等输出的网页也能进行压缩,
- SpringMVC and Struts2
dashuaifu
struts2springMVC
SpringMVC VS Struts2
1:
spring3开发效率高于struts
2:
spring3 mvc可以认为已经100%零配置
3:
struts2是类级别的拦截, 一个类对应一个request上下文,
springmvc是方法级别的拦截,一个方法对应一个request上下文,而方法同时又跟一个url对应
所以说从架构本身上 spring3 mvc就容易实现r
- windows常用命令行命令
dcj3sjt126com
windowscmdcommand
在windows系统中,点击开始-运行,可以直接输入命令行,快速打开一些原本需要多次点击图标才能打开的界面,如常用的输入cmd打开dos命令行,输入taskmgr打开任务管理器。此处列出了网上搜集到的一些常用命令。winver 检查windows版本 wmimgmt.msc 打开windows管理体系结构(wmi) wupdmgr windows更新程序 wscrip
- 再看知名应用背后的第三方开源项目
dcj3sjt126com
ios
知名应用程序的设计和技术一直都是开发者需要学习的,同样这些应用所使用的开源框架也是不可忽视的一部分。此前《
iOS第三方开源库的吐槽和备忘》中作者ibireme列举了国内多款知名应用所使用的开源框架,并对其中一些框架进行了分析,同样国外开发者
@iOSCowboy也在博客中给我们列出了国外多款知名应用使用的开源框架。另外txx's blog中详细介绍了
Facebook Paper使用的第三
- Objective-c单例模式的正确写法
jsntghf
单例iosiPhone
一般情况下,可能我们写的单例模式是这样的:
#import <Foundation/Foundation.h>
@interface Downloader : NSObject
+ (instancetype)sharedDownloader;
@end
#import "Downloader.h"
@implementation
- jquery easyui datagrid 加载成功,选中某一行
hae
jqueryeasyuidatagrid数据加载
1.首先你需要设置datagrid的onLoadSuccess
$(
'#dg'
).datagrid({onLoadSuccess :
function
(data){
$(
'#dg'
).datagrid(
'selectRow'
,3);
}});
2.onL
- jQuery用户数字打分评价效果
ini
JavaScripthtmljqueryWebcss
效果体验:http://hovertree.com/texiao/jquery/5.htmHTML文件代码:
<!DOCTYPE html>
<html xmlns="http://www.w3.org/1999/xhtml">
<head>
<title>jQuery用户数字打分评分代码 - HoverTree</
- mybatis的paramType
kerryg
DAOsql
MyBatis传多个参数:
1、采用#{0},#{1}获得参数:
Dao层函数方法:
public User selectUser(String name,String area);
对应的Mapper.xml
<select id="selectUser" result
- centos 7安装mysql5.5
MrLee23
centos
首先centos7 已经不支持mysql,因为收费了你懂得,所以内部集成了mariadb,而安装mysql的话会和mariadb的文件冲突,所以需要先卸载掉mariadb,以下为卸载mariadb,安装mysql的步骤。
#列出所有被安装的rpm package rpm -qa | grep mariadb
#卸载
rpm -e mariadb-libs-5.
- 利用thrift来实现消息群发
qifeifei
thrift
Thrift项目一般用来做内部项目接偶用的,还有能跨不同语言的功能,非常方便,一般前端系统和后台server线上都是3个节点,然后前端通过获取client来访问后台server,那么如果是多太server,就是有一个负载均衡的方法,然后最后访问其中一个节点。那么换个思路,能不能发送给所有节点的server呢,如果能就
- 实现一个sizeof获取Java对象大小
teasp
javaHotSpot内存对象大小sizeof
由于Java的设计者不想让程序员管理和了解内存的使用,我们想要知道一个对象在内存中的大小变得比较困难了。本文提供了可以获取对象的大小的方法,但是由于各个虚拟机在内存使用上可能存在不同,因此该方法不能在各虚拟机上都适用,而是仅在hotspot 32位虚拟机上,或者其它内存管理方式与hotspot 32位虚拟机相同的虚拟机上 适用。
- SVN错误及处理
xiangqian0505
SVN提交文件时服务器强行关闭
在SVN服务控制台打开资源库“SVN无法读取current” ---摘自网络 写道 SVN无法读取current修复方法 Can't read file : End of file found
文件:repository/db/txn_current、repository/db/current
其中current记录当前最新版本号,txn_current记录版本库中版本