Genome Biology:人体各部位微生物组时间序列分析
文章目录
- 人体各部位微生物组初探
- 摘要
- 背景
- 结果
- 结论
- 点评
- 主要结果
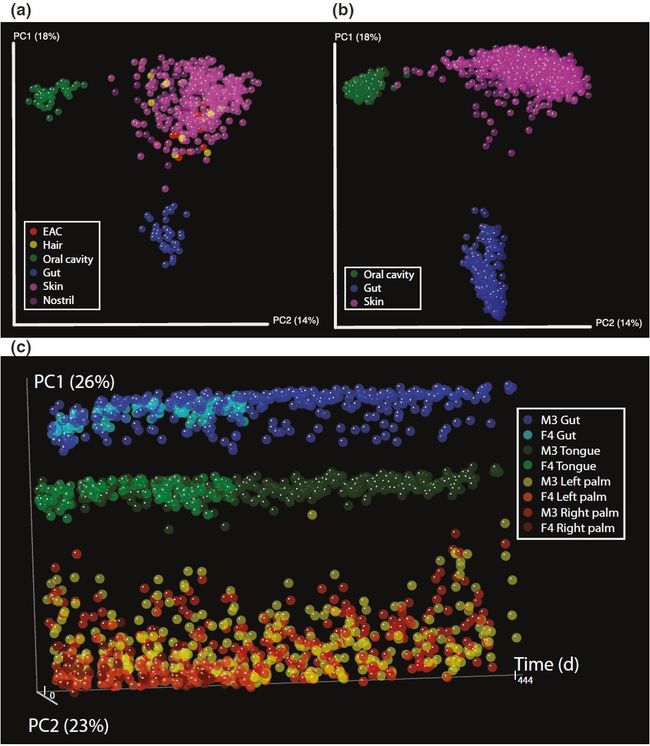
- 图1. 基于无权重UniFrac距离的PCoA
- 图2. 时间上的核心微生物组
- 图3. 群落中的成员关联
- 猜你喜欢
- 写在后面

人体各部位微生物组初探
Moving pictures of the human microbiome
Genome Biology, [14.028]
2011-05-30 Articles
DOI: https://doi.org/10.1186/gb-2011-12-5-r50
第一作者J Gregory Caporaso1
通讯作者:Rob Knight1,8*
其它作者:
Christian L Lauber, Elizabeth K Costello, Donna Berg-Lyons, Antonio Gonzalez, Jesse Stombaugh, Dan Knights, Pawel Gajer, Jacques Ravel, Noah Fierer, Jeffrey I Gordon
作者主要单位:
1美国科罗拉多大学化学与生物化学系(Department of Chemistry and Biochemistry, University of Colorado, 215 UCB, Boulder, CO, 80309, USA)
8霍华德休斯医学研究所(Howard Hughes Medical Institute, University of Colorado, 215 UCB, Boulder, CO, 80309, USA)
摘要
背景
了解人类微生物组的正常时间变化对于开发微生物组相关疾病(如肥胖症,克罗恩病,炎症性肠病和营养不良)的治疗方法至关重要。 然而,测序和计算技术一直是进行人类微生物组大量时间序列分析的限制因素。 在这里,我们提出了迄今为止最大的人类微生物组时间序列分析,覆盖了两个人的四个身体部位共396个时间点。
结果
我们发现,尽管身体部位和个体之间存在稳定的差异,但个体的微生物群在数月,数周甚至数天内存在明显的变异。 此外,在单个身体部位中仅发现总分类群的一小部分似乎在所有时间点都存在,表明没有核心时间微生物组存在高丰度(尽管可能存在一些微生物但低于检测阈值)。 更多的分类群似乎是持久但非永久性的群落成员。
结论
这里描述的DNA测序和计算进展提供了超越我们人类相关微生物生态学的不常见快照的能力,以及在身体栖息地和个体之间和长时间内的时间变化的高分辨率评估。 这种能力将使我们能够定义正常的变异和病理状态,并评估对治疗干预的反应。
点评
此文是微生物组时间序列分析的代表作品,目前引用700多次。同时本文中的所有作者,目前基本都成为了世界知名教授也是一大亮点。此文的数据,在QIIME和QIIME 2时代,同时入选为教程中的示例数据,所以刚入行的小伙伴,有必要读一下此文了解时间序列分析的基本实验设计和分析思路。
主要结果
图1. 基于无权重UniFrac距离的PCoA
Principal coordinates analysis of unweighted UniFrac distances between samples
(a)Costello等人的样本。 按取样部位着色显示不同身体部分微生物组的异同。
(b)M3,F4时间序列样本。 按身体部位着色显示不同身体部分微生物组的异同。
(c)M3,F4时间序列,PC1与时间(天)。 按时间轴展示探索时间梯度上的变化。
图(a,b)和(c)显示了两个独立的主坐标分析。 比较Costello等人454数据(a)、具有时间序列Illumina数据(b),这些数据是在每个样本500个序列的UniFrac距离的单个主坐标分析中生成的。图(c)不包含454数据,因此利用Illumina上可能增加的采样深度(对于UniFrac计算,每个样本均匀采样到5,000个序列)。
图2. 时间上的核心微生物组
Temporal core microbiome
物种级操作分类单位(OTU)的组成部分由核心微生物群组成,其中OTU必须存在的样本数被视为核心的一部分。
即按每个OTU在样本中出现的频率绘制的曲线。发现共有OTU随样本量上升而下降,只有极少OTU存在于所有样品中。
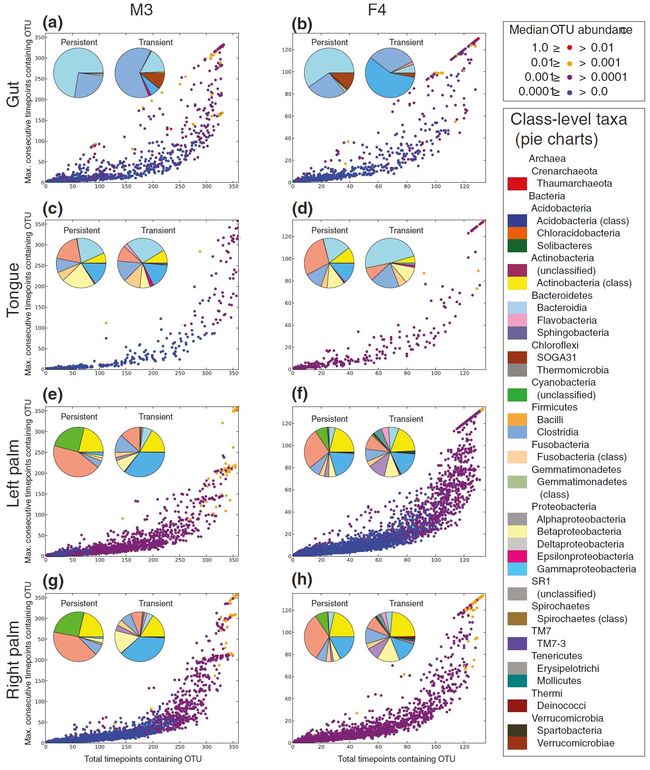
图3. 群落中的成员关联
Community membership
所有OTU的群落成员关系摘要(a)M3肠道,(b)F4肠道,(c)M3舌,(d)F4舌,(e)M3左掌,(f)F4左掌,(g)M3右 手掌,和(h)F4右手掌。 点是通过在它们出现的所有样本上计算的中值相对丰度而着色的OTU,并且饼图总结了作为持久和瞬态OTU观察到的类级别分类单元。突出共有和时间点特有OTU分类上的不同。
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()