NBT:牛瘤胃微生物组的参考基因组集
文章目录
- 牛瘤胃微生物组的参考基因组集
- 热心肠日报
- 摘要
- 主要结果
- 图1. 来自牛瘤胃4,941个末培养和Hungate系列基因组的进化树
- 图2. 末培养基因组与Hungate和发表数据比较
- 图3. Illumina和nonopare宏基因组装统计数据比较
- 图4. 预测碳水化合物代谢蛋白与CAZy数据库的相似度
- 图5. 蛋白质的分类学和功能分布
- 方法
- 宏基因组的组装和分箱
- 宏基因组分类
- 基因组质量和比较基因组
- 绵羊甲烷数据分析
- 瘤胃普查分析
- 纳米孔序列数据的组装和分析
- 蛋白质组分析
- 代码可用
- Reference
- 猜你喜欢
- 写在后面
[外链图片转存失败(img-4hUMgabG-1565163532280)( http://210.75.224.110/Note/LiuYongXin/190802NBTrumen/0.png)]
牛瘤胃微生物组的参考基因组集
用于瘤胃微生物组生物学和酶发现的4,941个瘤胃宏基因组组装基因组的汇编
Compendium of 4,941 rumen metagenome-assembled genomes for rumen microbiome biology and enzyme discovery
Nature Biotechnology [IF:31.864]
2019-08-02 Articles
DOI: https://doi.org/10.1038/s41587-019-0202-3
全文可开放获取 https://www.nature.com/articles/s41587-019-0202-3.pdf
第一作者:Robert D. Stewart1
通讯作者:Mick Watson1*
其它作者:Marc D. Auffret2, Amanda Warr1, Alan W. Walker3, Rainer Roehe2
作者单位:
1 英国爱丁堡大学,罗斯林研究所和皇家(迪克)兽医研究院(The Roslin Institute and the Royal (Dick) School of Veterinary Studies, University of Edinburgh, Easter Bush, UK)
2 苏格兰乡村学院,英国爱丁堡(Scotland’s Rural College, Edinburgh, UK)
3 英国阿伯丁大学,Rowett研究所(The Rowett Institute, University of Aberdeen, Aberdeen, UK)
热心肠日报
https://www.mr-gut.cn/papers/read/1066063689?kf=xread_daily
创作:刘永鑫 审核:刘永鑫 08月06日
原标题:用于瘤胃微生物组生物学和酶发现的4,941个瘤胃宏基因组组装基因组的汇编
- 瘤胃微生物组在反刍动物消化植物物质中非常重要,但大多数组分末被培养;
- 基于283个牛胃样本获得6.5T二、三代测序数据,组装分箱得到4941个宏基因组组装基因组(MAG),同时鉴定了40多万个碳水化合物代谢相关的基因;
- 本文提出一种组装工作流程,可获得80%完整的细菌和古细菌基因组;
- 此参考基因集将宏基因组数据可比对率从15%的提高至50-70%,有助于更好地了解瘤胃微生物组的结构和功能。
主编评语:反刍动物为全世界数十亿人提供必需的肉、奶等重要营养。瘤胃是一种特殊的胃,适应植物来源的复合多糖的分解。瘤胃微生物组的基因组编码数千种适于消化植物物质的酶,它们主导反刍动物饮食结构。本文对近三百个牛胃样本进行宏基因组二、三代混合测序,获得了近五千个宏基因组组装的基因和40多万个碳水化合物代谢相关基因,为深入研究牛瘤胃微生物组提供了参考基因组,可进一步挖掘功能基因和筛选高转化效率菌种提供基础。同时也是近期第三篇Nanopare技术参与发表在自然生物技术杂志在宏基因组领域的文章,可见新技术在宏基因组中应用带来的巨大优势。另两篇详见 https://www.mr-gut.cn/papers/read/1088940721
和 https://www.mr-gut.cn/papers/read/1074446025
更重要的是本文提供了宏基因组分箱、下游分析的全套流程和方法标准,值得参考,详见方法和代码。此外,一个重要的研究趋势是参考基因集已经有点过时了,要发表就得要参考基因组集,即知道功能,更要清楚功能的来源菌种。
摘要
反刍动物为全世界数十亿人提供必需的营养。瘤胃是一种特殊的胃,适应植物来源的复合多糖的分解。瘤胃微生物组的基因组编码数千种适于消化植物物质的酶,它们主导反刍动物饮食结构。作者使用来自283只反刍动物牛的大约6.5TB的短读长和长读长序列数据,组装了4,941个瘤胃微生物的宏基因组组装基因组(MAG)。作者提出了一种基因组分辨的宏基因组学工作流程,该工作流程能够组装至少80%完整的细菌和古细菌基因组。值得注意的是,我们获得了三个由长读长数据组装而成的单重叠群,即瘤胃细菌的全染色体组装,其中两个代表以前未知的瘤胃物种。使用我们的瘤胃基因组集合,我们预测并注释了大量的瘤胃蛋白。我们的瘤胃MAG集将瘤胃宏基因组测序序列的比对率从15%提高到50-70%。这些基因组和蛋白质资源将有助于更好地了解瘤胃微生物组的结构和功能。
Ruminants provide essential nutrition for billions of people worldwide. The rumen is a specialized stomach that is adapted to the breakdown of plant-derived complex polysaccharides. The genomes of the rumen microbiota encode thousands of enzymes adapted to digestion of the plant matter that dominates the ruminant diet. We assembled 4,941 rumen microbial metagenome-assembled genomes (MAGs) using approximately 6.5 terabases of short- and long-read sequence data from 283 ruminant cattle. We present a genome-resolved metagenomics workflow that enabled assembly of bacterial and archaeal genomes that were at least 80% complete. Of note, we obtained three single-contig, whole-chromosome assemblies of rumen bacteria, two of which represent previously unknown rumen species, assembled from long-read data. Using our rumen genome collection we predicted and annotated a large set of rumen proteins. Our set of rumen MAGs increases the rate of mapping of rumen metagenomic sequencing reads from 15% to 50–70%. These genomic and protein resources will enable a better understanding of the structure and functions of the rumen microbiota.
主要结果
图1. 来自牛瘤胃4,941个末培养和Hungate系列基因组的进化树
Fig. 1: Phylogenetic tree of 4,941 RUGs from the cattle rumen, additionally incorporating rumen genomes from the Hungate collection.
[外链图片转存失败(img-NAhiNAPr-1565163532282)(http://210.75.224.110/Note/LiuYongXin/190802NBTrumen/1.png)]
本树采用PhyloPhlAn基于同源蛋白序列连接后构建,并使用GraPhlAn可视化。标签展示只选择不重叠有信息的Hungate基因组名称(Hungate数据集有410个培养的瘤胃细菌和古菌)。
The tree was produced from concatenated protein sequences using PhyloPhlAn, and subsequently drawn using GraPhlAn. Labels show Hungate genome names, and were chosen to be informative but not overlap.
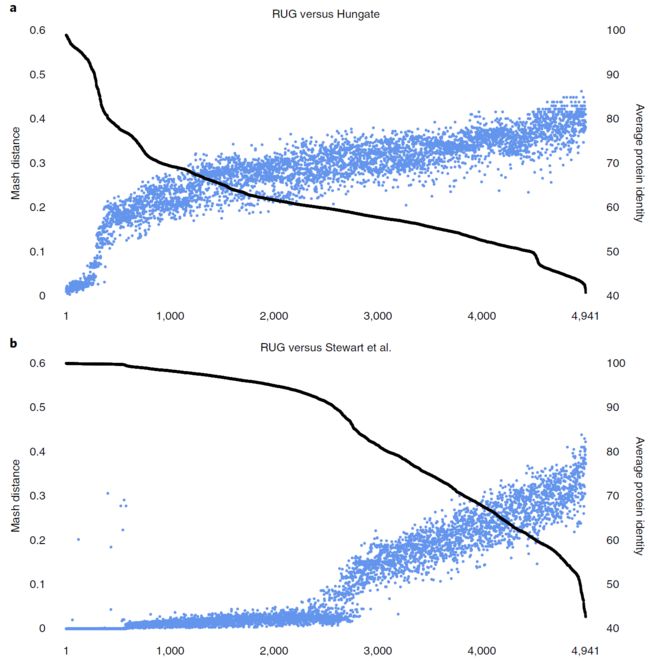
图2. 末培养基因组与Hungate和发表数据比较
Fig. 2: A comparison of the RUG dataset with the Hungate collection and previously published data.
a,b,4,941个RUG与Hungate集合的比较(a)和我们之前公布的Stewart等人的数据(b)。黑线表示蛋白质与最接近匹配的平均百分比(右手y轴),蓝点表示每个RUG与比较数据集中最接近的匹配之间的混合距离(k = 100,000, 衡量两个不同DNA序列之间的差异) 。 正如预期,高蛋白质相似度与低mash距离相关,反之亦然。 RUG由a和b的平均蛋白质相似度独立分类。 图2b中有一个明显的拐点,大约是沿x轴的一半,蛋白质同一性下降到90%以下,混合距离上升,整齐地展示了我们新的更大数据集所代表的新颖性。
a,b, A comparison of the 4,941 RUGs with the Hungate collection (a) and our previously published data from Stewart et al.8 (b). The black line indicates the average percentage protein identity with the closest match (right-hand y axis), and blue dots indicate the mash distance (k = 100,000) between each RUG and the closest match in the comparison dataset (a measure of dissimilarity between two DNA sequences). As expected, a high protein identity relates to a low mash distance, and vice versa. The RUGs are sorted independently by average protein identity for a and b. There is a clear inflection point in Fig. 2b, roughly half way along the x axis, where the protein identity dips below 90% and the mash distance rises, neatly demonstrating the novelty represented by our new larger dataset.
图3. Illumina和nonopare宏基因组装统计数据比较
Fig. 3: A comparison of Illumina and nanopore metagenomic assembly statistics.
彩色直方图显示了282 Illumina组装的统计分布,并突出显示了单个纳米孔组装。 a,N50值。 b,组装的总长度。 c,最长重叠群的长度。 纳米孔组装N50为268kb,比平均Illumina组装(4.7kb)长56倍以上,Illumina组装总长度通常更长(平均600M b),纳米孔组装总长度(长度为178Mb)不是 我们获得的组装中最短的,纳米孔组装产生3.8 Mb的最长重叠群,比Illumina组件的平均长度(479 kb)长7倍,比最长的单Illumina重叠群长1.74倍(1.38 Mb; 13个重叠群中的一个来自99.19%完全度且未培养的拟杆菌RUG14538)。 就直接比较而言,相同样品的仅Illumina组装的N50为12.2kb,总长度为247Mb,最长的重叠群为358kb。
The colored histograms show the distribution of statistics for 282 Illumina assemblies, and the single nanopore assembly is highlighted. a, N50 values. b, Total length of the assembly. c, Length of the longest contig. The nanopore assembly N50 of 268 kb was over 56 times longer than that for the average Illumina assembly (4.7 kb), the Illumina assemblies were often longer (average of 600 Mb), the nanopore assembly (at 178 Mb in length) was not the shortest of the assemblies we produced and the nanopore assembly produced the longest contig at 3.8 Mb, seven times longer than the average for the Illumina assemblies (479 kb) and 2.74 times longer than the longest single Illumina contig (1.38 Mb; one of 13 contigs from the 99.19% complete uncultured Bacteroidia bacterium RUG14538). In terms of a direct comparison, the Illumina-only assembly of the same sample had an N50 of 12.2 kb, a total length of 247 Mb and a longest contig of 358 kb.
图4. 预测碳水化合物代谢蛋白与CAZy数据库的相似度
Fig. 4: Maximum percentage identity between CAZyme-predicted proteins from the RUGs and the CAZy database.
GH,糖苷水解酶(n = 235,001); GT,糖基转移酶(n = 120,494); PL,多糖裂解酶(n = 6,834); CE,碳水化合物酯酶(n = 55,523); AA,辅助活性; CBM,碳水化合物结合模块(n = 23,928); SLH,S层同源结构域(n = 150); cohesin,黏连蛋白结构域(n = 80)。 中心线表示中值; 方框显示了四分位数范围; 并且胡须延伸到最极端的数据点,但数据点不超过盒子四分位数范围的1.5倍。
GH, glycoside hydrolase (n = 235,001); GT, glycosyl transferase (n = 120,494); PL, polysaccharide lyase (n = 6,834); CE, carbohydrate esterase (n = 55,523); AA, auxiliary activities; CBM, carbohydrate-binding module (n = 23,928); SLH, S-layer homology domain (n = 150); cohesin, cohesin domain (n = 80). Center lines indicate the median value; boxes show the interquartile range; and whiskers extend to the most extreme data point that is no more than 1.5 times the interquartile range from the box.
图5. 蛋白质的分类学和功能分布
Fig. 5: Taxonomic and functional distribution of proteins.
[外链图片转存失败(img-6zbKInp2-1565163532283)(http://210.75.224.110/Note/LiuYongXin/190802NBTrumen/5.png)]
顶部,12门和未知细菌的蛋白质总数。
中间,蛋白质组预测为CAZymes的百分比。
底部,八个CAZyme类的分布占预测CAZymes总数的比例。
Top, total number of proteins for 12 phyla and the group of unknown bacteria. Middle, percentage of the proteome predicted to be CAZymes. Bottom, distribution of eight CAZyme classes as a proportion of the total number of predicted CAZymes.
方法
宏基因组的组装和分箱
Metagenomic assembly and binning
该研究总共测序了282个样品,每个样品产生0.24-1.4亿个PE150碱基对的读长(7.2 - 42 GB)。 样品在五批48个样品和一批42个样品中进行测序(这42个样品批次是Stewart等人的唯一基础)。 另一个样品用于Stewart等人的Hi-C测序,并且来自该样品的宏基因组装的基因组包括在去冗余数据集中。
除非另有说明,否则所有使用的参数均为默认值。 使用如前所述的覆盖率和含量单独组装和分箱每个样品。 简而言之,每个样本使用idba_ud49(v.1.1.3)和选项’–num_threads 16 --pre_correction --min_contig 300’组装。 BWA MEM50(v.0.7.15)用于将读段比对至过滤的组装重叠群,Samtools(v.1.3.1)用于转换为BAM格式。 MetaBAT2(v.2.11.1)中的脚本jgi_summarize_bam_contig_depths用于计算生成的BAM文件的覆盖范围。 还使用MEGAHIT(v.1.1.1)为六批样品中的每一批产生了一个共同组装,参数‘–kmin-1pass -m 60e+10 --k-list 27,37,47,57,67,77,87 --min-contig-len 1000 -t 16’。
使用MetaBAT2和选项’–minContigLength 2000 --minContigDepth 2’将宏基因组分箱应用于单样品组件和共组装。 单样品分箱共生产37,153个箱,共组装分箱再生产23,335个箱。 所有60,743个箱子合并在一起,然后使用dRep(v.1.1.2)进行去重复。 dRep去重复工作流程选项为’dereplicate_wf -p 16 -comp 80 -con 10 -str 100 -strW 0’。 因此,在预过滤中,只有CheckM评估的箱(v.1.0.5)具有≥80%的完整性和≤10%的污染,用于成对去重复比较(n = 10,586)。 Bin评分为完整性 - 5×污染+ 0.5×log(N50),并且只有来自每个二级簇的最高评分RUG保留在去冗余组中。 对于我们的数据集,获得了4,941个去重复的RUG。
请注意,我们运行了连续的重复数据删除工作流程。 因此,我们之前发布的所有913个RUG(包括MetaBAT2和Hi-C)都已与新的RUG合并,并且已经去冗余。 因此,虽然此处发布的较新数据集中存在一些先前发布的RUG,但许多已被更高质量的新RUG取代。
补充数据5给出了每个样本中每个基因组的平均深度,如MetaBAT2(参考文献52)(v.2.11.1)包中的脚本jgi_summarize_bam_contig_depths所计算。
宏基因组分类
Metagenomic assignment
宏基因组分箱的输出只是一组含有假定基因组的DNA FASTA文件。使用CheckM(v.1.0.5)评估这些是否完整和污染。使用MAGpy分析了4,941个最佳分箱,这是一个Snakemake工作流程对分箱进行了一系列分析,包括CheckM(v.1.0.5); prodigal(v2.6.3)蛋白质预测; Pfam_Scan(v.1.6); DIAMOND(v.0.9.22.123)搜索UniProt TrEMBL; PhyloPhlAn(v.0.99)和sourmash(v.2.0.0)搜索所有公共细菌基因组。 MAGpy结果用于为每个分箱产生推定的分类学分配,如下所示:
-
如果分配给一个物种的蛋白质比例≥0.9且平均氨基酸同一性(average amino acid identity)≥0.95,则根据DIAMOND结果分配给种(species);
-
如果sourmash评分≥0.8,则根据sourmash结果分配给种(species);
-
如果PhyloPhlAn概率很高且分类水平是属(genus)或种,则根据PhyloPhlAn结果分配分类;
-
如果分配到一个属的蛋白质的比例≥0.9且平均氨基酸同一性≥0.9,则根据DIAMOND结果分配给属(genus);
-
如果PhyloPhlAn概率高或中等且水平是属(genus),则根据PhyloPhlAn结果分配到属;
-
如果PhyloPhlAn概率高或中等且水平为科(family),则根据PhyloPhlAn结果分配至科;
-
如果分配给一个科的蛋白质比例≥0.8且平均氨基酸同一性≥0.6,则根据DIAMOND结果分配给科(family);
-
如果PhyloPhlAn概率为高或中等且水平为目(order),则根据PhyloPhlAn结果分配给目;
-
如果分配给命令的蛋白质比例≥0.6且平均氨基酸同一性≥0.6,则根据DIAMOND结果分配给目(order);
-
如果PhyloPhlAn概率高或中等且等级为纲(class),则根据PhyloPhlAn结果分配给纲;
-
如果PhyloPhlAn概率高或中等且水平为门(phylum),则根据PhyloPhlAn结果分配给门;
-
其它,根据CheckM世系分配分类。
重要的是,在这个阶段,这些只是推断分类学。使用这些标签、FigTree(v.1.4.3),iTol(v.4.3)、PhyloPhlAn(v.0.99)和GraPhlAn(v.0.9.7)从连接的蛋白质子序列产生由RUG和来自Hungate集合的基因组组成的系统发育树。 注释在它们可能的地方得到了改进 - 例如,MAGpy只在类别上属水平分配一个分类,但基因组与在物种水平注释的Hungate 1,000基因组紧密聚类,注释将被更新。还使用FigTree在Bacteria-Archaea分支处手动设置根。
基因组质量和比较基因组
Genome quality and comparative genomics
使用CheckM(v.1.0.5)评估基因组完整性和污染(见上文)。 使用tRNAscan-SE(v.2.0.0)注释tRNA基因,并使用barrnap(v.0.9)预测16S rRNA基因。 使用promer用MUMmer(v.3.23)计算全基因组比对,以发现基因组之间的匹配率。 使用FastANI(v.1.1)计算氨基酸同一性ANI。 使用DIAMOND blastp(v.0.9.22.123)和MASH(v.2.0,参数’-k 21 -s100000’)将RUG与Hungate集合和我们之前的数据集进行比较。
使用如上所述的dRep对瘤胃超集合进行去冗余,对于大于99%ANI的菌去冗余’参数 -sa 0.99’,对于95%ANI的去冗余’参数-sa为0.95’。 使用UpSetR(v.1.3.3)绘制组之间的重叠。 读长物种分类使用kraken(v.0.10.5)计算,参数’–fastq-input --gzip-compressed --preload --paired’。
绵羊甲烷数据分析
Analysis of sheep methane data
基于Shi等人的低和高甲烷样品中数据,使用kraken分配到瘤胃超集数据库的不同分类水平。 得到的计数数据用作为DESeq2(v.1.22.2)的输入用于差分分析。 使用DESeq2中的plotPCA()函数创建主成分分析图,并使用gplots包(v.3.0.1.1)中的heatmap.2()函数创建热图。 对于株水平分析,从Shi等人的低甲烷和高甲烷样品中读长 使用BWA-MEM(v.0.7.15)直接与瘤胃超集数据库比对,并将每个基因组的主要比对计数用作DESeq2的输入。所有比较的P值由DESeq2计算并使用多重检验校正。
瘤胃普查分析
Rumen census analysis
每个数据集中每个基因组的平均深度和总深度(补充数据5)用作数据集中丰度的代表。 将Kraken(如上所述)与瘤胃超集数据库一起使用以计算所有样品中变形菌门丰度。
纳米孔序列数据的组装和分析
Assembly and analysis of nanopore sequence data
使用poRe(v.0.24)提取纳米孔读长并进行质量控制,并使用具有默认设置和基因组大小= 150Mb参数下的Canu(v.1.8)组装。 使用MAGpy分析所得组装结果。使用Minimap2(v.2.12)将Illumina的短读长数据比对纳米孔组装结果(参数-x sr),再使用Nanopolish(v.0.10.2)和Racon(v.1.3.1)校正组装结果 。 使用IDEEL(https://github.com/mw55309/ideel)提取查询与目标长度数据并绘图。 使用promer使用MUMmer(v.3.23)计算全基因组比对,以发现基因组之间的匹配。 使用Prokka(v.1.13.3)注释三个完整的纳米孔细菌基因组及其Illumina对应物。 创建纳米孔组装结果,其最小重叠长度为1 kb; 因此在比较之前Illumina组装有相似的限制条件。
蛋白质组分析
Proteome analysis
使用Prodigal(v.2.6.3)和选项’-p meta’预测蛋白质。 使用DIAMOND,针对KEGG(2018年9月15日下载),UniRef100,UniRef90和UniRef50(2018年10月3日下载)和CAZy(dbCAN2版本,2018年7月31日)搜索每种蛋白质。 蛋白质预测结果由CD-HIT(v.4.7)以100%,90%和50%的同一性聚类,与UniRef的方法类似。
使用dbCAN2和HMMER(v.3.1b2)针对CAZy数据库搜索所有蛋白质预测,并使用PULpy预测拟杆菌RUG的PUL。
代码可用
Code availability
比较基因组分析采用MAGpy (https://github.com/WatsonLab/MAGpy),它于去年发表于Bioinformatics,用于下游分析宏基因组组装基因组的分析流程; PUL分析使用PULpy (https://github.com/WatsonLab/PULpy),用于多糖预测的流程;分析nanopore 数据的插入和缺失采用using IDEEL (https://github.com/mw55309/ideel)
注:WatsonLab即为本文通讯作者
Reference
文章链接:https://www.nature.com/articles/s41587-019-0202-3
PDF:https://www.nature.com/articles/s41587-019-0202-3.pdf
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA