相似基因
题目背景
大家都知道,基因可以看作一个碱基对序列。它包含了44种核苷酸,简记作A,C,G,TA,C,G,T。生物学家正致力于寻找人类基因的功能,以利用于诊断疾病和发明药物。
在一个人类基因工作组的任务中,生物学家研究的是:两个基因的相似程度。因为这个研究对疾病的治疗有着非同寻常的作用。
题目描述
两个基因的相似度的计算方法如下:
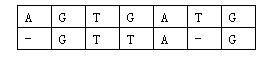
对于两个已知基因,例如AGTGATGAGTGATG和GTTAGGTTAG,将它们的碱基互相对应。当然,中间可以加入一些空碱基-,例如:
这样,两个基因之间的相似度就可以用碱基之间相似度的总和来描述,碱基之间的相似度如下表所示:
那么相似度就是:(-3)+5+5+(-2)+(-3)+5+(-3)+5=9(−3)+5+5+(−2)+(−3)+5+(−3)+5=9。因为两个基因的对应方法不唯一,例如又有:
相似度为:(-3)+5+5+(-2)+5+(-1)+5=14(−3)+5+5+(−2)+5+(−1)+5=14。规定两个基因的相似度为所有对应方法中,相似度最大的那个。
输入输出格式
输入格式:
共两行。每行首先是一个整数,表示基因的长度;隔一个空格后是一个基因序列,序列中只含A,C,G,TA,C,G,T四个字母。1 \le1≤序列的长度\le 100≤100。
输出格式:
仅一行,即输入基因的相似度。
输入输出样例
输入样例#1: 复制
7 AGTGATG 5 GTTAG
输出样例#1: 复制
14
线性DP。
可以类比于求最大公共子序列。设d[i][j]为到a串的i与b串的j为止的最大相似度。则有转移方程:
f[i][j]=max{ f[i-1][j-1]+cost(a[i],b[j]),f[i][j-1]+cost(‘-’,b[j]),f[i-1][j]+cost(a[i],’-’) }
只有三种状态,f[i-1][j-1],f[i-1][j]和f[i][j-1]
#include
#include
#define MAXN 110
using namespace std;
int f[MAXN][MAXN],c[333];
int w[5][5]={
{5,-1,-2,-1,-3},
{-1,5,-3,-2,-4},
{-2,-3,5,-2,-2},
{-1,-2,-2,5,-1},
{-3,-4,-2,-1,-1<<30}
};
int n,m;
char a[MAXN],b[MAXN];
int cost(char x, char y)
{
return w[c[x]][c[y]];
}
int main()
{
c['A']=0; c['C']=1; c['G']=2; c['T']=3; c['-']=4;
scanf("%d%s%d%s",&m,a+1,&n,b+1);
for(int i = 1; i <= m; ++i) //b是空串,a和‘-’配对
f[i][0] = f[i-1][0] + cost(a[i],'-');
for(int i = 1; i <= n; ++i)//a是空串,b和 ‘-’配对
f[0][i] = f[0][i-1] + cost('-',b[i]);
for(int i = 1; i <= m; ++i)
{
for(int j = 1; j <= n; ++j)
{
f[i][j] = f[i-1][j-1]+cost(a[i],b[j]);
f[i][j] = max(f[i][j],f[i-1][j]+cost(a[i],'-'));
f[i][j] = max(f[i][j],f[i][j-1]+cost('-',b[j]));
}
}
printf("%d",f[m][n]);
return 0;
}