数据挖掘-层次聚类与K均值算法的R实现
本次为学生时期所写的实验报告,代码程序为课堂学习和自学,对网络程序有所参考,如有雷同,望指出出处,谢谢!
基础知识来自教材:李航的《统计学习方法》
本人小白,仍在不断学习中,有错误的地方恳请大佬指出,谢谢!
一、层次聚类代码
1.前置预备函数
##############1:计算类与类之间的欧式距离##########
my_dist<-function(X,Y,p){
#X,Y为两个类,p为变量的维数
X=matrix(X,ncol=p)
Y=matrix(Y,ncol=p)
n1=nrow(X)
n2=nrow(Y)
if(n1==1&n2==1){ #两向量距离
d=sqrt((X-Y)%*%t(X-Y))
}else if(n1!=1&n2==1){ #单个向量到类(多个向量)之间的距离(最短距离法)
d=min(as.matrix(dist(rbind(Y,X)))[-1,1])
}else if(n1==1&n2!=1){
d=min(as.matrix(dist(rbind(X,Y)))[-1,1])
}else if(n1!=1&n2!=1){ #类(多向量)到类(多向量)之间的距离(最短距离法)
d=min(as.matrix(dist(rbind(X,Y)))[-c(1:n1),1:n1])
}
d
}
##############2:寻找指定数据在原始数据矩阵中的位置##########
index_find<-function(X,data,p){ #data为给定的数据
data=matrix(data,ncol=p)
n=nrow(data)
index=0
for (j in 1:nrow(data)) {
for (k in 1:nrow(X)) {
TF=as.numeric(data[j,]==X[k,])
if(sum(TF)==p){
index=append(index,k)
}
}
}
index=index[-1]
}
##############3:为保证不出现分支,确定数据在聚类图中的特定排序#########
order_find<-function(indexlist,n){
order=0
#基本思想:从后往前,提取每一次聚类的两个类中含有数据较少的那个类中的数据
for (k in length(indexlist):1) {
nk=rep(NA,length(indexlist[[k]]))
for (m in 1:length(indexlist[[k]])) {
nk[m]=length(indexlist[[k]][[m]])
}
TF=as.numeric(nk==c(1,1))
if(sum(TF)!=2){
order=append(order,indexlist[[k]][[which.min(nk)]])
}else if(sum(TF)==2){
order=append(order,indexlist[[k]][[1]])
order=append(order,indexlist[[k]][[2]])
}
if(length(order)==n+1){
order=order[-1]
break
}
}
order
}2.层次聚类代码编写
############编写层次聚类函数#####
my_clust<-function(X){ #使用最短距离法
X=as.matrix(X) #X转化为矩阵,便于后续计算
n=dim(X)[1] #n为样本数
p=dim(X)[2] #p为向量维数
C_all=matrix(1:n,byrow=TRUE,nrow = n,ncol = n)
#每一行记录每一次聚类后各向量对应的类别
distance_record=rep(0,n-1) #记录聚类时的最短距离
merge=matrix(NA,ncol = 2,nrow = n-1)
#merge记录聚类情况:每一行表示一次聚类(概念设定与hclust输出模型中的“merge”相同):
#两负数表示两个单向量聚为一类,
#一正一负表示负的那个单向量聚到正的数值所在的类
#两正则表示两个大类聚合为一类
indexlist<-list() #记录每一次聚的两个类的标签
#初始距离矩阵
D_dist=matrix(0,nrow = n,ncol = n)
D_dist[1:n,1:n] = as.matrix(dist(X)) #转化为矩阵格式(此时为对角矩阵且对角线元素为0)
D_dist[D_dist==0]=round(max(D_dist))+1 #将0全部转化为最大值
#返回距离最小的两个类
index=unique(which(D_dist[1:n,1:n]==min(D_dist[1:n,1:n])
arr.ind=TRUE)[1,])
#第一次聚类:两个单向量聚为一类:故merge第一行为两负数
merge[1,]=-index
#记录距离最小值
distance_record[1]=min(D_dist[1:n,1:n])
#给新类加上标签
for (m in 1:length(index)) {
C_all[2:n,which(C_all[1,]==index[m])]=n+1
}
#开始迭代层次聚类
i=2
while (i<=n) {
#各自把X属于同一类的提取出来
dataselected=split(X, C_all[i,],drop = FALSE)
classnum=length(dataselected) #记录此时类别数
D_dist=matrix(0,nrow = classnum,ncol = classnum)
#计算各类之间的距离
for (j in 1:classnum) {
for (k in j:classnum) {
if(j==k){
D_dist[j,k]=0
}else{
D_dist[j,k]=my_dist(dataselected[[j]],dataselected[[k]],p)
}
}
}
D_dist[D_dist==0]=round(max(D_dist),0)+1
#记录此次迭代中各类间距离中的最小值
distance_record[i]=min(D_dist[1:classnum,1:classnum])
#记录距离最小的两类在列表中的下标
index_pre=unique(which(D_dist[1:classnum,1:classnum]==min(D_dist[1:classnum,1:classnum]),arr.ind=TRUE)[1,])
#返回距离最小的两类在原始数据矩阵中的下标
index_list=list()
for (j in 1:length(index_pre)) {
data=dataselected[[index_pre[j]]]
index_list[[j]]=index_find(X,data,p)
}
#index_list为长度为2的列表,元素分别为两个类对应的数据在原始矩阵中的位置
index_list
#indexlist记录此次聚类情况
indexlist[[i-1]]=index_list
#此次聚类中merge值确定
n1=length(index_list[[1]])
n2=length(index_list[[2]])
if(n1==1&n2==1){
for (j in 1:2) {
merge[i,j]=-index_list[[j]]
}
}else if(n1!=1&n2==1){
merge[i,1]=-index_list[[2]]
merge[i,2]=C_all[i,][(index_list[[1]])[1]]-n
}else if(n1==1&n2!=1){
merge[i,1]=-index_list[[1]]
merge[i,2]=C_all[i,][(index_list[[2]])[1]]-n
}else if(n1!=1&n2!=1){
merge[i,1]=C_all[i,][(index_list[[1]])[1]]-n
merge[i,2]=C_all[i,][(index_list[[2]])[1]]-n
}
index=unlist(index_list)
#在此次迭代对应的矩阵的行位置记录原始各数据对应的类别标签,标签相同的为同一类
for (m in 1:length(index)) {
C_all[(i+1):n,which(C_all[1,]==index[m])]=n+i
}
#收敛条件:全部聚为一类时收敛
if(length(unique(C_all[i+1,]))==1){
break
}else{
i=i+1
}
}
list(C_all=C_all,merge=merge,height=distance_record,indexlist=indexlist)
}
3.检验代码
#数据:取iris中部分数据进行聚类
X=iris[1:20,-5]
X
#自编代码的结果
result=my_clust(X)
result$C_all #每一次聚类的情况(数字相同的为同一类)
result$merge #聚类过程记录
result$height #每次聚类时的最短距离的值
result$indexlist #聚类过程记录
n=nrow(X)
order_find(result$indexlist,n) #聚类图中的排序
#系统代码的结果
hc=hclust(dist(X),method = "single")
plot(hc)
hc$merge
hc$height
hc$order
hc$labels
plot(hc) 二、k均值算法代码
####函数编写
my_kmeans<-function(X,k,ddmax){
X=as.matrix(X)
n=dim(X)[1]
p=dim(X)[2]
C_all=matrix(NA,nrow = ddmax+1,ncol = n)
C_all[1,]=rep(0,n)
M=X[sample(1:n,k,replace=F),]
i=2
while (i<=(ddmax+1)) {
#计算聚类C
D=as.matrix(dist(rbind(X,M)))[1:n,(n+1):(n+k)]
colnames(D)=NULL
C_all[i,]=apply(D,1,which.min)
#计算新的类中心M
for (j in 1:k) {
M[j,]=apply(X[which(C_all[i,]==j),],2,mean)
}
#可视化
minx = min(X[,1])
maxx = max(X[,1])
miny = min(X[,2])
maxy = max(X[,2])
plot(X[which(C_all[i,]==1),1],X[which(C_all[i,]==1),2],
pch =1,xlim = c(minx, maxx),ylim = c(miny, maxy),col=2)
points(M[1,1],M[1,2],pch = 2,cex=1.5,col=2,lwd=2.5)
for (j in 2:k) {
points(X[which(C_all[i,]==j),1],X[which(C_all[i,]==j),2], pch = j,col=2*j)
points(M[j,1],M[j,2],pch = 2*j,col=2*j,cex=1.5,lwd=2.5)
}
#收敛判断
sum=sum(as.numeric(C_all[i-1,]==C_all[i,]))
if(sum==n){
C=C_all[i,]
break
}else{
i=i+1
}
}
list(C=C,M=M)
}检验代码
####检验(iris数据集二维数据)####
X<-iris[,-c(3,4,5)]
#设k=3,规定聚为3类
result=my_kmeans(X,k=3,ddmax=50)
result
C_all=result$C
iris<-read.csv("F:/大三下/数据挖掘/UCI数据集/Iris/iris.data",header = F)
iris[,5][iris[,5]=="Iris-setosa"]=2
iris[,5][iris[,5]=="Iris-versicolor"]=3
iris[,5][iris[,5]=="Iris-virginica"]=1
#聚类准确率
corr_prop=sum(as.numeric(C_all==iris[,5]))/nrow(iris)
corr_prop二、检验结果与分析
(一)层次聚类
1.自编函数
(1)聚类过程
> result$merge #聚类过程记录
[,1] [,2]
[1,] -18 -1
[2,] -5 1
[3,] -2 -13
[4,] -8 2
[5,] -10 3
[6,] -12 4
[7,] -3 -4
[8,] -20 6
[9,] -7 7
[10,] 5 9
[11,] -9 10
[12,] 8 11
[13,] -6 -19
[14,] -11 12
[15,] -17 14
[16,] 13 15
[17,] -14 16
[18,] -15 17
[19,] -16 18
分析:此即为聚类过程描述:第一行:即18、1聚为一类; 第二行:5与第一个大类聚为一类; 第三行:即2、13聚为一个新的类......第十九行:16与聚成的第18类聚为一类。
(2)每次聚类时的类间距离(此算法中为最短距离)
> result$height #每次聚类时的最短距离的值
[1] 0.1000000 0.1414214 0.1414214 0.1732051 0.1732051 0.2236068 0.2449490 0.2645751
[9] 0.2645751 0.2645751 0.3000000 0.3000000 0.3316625 0.3316625 0.3464102 0.3464102
[17] 0.3464102 0.4690416 0.5477226
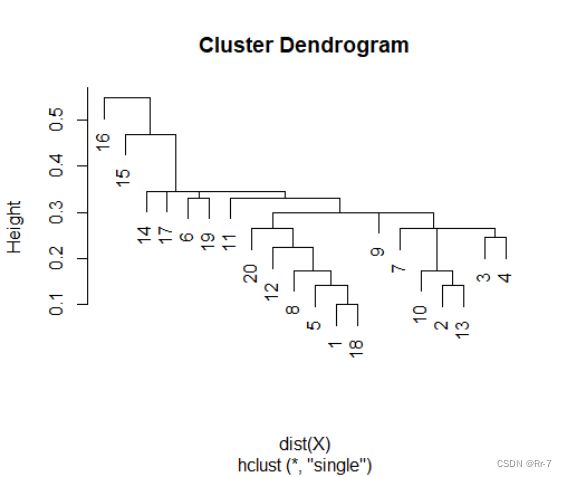
(3)聚类图中的排序
> order_find(result$indexlist,n) #聚类图中的排序
[1] 16 15 14 6 19 17 11 6 19 1 5 8 12 18 20 9 2 10 13 7
分析:从后往前,提取每一次聚类的两个类中含有数据较少的那个类中的数据
2.系统自带hclust函数
(1)聚类过程
> hc$merge
[,1] [,2]
[1,] -1 -18
[2,] -5 1
[3,] -2 -13
[4,] -8 2
[5,] -10 3
[6,] -12 4
[7,] -3 -4
[8,] -20 6
[9,] 5 7
[10,] -7 9
[11,] -9 10
[12,] 8 11
[13,] -6 -19
[14,] -11 12
[15,] 13 14
[16,] -17 15
[17,] -14 16
[18,] -15 17
[19,] -16 18
(2)每次聚类时的类间距离(此算法中为最短距离)
> hc$height
[1] 0.1000000 0.1414214 0.1414214 0.1732051 0.1732051 0.2236068 0.2449490 0.2645751
[9] 0.2645751 0.2645751 0.3000000 0.3000000 0.3316625 0.3316625 0.3464102 0.3464102
[17] 0.3464102 0.4690416 0.5477226
(3)聚类图中的排序
> hc$order
[1] 16 15 14 17 6 19 11 20 12 8 5 1 18 9 7 10 2 13 3 4
(4)聚类图
分析:综上可得自编函数的聚类结果与系统hclust的聚类结果一致,认为代码有效。
(二)k均值聚类
#此次聚类规定k=3
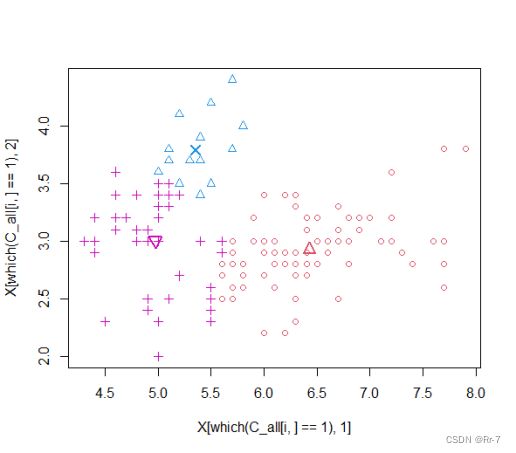
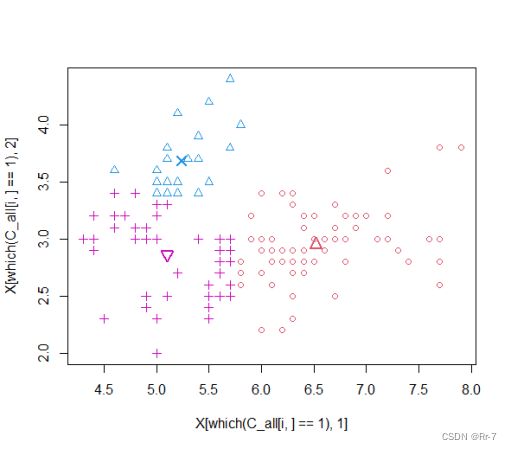
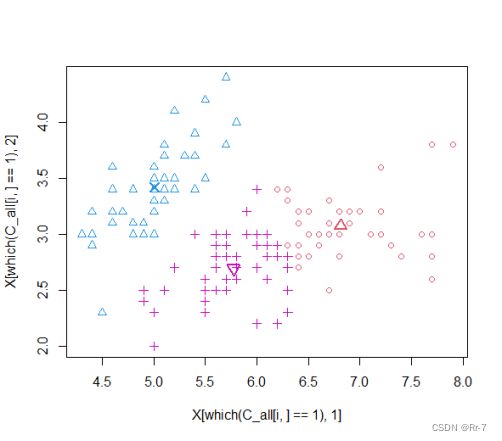
1.聚类结果:
> result
$C
[1] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
[37] 2 2 2 2 2 2 2 2 2 2 2 2 2 2 1 1 1 3 1 3 1 3 1 3 3 3 3 3 3 1 3 3 3 3 3 3
[73] 3 3 1 1 1 1 3 3 3 3 3 3 3 3 1 3 3 3 3 3 3 3 3 3 3 3 3 3 1 3 1 1 1 1 3 1
[109] 1 1 1 1 1 3 3 1 1 1 1 3 1 3 1 3 1 1 3 3 1 1 1 1 1 3 3 1 1 1 3 1 1 1 3 1
[145] 1 1 3 1 1 3
$M
V1 V2
[1,] 6.812766 3.074468
[2,] 5.006000 3.418000
[3,] 5.773585 2.692453
2.聚类正确率
> corr_prop
[1] 0.82
3.聚类过程部分散点图:
综上,聚类结果与原数据结果相比正确率较高,认为代码均有效。
三、小结
本次实验主要编写了层次聚类算法和k均值聚类算法,对于层次聚类过程:聚类结果非常清晰,而且唯一确定。但聚类结果非常依赖距离函数的选择,且聚类的时间空间复杂度均很大,在R运算时,若数据较大,运算时间将会变得很长。对于k均值聚类:使用逐步迭代的算法,计算出正确的分类使得损失函数达到最小,其聚类的时间空间复杂度相比层次聚类都大大减小。但其缺点是无法确定最佳的k,即无法使用先前的交叉验证算法,无法使用AIC、BIC准则等对k值进行选择。总的来说两种方法各有利弊,但从实际运用角度来看,k均值的使用更为广泛。