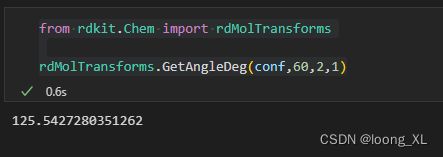
- python画出分子化学空间分布(UMAP)
Sakaiay
python
利用umap画出分子化学空间分布图安装pipinstallumap-learn下面是用一个数据集举的例子importtorchimportumapimportpandasaspdimportnumpyasnpimportmatplotlib.pyplotaspltimportseabornassnsfromsklearn.manifoldimportTSNEfromrdkit.Chemimport
- 静态库制作和使用
打酱油的;
linux文件系统编程静态库linuxC语言制作编译
静态库可以认为是一些目标代码的集合,是在可执行程序运行前就已经加入到执行码中,成为执行程序的一部分。按照习惯,一般以“.a”做为文件后缀名。静态库的命名一般分为三个部分:前缀:lib库名称:自己定义即可后缀:.a所以最终的静态库的名字应该为:libxxx.a1)静态库制作步骤1:将c源文件生成对应的.o文件deng@itcast:~/test/3static_lib$gcc-cadd.c-oadd
- Caddy 自动HTTPS 反向代理、重定向、静态页面 - docker版
記億揺晃着的那天
httpsdocker网络协议
简介Caddy是一个通用的、易于使用的Web服务器,具有以下特点:快速:Caddy使用Go语言编写,以高性能著称。安全:Caddy支持HTTPS、自动证书生成、HTTP/2等安全功能。易用:Caddy的配置文件简单易懂,易于配置。功能丰富:Caddy支持多种功能,包括反向代理、负载均衡、缓存、日志记录等。Caddy可以用于各种场景,包括:托管静态网站托管Web应用作为反向代理作为负载均衡器Cadd
- mingw64下动态库和静态库链接的真正区别和用法(详细)
IncludeFun
windowsmicrosoftc++linuxgnu
笔者一直以来都对mingw64下动态库和静态库链接的真正区别和用法存疑,于是做了一些测试,这篇文章记录了测试过程和测试结果,如果只想知道结果可以跳转到文章末尾一、准备工作首先准备三个测试文件其中add.c最终会被编译会静态库或动态库,被测试文件main.c调用使用如下命令构建静态库gcc-cadd.c-oadd.oarrcslibadd.aadd.o得到静态库如下:接着使用如下命令构建动态库gcc
- 【C语言】动态库
榕树子
linuxcc语言
0.文件清单1.生成add.o2.生成共享库libadd.so3.编译主程序app4.解决方法0.文件清单add.c#includeintadd(){printf("addinit\n");return0;}add.hintadd();main.c#includeintmian(){add();return0;}1.生成add.ogcc-cadd.c-fPIC2.生成共享库libadd.sogcc
- spfa处理差分约束
钊气蓬勃.
c++算法蓝桥杯
差分约束是一群不等关系然后求可行解或者最小值最大值的情况1.求最大值,用最短路,也就是符号要(a)>=(b)+cadd(b,a,c)2.求最小值,用最长路,也就是符号要(a)=b且b>=ax==2说明b>a则b>=a+1x==3说明a>=bx==4说明a>b则a>=b+1x==5说明b>=a因为保证每个小孩都有一个糖果,则每个小孩>=0+1#includeusingnamespacestd;con
- 动静态库的创建 | 使用 | 加载
深度搜索
linux运维服务器动静态库
动静态库的介绍静态库(.a):在程序编译的时候,把库的代码链接(拷贝)到可执行程序。动态库(.so):在程序的执行时,链接动态库的代码。多个程序同时共享代码。创建一个静态库:创建计算器,带有加减乘除功能。声明和定义分离。创建文件所有文件:头文件.h源文件.c静态库的打包一:生成.o文件利用指令gcc-cgcc-cAdd.cSub.cDiv.cMul.c为了简化操作,我们只测试加法功能创建testa
- python版本3.7情况下安装rdkit,torch_geometric,numpy的过程
CWQF-T
pythonnumpy深度学习
前言在上一篇中,我是在anaconda自带的python3.9的环境下安装的torch_geometric,相对来说步骤比较少,一个下午就安装好了,但是在最近接触的一篇论文中需要用到rdkit这个包,我尝试在这个虚拟环境下安装,但是出现了如下提示,表示此包和python3.9不适配-rdkit->python[version='2.7.*|3.5.*|3.6.*|>=2.7,=3.5,=3.6,=
- rdkit安装
qq_46572534
深度学习pythonanaconda
不夸张,我用过最香的rdkit安装引用:https://anaconda.org/rdkit/rdkit
- rdkit 小分子库安装使用;PyRosetta大分子库安装
loong_XL
知识点CADD/AIDDpythonpycharm开发语言
1、rdkit安装使用使用教程参考:https://sites.google.com/view/ml-basics/dealing-with-moleculesgithub地址:https://github.com/rdkit/rdkit1)pip安装python-mpipinstallrdkitpython-c"fromrdkitimportChem;print(Chem.MolToMolBlo
- pthon3.8版本之后rdkit安装之后缺少dll文件的问题
Ai_Chem
python
pthon3.8版本之后rdkit安装之后缺少dll文件的问题描述原因解决方法描述出现:fromrdkit.Chem.rdmolfilesimport*ImportError:DLLloadfailedwhileimportingrdmolfiles:找不到指定的程序。原因使用下面两种方法都是安装的之前版本的rdkit,python3.8之后现在应该是叫rdkit-pypi//两种出错的安装方式c
- RDkit的安装
Alkaid路
conda
首先在官网找到安装教程https://www.rdkit.org/docs/Install.html:2.提示需要更新conda,输入y,回车;3.更新完毕后激活刚刚创建的虚拟环境;4.检验rdkit是否安装成功:输入python进入python环境;importrdkitrdkit.__version__安装成功
- 【RDKit】关于RDKit安装问题,python兼容性问题(最后有RDKit的正确安装方法)
DJ.马
RDKitpython开发语言
//两种出错的安装方式condainstall-cconda-forgerdkitcondainstall-crdkitrdkit使用上面两种方法安装会出现如下报错:fromrdkit.Chem.rdmolfilesimport*ImportError:DLLloadfailedwhileimportingrdmolfiles:找不到指定的程序。切记不要直接conda安装使用下面两种方法都是安装的
- makefile的机制,执行命令的流程分析
职业UI仔
makemakefliecmake笔记linux
makefile的机制,流程分析calc:add.osub.omulti.ogccadd.osub.omulti.ocalc.cpp-ocalcadd.o:add.cppgcc-cadd.cpp-oadd.osub.o:sub.cppgcc-csub.cpp-osub.omulti.o:multi.cppgcc-cmulti.cpp-omulti.oclean:rm-rf*.ocalc按照这个例子
- makefile里面的变量使用,系统变量
职业UI仔
makemakefliecmake笔记linux
文章目录makefile里面的变量使用makefile里面的变量使用calc:add.osub.omulti.ogccadd.osub.omulti.ocalc.cpp-ocalcadd.o:add.cppgcc-cadd.cpp-oadd.osub.o:sub.cppgcc-csub.cpp-osub.omulti.o:multi.cppgcc-cmulti.cpp-omulti.oclean:
- Pycharm使用Conda创建的新环境,并使用terminal下载rdkit
贩卖梦境
RNN用于分子生成pythonanacondapycharm
1.配置anaconda环境变量已经下载安装好anaconda之后,没有添加系统变量,找不到位置,所有系统无法识别conda命令。添加对应的Anaconda环境变量:(以自己的anaconda安装路径为准)D:\programe\anaconda;D:\programe\anaconda\Library\mingw-w64\bin;D:\programe\anaconda\Library\usr\
- 技术动态 | 知识图谱遇上RAG行业问答:回顾知识检索增强范式、挑战及与知识图谱的结合...
开放知识图谱
知识图谱人工智能
转载公众号|老刘说NLP今天我们来看看AIDD2023(https://aidd.vip/dhrc-sz2023)中有关知识图谱与行业问答上的一些有趣的分享,供大家一起参考。一、知识检索增强范式、拷问、评估与选型同济大学王昊奋老师的《知识增强大模型:垂域落地的最后一公里》报告中,很有启发性地介绍了知识增强大模型的一些思考,特别棒,择其中几个有趣的点,分享给大家。1、RAGvsFine-tuning
- 一起学 RDKit Cookbook (1)
wufeil
药物设计rdkitpython机器学习深度学习
这部分内容来自于RDKIT的简单教程:https://www.rdkit.org/docs/Cookbook.htmlRDKIT算是化学生物的神器了,以前每一次都是即时查接口,现在按照这个简易教程走一遍,增加感觉。最好的办法就是全程过一遍。当然啦,在这些过程中,也有我自己对RDKit的一些理解吧。如果有不对的地方,请多多指正!一、画分子Chem.MolFromSmiles()输入的字符串是SMIL
- 机器学习模型的超参数优化用于分子性质预测
wufeil
药物设计机器学习深度学习python
在《预测化学分子的nlogP——基于sklearn,deepchem,DGL,Rdkit的图卷积网络模型》中简单介绍了sklearn模型的使用方法。现在来介绍一下,如何对sklearn模型进行超参数优化。要想获得更好的模型,离不开超参数优化。这里的目的是:示例使用交叉验证结合网格搜索,对机器学习模型进行超参数优化。一、导入相关模块主要是pandas和sklearn的模型及模型评价指标。import
- 如何在linux下制作静态库和动态库
羊羊羊i
linuxlinux运维服务器
目录静态库(.a)制作静态库如何使用静态库动态库(.so)制作动态库如何使用动态库(.so)静态库(.a)静态库顾名思义是静态的,即程序编译链接时,会把静态库的代码链接到可执行文件中。运行时不需要静态库(将静态库删了也能运行)。相当于静态库的代码复制到了可执行文件中。制作静态库分别有如下add.cadd.hsub.csub.h四个文件格式。首先把.c文件都编译成.o文件。使用下面命令//-c编译到
- DeepPurpose 生物化学深度学习库;蛋白靶点小分子药物对接亲和力预测虚拟筛选
loong_XL
CADD/AIDD深度学习人工智能CADD药物对接药物筛选
参考:https://blog.csdn.net/c9Yv2cf9I06K2A9E/article/details/107649770https://github.com/kexinhuang12345/DeepPurpose##安装pipinstallDeepPurposerdkitDeepPurpose包括:数据:关联TDC库下载,是同一作者开发的https://blog.csdn.net/w
- rdkit&networkx | smiles与graph与邻接矩阵相互转换
ASKCOS
AIDDCADD化学生物
fromrdkitimportrdBase,Chemfromrdkit.ChemimportAllChem,Drawfromrdkit.Chem.DrawimportrdMolDraw2DfromIPython.displayimportSVGfrommatplotlib.colorsimportColorConverterimportnetworkxasnximportargparse
- rdkit&python | 标准化检查分子正确性
ASKCOS
AIDDCADD化学生物pythonrdkit
分子结构处理MolecularSanitization默认情况下,RDKit读入分子的时候会进行分子结构检查处理(MolecularSanitization)。结构处理的过程中会自动计算一些有用信息,如杂化、环信息等,保证分子的结构在化学上是合理的:价键合理。Sanitization的检查过程,包含11步。clearComputedProps:清除已有的分子、原子、键的计算性质,如Thisstep
- rdkit&python | 修正化合物中原子价态错误
ASKCOS
AIDDCADD化学生物
错误smiles价态纠正mol=Chem.MolFromSmiles('C[CH2+]1(C#N)CC1')RDKitERROR:[08:03:09]Explicitvalenceforatom#1C,5,isgreaterthanpermittedBOND_TYPE=[0,Chem.rdchem.BondType.SINGLE,Chem.rdc
- rdkit&nlp | smiles数据扩增与smiles标准化
ASKCOS
AIDDCADD化学生物python自然语言处理pytorch
fromrdkitimportChemfromrdkit.Chem.DrawimportIPythonConsolesmi='CC(=O)OC1=CC=CC=C1C(=O)O'mol=Chem.MolFromSmiles(smi)用doRandom产生随机smilesrandom_equivalent_smiles=Ch
- rdkit&python | 消除smiles中的立体结构信息
ASKCOS
AIDDCADD化学生物pythonrdkit
fromrdkitimportChemsmi='CCC/C=C/CC'm=Chem.MolFromSmiles(smi)Chem.MolToSmiles(m,isomericSmiles=False)‘CCC=CCCC’smi='CCCC[C@@H](I)C'm=Chem
- rdkit&python | 基于类药性规则对分子进行筛选
ASKCOS
AIDDCADD化学生物化学信息学
类药性类药性指化合物与已知药物的相似性。具有类药性的化合物并不是药物,但是具有成为药物的可能,这一类化合物称为类药性分子或药物类似物分子。类药性是一个模糊的概念,在药物研发中,类药性研究基于先导化合物之上,可以说类药性分子是高质量的先导化合物。Lipinski五规则类药五原则,是辉瑞公司资深药物化学家ChristopherA.Lipinski在1997年提出的筛选类药分子的基本法则,符合Lipin
- rdkit&python | 超全化合物分子清理方法
ASKCOS
AIDDCADD化学生物rdkit生物信息学化学信息学
分子结构处理MolecularSanitization默认情况下,RDKit读入分子的时候会进行分子结构检查处理(MolecularSanitization)。结构处理的过程中会自动计算一些有用信息,如杂化、环信息等,保证分子的结构在化学上是合理的:价键合理。Sanitization的检查过程,包含11步。clearComputedProps:清除已有的分子、原子、键的计算性质,如Thisstep
- chemoinformatics&python | 化学信息学软件包安装方法集合
ASKCOS
AIDDCADD化学生物RDKit化学信息学
rdkit化学信息学condainstall-cconda-forgerdkitcondainstall-crdkitrdkitpipinstallrdkit-pypi谷歌colab上安装rdkit!curl-Lordkit_installer.pyhttps://git.io/fxiPZimportrdkit_installer%timerdkit_installer.install()indi
- rdkit&python | 分子3D构象与结构优化与描述符计算
ASKCOS
AIDDCADD化学生物rdkitAIpython化学
导入库fromrdkit.ChemimportAllChemfromrdkit.Chem.DescriptorsimportrdMolDescriptorsfromrdkitimportChemfromrdkit.Chem.DrawimportIPythonConsolesmi="COC(=O)Cc1cc(F)cc2c1[C@@H](C)CO2"mol=Chem.MolFromSmile
- ASM系列四 利用Method 组件动态注入方法逻辑
lijingyao8206
字节码技术jvmAOP动态代理ASM
这篇继续结合例子来深入了解下Method组件动态变更方法字节码的实现。通过前面一篇,知道ClassVisitor 的visitMethod()方法可以返回一个MethodVisitor的实例。那么我们也基本可以知道,同ClassVisitor改变类成员一样,MethodVIsistor如果需要改变方法成员,注入逻辑,也可以
- java编程思想 --内部类
百合不是茶
java内部类匿名内部类
内部类;了解外部类 并能与之通信 内部类写出来的代码更加整洁与优雅
1,内部类的创建 内部类是创建在类中的
package com.wj.InsideClass;
/*
* 内部类的创建
*/
public class CreateInsideClass {
public CreateInsideClass(
- web.xml报错
crabdave
web.xml
web.xml报错
The content of element type "web-app" must match "(icon?,display-
name?,description?,distributable?,context-param*,filter*,filter-mapping*,listener*,servlet*,s
- 泛型类的自定义
麦田的设计者
javaandroid泛型
为什么要定义泛型类,当类中要操作的引用数据类型不确定的时候。
采用泛型类,完成扩展。
例如有一个学生类
Student{
Student(){
System.out.println("I'm a student.....");
}
}
有一个老师类
- CSS清除浮动的4中方法
IT独行者
JavaScriptUIcss
清除浮动这个问题,做前端的应该再熟悉不过了,咱是个新人,所以还是记个笔记,做个积累,努力学习向大神靠近。CSS清除浮动的方法网上一搜,大概有N多种,用过几种,说下个人感受。
1、结尾处加空div标签 clear:both 1 2 3 4
.div
1
{
background
:
#000080
;
border
:
1px
s
- Cygwin使用windows的jdk 配置方法
_wy_
jdkwindowscygwin
1.[vim /etc/profile]
JAVA_HOME="/cgydrive/d/Java/jdk1.6.0_43" (windows下jdk路径为D:\Java\jdk1.6.0_43)
PATH="$JAVA_HOME/bin:${PATH}"
CLAS
- linux下安装maven
无量
mavenlinux安装
Linux下安装maven(转) 1.首先到Maven官网
下载安装文件,目前最新版本为3.0.3,下载文件为
apache-maven-3.0.3-bin.tar.gz,下载可以使用wget命令;
2.进入下载文件夹,找到下载的文件,运行如下命令解压
tar -xvf apache-maven-2.2.1-bin.tar.gz
解压后的文件夹
- tomcat的https 配置,syslog-ng配置
aichenglong
tomcathttp跳转到httpssyslong-ng配置syslog配置
1) tomcat配置https,以及http自动跳转到https的配置
1)TOMCAT_HOME目录下生成密钥(keytool是jdk中的命令)
keytool -genkey -alias tomcat -keyalg RSA -keypass changeit -storepass changeit
- 关于领号活动总结
alafqq
活动
关于某彩票活动的总结
具体需求,每个用户进活动页面,领取一个号码,1000中的一个;
活动要求
1,随机性,一定要有随机性;
2,最少中奖概率,如果注数为3200注,则最多中4注
3,效率问题,(不能每个人来都产生一个随机数,这样效率不高);
4,支持断电(仍然从下一个开始),重启服务;(存数据库有点大材小用,因此不能存放在数据库)
解决方案
1,事先产生随机数1000个,并打
- java数据结构 冒泡排序的遍历与排序
百合不是茶
java
java的冒泡排序是一种简单的排序规则
冒泡排序的原理:
比较两个相邻的数,首先将最大的排在第一个,第二次比较第二个 ,此后一样;
针对所有的元素重复以上的步骤,除了最后一个
例题;将int array[]
- JS检查输入框输入的是否是数字的一种校验方法
bijian1013
js
如下是JS检查输入框输入的是否是数字的一种校验方法:
<form method=post target="_blank">
数字:<input type="text" name=num onkeypress="checkNum(this.form)"><br>
</form>
- Test注解的两个属性:expected和timeout
bijian1013
javaJUnitexpectedtimeout
JUnit4:Test文档中的解释:
The Test annotation supports two optional parameters.
The first, expected, declares that a test method should throw an exception.
If it doesn't throw an exception or if it
- [Gson二]继承关系的POJO的反序列化
bit1129
POJO
父类
package inheritance.test2;
import java.util.Map;
public class Model {
private String field1;
private String field2;
private Map<String, String> infoMap
- 【Spark八十四】Spark零碎知识点记录
bit1129
spark
1. ShuffleMapTask的shuffle数据在什么地方记录到MapOutputTracker中的
ShuffleMapTask的runTask方法负责写数据到shuffle map文件中。当任务执行完成成功,DAGScheduler会收到通知,在DAGScheduler的handleTaskCompletion方法中完成记录到MapOutputTracker中
- WAS各种脚本作用大全
ronin47
WAS 脚本
http://www.ibm.com/developerworks/cn/websphere/library/samples/SampleScripts.html
无意中,在WAS官网上发现的各种脚本作用,感觉很有作用,先与各位分享一下
获取下载
这些示例 jacl 和 Jython 脚本可用于在 WebSphere Application Server 的不同版本中自
- java-12.求 1+2+3+..n不能使用乘除法、 for 、 while 、 if 、 else 、 switch 、 case 等关键字以及条件判断语句
bylijinnan
switch
借鉴网上的思路,用java实现:
public class NoIfWhile {
/**
* @param args
*
* find x=1+2+3+....n
*/
public static void main(String[] args) {
int n=10;
int re=find(n);
System.o
- Netty源码学习-ObjectEncoder和ObjectDecoder
bylijinnan
javanetty
Netty中传递对象的思路很直观:
Netty中数据的传递是基于ChannelBuffer(也就是byte[]);
那把对象序列化为字节流,就可以在Netty中传递对象了
相应的从ChannelBuffer恢复对象,就是反序列化的过程
Netty已经封装好ObjectEncoder和ObjectDecoder
先看ObjectEncoder
ObjectEncoder是往外发送
- spring 定时任务中cronExpression表达式含义
chicony
cronExpression
一个cron表达式有6个必选的元素和一个可选的元素,各个元素之间是以空格分隔的,从左至右,这些元素的含义如下表所示:
代表含义 是否必须 允许的取值范围 &nb
- Nutz配置Jndi
ctrain
JNDI
1、使用JNDI获取指定资源:
var ioc = {
dao : {
type :"org.nutz.dao.impl.NutDao",
args : [ {jndi :"jdbc/dataSource"} ]
}
}
以上方法,仅需要在容器中配置好数据源,注入到NutDao即可.
- 解决 /bin/sh^M: bad interpreter: No such file or directory
daizj
shell
在Linux中执行.sh脚本,异常/bin/sh^M: bad interpreter: No such file or directory。
分析:这是不同系统编码格式引起的:在windows系统中编辑的.sh文件可能有不可见字符,所以在Linux系统下执行会报以上异常信息。
解决:
1)在windows下转换:
利用一些编辑器如UltraEdit或EditPlus等工具
- [转]for 循环为何可恨?
dcj3sjt126com
程序员读书
Java的闭包(Closure)特征最近成为了一个热门话题。 一些精英正在起草一份议案,要在Java将来的版本中加入闭包特征。 然而,提议中的闭包语法以及语言上的这种扩充受到了众多Java程序员的猛烈抨击。
不久前,出版过数十本编程书籍的大作家Elliotte Rusty Harold发表了对Java中闭包的价值的质疑。 尤其是他问道“for 循环为何可恨?”[http://ju
- Android实用小技巧
dcj3sjt126com
android
1、去掉所有Activity界面的标题栏
修改AndroidManifest.xml 在application 标签中添加android:theme="@android:style/Theme.NoTitleBar"
2、去掉所有Activity界面的TitleBar 和StatusBar
修改AndroidManifes
- Oracle 复习笔记之序列
eksliang
Oracle 序列sequenceOracle sequence
转载请出自出处:http://eksliang.iteye.com/blog/2098859
1.序列的作用
序列是用于生成唯一、连续序号的对象
一般用序列来充当数据库表的主键值
2.创建序列语法如下:
create sequence s_emp
start with 1 --开始值
increment by 1 --増长值
maxval
- 有“品”的程序员
gongmeitao
工作
完美程序员的10种品质
完美程序员的每种品质都有一个范围,这个范围取决于具体的问题和背景。没有能解决所有问题的
完美程序员(至少在我们这个星球上),并且对于特定问题,完美程序员应该具有以下品质:
1. 才智非凡- 能够理解问题、能够用清晰可读的代码翻译并表达想法、善于分析并且逻辑思维能力强
(范围:用简单方式解决复杂问题)
- 使用KeleyiSQLHelper类进行分页查询
hvt
sql.netC#asp.nethovertree
本文适用于sql server单主键表或者视图进行分页查询,支持多字段排序。KeleyiSQLHelper类的最新代码请到http://hovertree.codeplex.com/SourceControl/latest下载整个解决方案源代码查看。或者直接在线查看类的代码:http://hovertree.codeplex.com/SourceControl/latest#HoverTree.D
- SVG 教程 (三)圆形,椭圆,直线
天梯梦
svg
SVG <circle> SVG 圆形 - <circle>
<circle> 标签可用来创建一个圆:
下面是SVG代码:
<svg xmlns="http://www.w3.org/2000/svg" version="1.1">
<circle cx="100" c
- 链表栈
luyulong
java数据结构
public class Node {
private Object object;
private Node next;
public Node() {
this.next = null;
this.object = null;
}
public Object getObject() {
return object;
}
public
- 基础数据结构和算法十:2-3 search tree
sunwinner
Algorithm2-3 search tree
Binary search tree works well for a wide variety of applications, but they have poor worst-case performance. Now we introduce a type of binary search tree where costs are guaranteed to be loga
- spring配置定时任务
stunizhengjia
springtimer
最近因工作的需要,用到了spring的定时任务的功能,觉得spring还是很智能化的,只需要配置一下配置文件就可以了,在此记录一下,以便以后用到:
//------------------------定时任务调用的方法------------------------------
/**
* 存储过程定时器
*/
publi
- ITeye 8月技术图书有奖试读获奖名单公布
ITeye管理员
活动
ITeye携手博文视点举办的8月技术图书有奖试读活动已圆满结束,非常感谢广大用户对本次活动的关注与参与。
8月试读活动回顾:
http://webmaster.iteye.com/blog/2102830
本次技术图书试读活动的优秀奖获奖名单及相应作品如下(优秀文章有很多,但名额有限,没获奖并不代表不优秀):
《跨终端Web》
gleams:http